+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 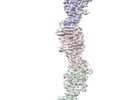 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of OmcZ nanowire from Geobacter sulfurreducens | |||||||||
 マップデータ マップデータ | map of octaheme cytochrome OmcZ nanowire from G. sulfurreducens | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | nanowires / PROTEIN FIBRIL | |||||||||
| 機能・相同性 | IPT/TIG domain / Multiheme cytochrome superfamily / IPT domain / Immunoglobulin E-set / Immunoglobulin-like fold /  シトクロムc シトクロムc 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Geobacter sulfurreducens (バクテリア) / Geobacter sulfurreducens (バクテリア) /   Geobacter sulfurreducens PCA (バクテリア) Geobacter sulfurreducens PCA (バクテリア) | |||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.4 Å クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Gu Y / Srikanth V / Malvankar NS / Samatey FA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2023 ジャーナル: Nat Microbiol / 年: 2023タイトル: Structure of Geobacter cytochrome OmcZ identifies mechanism of nanowire assembly and conductivity. 著者: Yangqi Gu / Matthew J Guberman-Pfeffer / Vishok Srikanth / Cong Shen / Fabian Giska / Kallol Gupta / Yuri Londer / Fadel A Samatey / Victor S Batista / Nikhil S Malvankar /   要旨: OmcZ nanowires produced by Geobacter species have high electron conductivity (>30 S cm). Of 111 cytochromes present in G. sulfurreducens, OmcZ is the only known nanowire-forming cytochrome ...OmcZ nanowires produced by Geobacter species have high electron conductivity (>30 S cm). Of 111 cytochromes present in G. sulfurreducens, OmcZ is the only known nanowire-forming cytochrome essential for the formation of high-current-density biofilms that require long-distance (>10 µm) extracellular electron transport. However, the mechanisms underlying OmcZ nanowire assembly and high conductivity are unknown. Here we report a 3.5-Å-resolution cryogenic electron microscopy structure for OmcZ nanowires. Our structure reveals linear and closely stacked haems that may account for conductivity. Surface-exposed haems and charge interactions explain how OmcZ nanowires bind to diverse extracellular electron acceptors and how organization of nanowire network re-arranges in different biochemical environments. In vitro studies explain how G. sulfurreducens employ a serine protease to control the assembly of OmcZ monomers into nanowires. We find that both OmcZ and serine protease are widespread in environmentally important bacteria and archaea, thus establishing a prevalence of nanowire biogenesis across diverse species and environments. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23481.map.gz emd_23481.map.gz | 12.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23481-v30.xml emd-23481-v30.xml emd-23481.xml emd-23481.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23481_fsc.xml emd_23481_fsc.xml | 13.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23481.png emd_23481.png | 94.7 KB | ||
| マスクデータ |  emd_23481_msk_1.map emd_23481_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| その他 |  emd_23481_half_map_1.map.gz emd_23481_half_map_1.map.gz emd_23481_half_map_2.map.gz emd_23481_half_map_2.map.gz | 171.4 MB 171.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23481 http://ftp.pdbj.org/pub/emdb/structures/EMD-23481 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23481 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23481 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7lq5MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23481.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23481.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | map of octaheme cytochrome OmcZ nanowire from G. sulfurreducens | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.828 Å | ||||||||||||||||||||
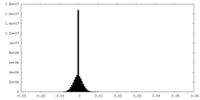
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_23481_msk_1.map emd_23481_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map of octaheme cytochrome OmcZ nanowire from G. sulfurreducens
| ファイル | emd_23481_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map of octaheme cytochrome OmcZ nanowire from G. sulfurreducens | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map of octaheme cytochrome OmcZ nanowire from G. sulfurreducens
| ファイル | emd_23481_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map of octaheme cytochrome OmcZ nanowire from G. sulfurreducens | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Polymerized cytochrome OmcZ from Geobacter sulfurreducens
| 全体 | 名称: Polymerized cytochrome OmcZ from Geobacter sulfurreducens |
|---|---|
| 要素 |
|
-超分子 #1: Polymerized cytochrome OmcZ from Geobacter sulfurreducens
| 超分子 | 名称: Polymerized cytochrome OmcZ from Geobacter sulfurreducens タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) |
-分子 #1: Cytochrome c
| 分子 | 名称: Cytochrome c / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Geobacter sulfurreducens PCA (バクテリア) / 株: ATCC 51573 / DSM 12127 / PCA Geobacter sulfurreducens PCA (バクテリア) / 株: ATCC 51573 / DSM 12127 / PCA |
| 分子量 | 理論値: 27.686797 KDa |
| 配列 | 文字列: AVPPPPVNQF LGIYDTKFPN LTKADCLECH VSDTVLVQQH HALINTVTPP ASCINTSGTV PPTLATGCHV MVPDGSGGFT FQDFRNCFN CHTQTPHHTS PAAVAKDCKY CHGNFIDNPL DGHYIPTYSA SSVTPMPSGR SVTATDGNVV IVQGCEACHQ A APNAIDPK ...文字列: AVPPPPVNQF LGIYDTKFPN LTKADCLECH VSDTVLVQQH HALINTVTPP ASCINTSGTV PPTLATGCHV MVPDGSGGFT FQDFRNCFN CHTQTPHHTS PAAVAKDCKY CHGNFIDNPL DGHYIPTYSA SSVTPMPSGR SVTATDGNVV IVQGCEACHQ A APNAIDPK TNTVRPIFSN QDTHHGTGIT DCNLCHNTSS NVPIRQCEVC HGVNSLHNIQ KDSPNAANLG TVKPGLEDLG WG HIGNNWD CQGCHWSWFG N UniProtKB:  シトクロムc シトクロムc |
-分子 #2: HEME C
| 分子 | 名称: HEME C / タイプ: ligand / ID: 2 / コピー数: 24 / 式: HEC |
|---|---|
| 分子量 | 理論値: 618.503 Da |
| Chemical component information |  ChemComp-HEC: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 10.5 / 構成要素 - 濃度: 15.0 mM / 構成要素 - 式: C2H7NO / 構成要素 - 名称: Ethanolamine エタノールアミン エタノールアミン |
|---|---|
| グリッド | モデル: C-flat-2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 30 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 2-10 / 撮影したグリッド数: 1 / 実像数: 14508 / 平均露光時間: 0.2 sec. / 平均電子線量: 1.2 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Initial fitting was done with Chimera and refinement was done in Phenix |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 当てはまり具合の基準: Correlation coefficient |
| 得られたモデル |  PDB-7lq5: |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X