+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human SetD3 methyl-transferase in complex with 2A protease from Coxsackievirus B3 | |||||||||
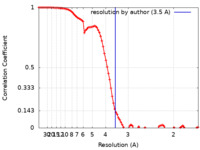
 マップデータ マップデータ | From cryosparc 2 non uniform refinement job, final map, filtered by FSC (3.5A), sharpened with b-factor of 180. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Methyltransferase / viral protease / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein-histidine N-methyltransferase / peptidyl-histidine methylation / regulation of uterine smooth muscle contraction / protein-L-histidine N-tele-methyltransferase activity / actin modification / symbiont-mediated suppression of host NF-kappaB cascade / symbiont-mediated perturbation of host transcription / histone H3K36 methyltransferase activity / histone H3K4 methyltransferase activity / positive regulation of muscle cell differentiation ...protein-histidine N-methyltransferase / peptidyl-histidine methylation / regulation of uterine smooth muscle contraction / protein-L-histidine N-tele-methyltransferase activity / actin modification / symbiont-mediated suppression of host NF-kappaB cascade / symbiont-mediated perturbation of host transcription / histone H3K36 methyltransferase activity / histone H3K4 methyltransferase activity / positive regulation of muscle cell differentiation / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / picornain 2A / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / PKMTs methylate histone lysines / nucleoside-triphosphate phosphatase / protein complex oligomerization / monoatomic ion channel activity / actin binding / symbiont-mediated suppression of host gene expression / DNA replication / RNA polymerase II-specific DNA-binding transcription factor binding / RNA helicase activity / transcription coactivator activity / induction by virus of host autophagy / RNA-directed RNA polymerase / viral RNA genome replication / cysteine-type endopeptidase activity / RNA-dependent RNA polymerase activity / virus-mediated perturbation of host defense response / DNA-templated transcription / host cell nucleus / virion attachment to host cell / chromatin / structural molecule activity / positive regulation of DNA-templated transcription / ATP hydrolysis activity / positive regulation of transcription by RNA polymerase II / proteolysis / RNA binding / nucleoplasm / ATP binding / membrane / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Coxsackievirus B3 (strain Nancy) (コクサッキーウイルス) / Coxsackievirus B3 (strain Nancy) (コクサッキーウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Verba KA / Schulze-Gahmen U | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structure-function analysis of enterovirus protease 2A in complex with its essential host factor SETD3. 著者: Christine E Peters / Ursula Schulze-Gahmen / Manon Eckhardt / Gwendolyn M Jang / Jiewei Xu / Ernst H Pulido / Conner Bardine / Charles S Craik / Melanie Ott / Or Gozani / Kliment A Verba / ...著者: Christine E Peters / Ursula Schulze-Gahmen / Manon Eckhardt / Gwendolyn M Jang / Jiewei Xu / Ernst H Pulido / Conner Bardine / Charles S Craik / Melanie Ott / Or Gozani / Kliment A Verba / Ruth Hüttenhain / Jan E Carette / Nevan J Krogan /  要旨: Enteroviruses cause a number of medically relevant and widespread human diseases with no approved antiviral therapies currently available. Host-directed therapies present an enticing option for this ...Enteroviruses cause a number of medically relevant and widespread human diseases with no approved antiviral therapies currently available. Host-directed therapies present an enticing option for this diverse genus of viruses. We have previously identified the actin histidine methyltransferase SETD3 as a critical host factor physically interacting with the viral protease 2A. Here, we report the 3.5 Å cryo-EM structure of SETD3 interacting with coxsackievirus B3 2A at two distinct interfaces, including the substrate-binding surface within the SET domain. Structure-function analysis revealed that mutations of key residues in the SET domain resulted in severely reduced binding to 2A and complete protection from enteroviral infection. Our findings provide insight into the molecular basis of the SETD3-2A interaction and a framework for the rational design of host-directed therapeutics against enteroviruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23441.map.gz emd_23441.map.gz | 59.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23441-v30.xml emd-23441-v30.xml emd-23441.xml emd-23441.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23441_fsc.xml emd_23441_fsc.xml | 9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23441.png emd_23441.png | 137 KB | ||
| マスクデータ |  emd_23441_msk_1.map emd_23441_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-23441.cif.gz emd-23441.cif.gz | 6.9 KB | ||
| その他 |  emd_23441_half_map_1.map.gz emd_23441_half_map_1.map.gz emd_23441_half_map_2.map.gz emd_23441_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23441 http://ftp.pdbj.org/pub/emdb/structures/EMD-23441 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23441 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23441 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23441_validation.pdf.gz emd_23441_validation.pdf.gz | 946.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23441_full_validation.pdf.gz emd_23441_full_validation.pdf.gz | 946 KB | 表示 | |
| XML形式データ |  emd_23441_validation.xml.gz emd_23441_validation.xml.gz | 16.5 KB | 表示 | |
| CIF形式データ |  emd_23441_validation.cif.gz emd_23441_validation.cif.gz | 21.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23441 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23441 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23441 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23441 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7lmsMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23441.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23441.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | From cryosparc 2 non uniform refinement job, final map, filtered by FSC (3.5A), sharpened with b-factor of 180. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.835 Å | ||||||||||||||||||||
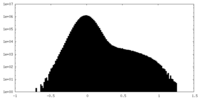
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_23441_msk_1.map emd_23441_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
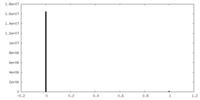
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_23441_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
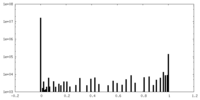
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_23441_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
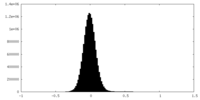
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of Coxsackievirus B3 2A protease and human SetD3 ...
| 全体 | 名称: Ternary complex of Coxsackievirus B3 2A protease and human SetD3 methyltransferase |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of Coxsackievirus B3 2A protease and human SetD3 ...
| 超分子 | 名称: Ternary complex of Coxsackievirus B3 2A protease and human SetD3 methyltransferase タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Coxsackievirus B3 (strain Nancy) (コクサッキーウイルス) Coxsackievirus B3 (strain Nancy) (コクサッキーウイルス) |
| 分子量 | 理論値: 84 KDa |
-分子 #1: Protease 2A
| 分子 | 名称: Protease 2A / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: picornain 2A |
|---|---|
| 由来(天然) | 生物種:  Coxsackievirus B3 (strain Nancy) (コクサッキーウイルス) Coxsackievirus B3 (strain Nancy) (コクサッキーウイルス)株: Nancy |
| 分子量 | 理論値: 16.584482 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNAMGQQSGA VYVGNYRVVN RHLATSADWQ NCVWESYNRD LLVSTTTAHG CDIIARCQCT TGVYFCASKN KHYPISFEGP GLVEVQESE YYPRRYQSHV LLAAGFSEPG DAGGILRCEH GVIGIVTMGG EGVVGFADIR DLLWLEDDAM EQ UniProtKB: Genome polyprotein |
-分子 #2: Actin-histidine N-methyltransferase
| 分子 | 名称: Actin-histidine N-methyltransferase / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: protein-histidine N-methyltransferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 67.483117 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNAGKKSRVK TQKSGTGATA TVSPKEILNL TSELLQKCSS PAPGPGKEWE EYVQIRTLVE KIRKKQKGLS VTFDGKREDY FPDLMKWAS ENGASVEGFE MVNFKEEGFG LRATRDIKAE ELFLWVPRKL LMTVESAKNS VLGPLYSQDR ILQAMGNIAL A FHLLCERA ...文字列: SNAGKKSRVK TQKSGTGATA TVSPKEILNL TSELLQKCSS PAPGPGKEWE EYVQIRTLVE KIRKKQKGLS VTFDGKREDY FPDLMKWAS ENGASVEGFE MVNFKEEGFG LRATRDIKAE ELFLWVPRKL LMTVESAKNS VLGPLYSQDR ILQAMGNIAL A FHLLCERA SPNSFWQPYI QTLPSEYDTP LYFEEDEVRY LQSTQAIHDV FSQYKNTARQ YAYFYKVIQT HPHANKLPLK DS FTYEDYR WAVSSVMTRQ NQIPTEDGSR VTLALIPLWD MCNHTNGLIT TGYNLEDDRC ECVALQDFRA GEQIYIFYGT RSN AEFVIH SGFFFDNNSH DRVKIKLGVS KSDRLYAMKA EVLARAGIPT SSVFALHFTE PPISAQLLAF LRVFCMTEEE LKEH LLGDS AIDRIFTLGN SEFPVSWDNE VKLWTFLEDR ASLLLKTYKT TIEEDKSVLK NHDLSVRAKM AIKLRLGEKE ILEKA VKSA AVNREYYRQQ MEEKAPLPKY EESNLGLLES SVGDSRLPLV LRNLEEEAGV QDALNIREAI SKAKATENGL VNGENS IPN GTRSENESLN QESKRAVEDA KGSSSDSTAG VKE UniProtKB: Actin-histidine N-methyltransferase |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #4: S-ADENOSYL-L-HOMOCYSTEINE
| 分子 | 名称: S-ADENOSYL-L-HOMOCYSTEINE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: SAH |
|---|---|
| 分子量 | 理論値: 384.411 Da |
| Chemical component information |  ChemComp-SAH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.34 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.2 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 詳細: 17mA | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 1200 / 平均露光時間: 6.0 sec. / 平均電子線量: 68.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-7lms: |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X