+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2161 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
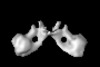
| タイトル | Structure of the yeast F1Fo-ATP synthase dimer | |||||||||
 マップデータ マップデータ | Subtomgram average of Yeast ATP synthase dimers in mitochondrial membranes | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mitochondria / Subtomogram average | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / : / Formation of ATP by chemiosmotic coupling / Cristae formation / Mitochondrial protein degradation / Mitochondrial protein degradation / : / : / proton transmembrane transporter activity / proton motive force-driven ATP synthesis ...: / : / Formation of ATP by chemiosmotic coupling / Cristae formation / Mitochondrial protein degradation / Mitochondrial protein degradation / : / : / proton transmembrane transporter activity / proton motive force-driven ATP synthesis / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase complex / H+-transporting two-sector ATPase / proton-transporting ATP synthase activity, rotational mechanism / ADP binding / mitochondrial intermembrane space / mitochondrial inner membrane / lipid binding / ATP hydrolysis activity / mitochondrion / ATP binding / identical protein binding / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 37.0 Å | |||||||||
 データ登録者 データ登録者 | Davies KM / Kuehlbrandt W | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2012 ジャーナル: Proc Natl Acad Sci U S A / 年: 2012タイトル: Structure of the yeast F1Fo-ATP synthase dimer and its role in shaping the mitochondrial cristae. 著者: Karen M Davies / Claudio Anselmi / Ilka Wittig / José D Faraldo-Gómez / Werner Kühlbrandt /  要旨: We used electron cryotomography of mitochondrial membranes from wild-type and mutant Saccharomyces cerevisiae to investigate the structure and organization of ATP synthase dimers in situ. Subtomogram ...We used electron cryotomography of mitochondrial membranes from wild-type and mutant Saccharomyces cerevisiae to investigate the structure and organization of ATP synthase dimers in situ. Subtomogram averaging of the dimers to 3.7 nm resolution revealed a V-shaped structure of twofold symmetry, with an angle of 86° between monomers. The central and peripheral stalks are well resolved. The monomers interact within the membrane at the base of the peripheral stalks. In wild-type mitochondria ATP synthase dimers are found in rows along the highly curved cristae ridges, and appear to be crucial for membrane morphology. Strains deficient in the dimer-specific subunits e and g or the first transmembrane helix of subunit 4 lack both dimers and lamellar cristae. Instead, cristae are either absent or balloon-shaped, with ATP synthase monomers distributed randomly in the membrane. Computer simulations indicate that isolated dimers induce a plastic deformation in the lipid bilayer, which is partially relieved by their side-by-side association. We propose that the assembly of ATP synthase dimer rows is driven by the reduction in the membrane elastic energy, rather than by direct protein contacts, and that the dimer rows enable the formation of highly curved ridges in mitochondrial cristae. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2161.map.gz emd_2161.map.gz | 538.9 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2161-v30.xml emd-2161-v30.xml emd-2161.xml emd-2161.xml | 14.1 KB 14.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-2161.png EMD-2161.png emd_2161.png emd_2161.png | 1.1 MB 1.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2161 http://ftp.pdbj.org/pub/emdb/structures/EMD-2161 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2161 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2161 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2161_validation.pdf.gz emd_2161_validation.pdf.gz | 178.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2161_full_validation.pdf.gz emd_2161_full_validation.pdf.gz | 177.4 KB | 表示 | |
| XML形式データ |  emd_2161_validation.xml.gz emd_2161_validation.xml.gz | 4.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2161 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2161 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2161 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2161 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2161.map.gz / 形式: CCP4 / 大きさ: 619.1 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2161.map.gz / 形式: CCP4 / 大きさ: 619.1 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomgram average of Yeast ATP synthase dimers in mitochondrial membranes | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.76 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : ATP synthase dimer from mitochondria of Saccharomyces cerevisiae
| 全体 | 名称: ATP synthase dimer from mitochondria of Saccharomyces cerevisiae |
|---|---|
| 要素 |
|
-超分子 #1000: ATP synthase dimer from mitochondria of Saccharomyces cerevisiae
| 超分子 | 名称: ATP synthase dimer from mitochondria of Saccharomyces cerevisiae タイプ: sample / ID: 1000 詳細: Average of subvolumes extracted from tomograms of mitochondrial membrane fragments Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 1.2 MDa |
-超分子 #1: F1Fo-ATP synthase
| 超分子 | 名称: F1Fo-ATP synthase / タイプ: organelle_or_cellular_component / ID: 1 / コピー数: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 1.2 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: 250mM Trehalose, 10nm Tris-HCl pH7.4 |
|---|---|
| グリッド | 詳細: 300 mesh copper grid with quantifoil support film (R2/2), glow discharged |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 100 K / 装置: HOMEMADE PLUNGER / 手法: Single side manual blotting for 5 seconds. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 平均: 80 K |
| アライメント法 | Legacy - 非点収差: Standard procedure |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Tridiem 863 エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 日付 | 2009年3月11日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 1000 (2k x 2k) 平均電子線量: 160 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 24500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 7.0 µm / 最小 デフォーカス(公称値): 7.0 µm / 倍率(公称値): 41000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / Tilt series - Axis1 - Min angle: -50 ° / Tilt series - Axis1 - Max angle: 60 ° |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Tomogram reconstruction performed in IMOD. Average number of tilts used in the 3D reconstructions: 75. Average tomographic tilt angle increment: 1.5. |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 37.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称:  IMOD / 詳細: Final map calculated from 121 subvolumes IMOD / 詳細: Final map calculated from 121 subvolumes |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D / Chain - #4 - Chain ID: E / Chain - #5 - Chain ID: F / Chain - #6 - Chain ID: G / Chain - #7 - Chain ID: H / Chain - #8 - Chain ID: I / Chain - #9 - Chain ID: J / Chain - #10 - Chain ID: K / Chain - #11 - Chain ID: L / Chain - #12 - Chain ID: M / Chain - #13 - Chain ID: N / Chain - #14 - Chain ID: O / Chain - #15 - Chain ID: P / Chain - #16 - Chain ID: Q / Chain - #17 - Chain ID: R / Chain - #18 - Chain ID: S |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: Rigid body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-4b2q: |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: Rigid body with sequential fit command. Chain A extended using residues 180-207 of chain T from 2WSS |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-4b2q: |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)