+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21023 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single-particle cryo-EM reconstruction of rabbit muscle aldolase using 200 keV | |||||||||
 マップデータ マップデータ | Single-particle cryo-EM reconstruction of rabbit muscle aldolase | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | homo-4-mer / lyase / D-fructose / glycolysis | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of Arp2/3 complex-mediated actin nucleation / fructose-bisphosphate aldolase / fructose-bisphosphate aldolase activity / M band / I band / fructose 1,6-bisphosphate metabolic process / glycolytic process / protein homotetramerization / positive regulation of cell migration / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.13 Å | |||||||||
 データ登録者 データ登録者 | Wu M / Lander GC | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Struct Biol X / 年: 2020 ジャーナル: J Struct Biol X / 年: 2020タイトル: Sub-2 Angstrom resolution structure determination using single-particle cryo-EM at 200 keV. 著者: Mengyu Wu / Gabriel C Lander / Mark A Herzik /  要旨: Although the advent of direct electron detectors (DEDs) and software developments have enabled the routine use of single-particle cryogenic electron microscopy (cryo-EM) for structure determination ...Although the advent of direct electron detectors (DEDs) and software developments have enabled the routine use of single-particle cryogenic electron microscopy (cryo-EM) for structure determination of well-behaved specimens to high-resolution, there nonetheless remains a discrepancy between the resolutions attained for biological specimens and the information limits of modern transmission electron microscopes (TEMs). Instruments operating at 300 kV equipped with DEDs are the current paradigm for high-resolution single-particle cryo-EM, while 200 kV TEMs remain comparatively underutilized for purposes beyond sample screening. Here, we expand upon our prior work and demonstrate that one such 200 kV microscope, the Talos Arctica, equipped with a K2 DED is capable of determining structures of macromolecules to as high as ∼1.7 Å resolution. At this resolution, ordered water molecules are readily assigned and holes in aromatic residues can be clearly distinguished in the reconstructions. This work emphasizes the utility of 200 kV electrons for high-resolution single-particle cryo-EM and applications such as structure-based drug design. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21023.map.gz emd_21023.map.gz | 59.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21023-v30.xml emd-21023-v30.xml emd-21023.xml emd-21023.xml | 21.1 KB 21.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21023_fsc.xml emd_21023_fsc.xml | 18.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21023.png emd_21023.png | 231.9 KB | ||
| マスクデータ |  emd_21023_msk_1.map emd_21023_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-21023.cif.gz emd-21023.cif.gz | 6.6 KB | ||
| その他 |  emd_21023_additional.map.gz emd_21023_additional.map.gz emd_21023_half_map_1.map.gz emd_21023_half_map_1.map.gz emd_21023_half_map_2.map.gz emd_21023_half_map_2.map.gz | 409.7 MB 410.9 MB 411.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21023 http://ftp.pdbj.org/pub/emdb/structures/EMD-21023 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21023 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21023 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21023_validation.pdf.gz emd_21023_validation.pdf.gz | 920.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21023_full_validation.pdf.gz emd_21023_full_validation.pdf.gz | 920.3 KB | 表示 | |
| XML形式データ |  emd_21023_validation.xml.gz emd_21023_validation.xml.gz | 25.6 KB | 表示 | |
| CIF形式データ |  emd_21023_validation.cif.gz emd_21023_validation.cif.gz | 32.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21023 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21023 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21023 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21023 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6v20MC  6v21C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10338 (タイトル: Rabbit muscle aldolase movies obtained using a Talos Arctica (200 kV) equipped with a K2 EMPIAR-10338 (タイトル: Rabbit muscle aldolase movies obtained using a Talos Arctica (200 kV) equipped with a K2Data size: 543.3 Data #1: Rabbit muscle aldolase movies obtained using a Talos Arctica (200 kV) equipped with a K2 [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21023.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21023.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Single-particle cryo-EM reconstruction of rabbit muscle aldolase | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.562 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
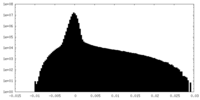
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_21023_msk_1.map emd_21023_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
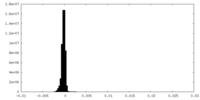
| 密度ヒストグラム |
-追加マップ: Unsharpened map
| ファイル | emd_21023_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
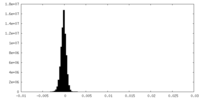
| 密度ヒストグラム |
-ハーフマップ: Odd half map
| ファイル | emd_21023_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Odd half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Even half map
| ファイル | emd_21023_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Even half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Aldolase from rabbit muscle
| 全体 | 名称: Aldolase from rabbit muscle |
|---|---|
| 要素 |
|
-超分子 #1: Aldolase from rabbit muscle
| 超分子 | 名称: Aldolase from rabbit muscle / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Lyophilized rabbit muscle aldolase purchased from Sigma Aldrich was further purified to homogeneity. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 150 KDa |
-分子 #1: Fructose-bisphosphate aldolase A
| 分子 | 名称: Fructose-bisphosphate aldolase A / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: fructose-bisphosphate aldolase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 37.302602 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HSHPALTPEQ KKELSDIAHR IVAPGKGILA ADESTGSIAK RLQSIGTENT EENRRFYRQL LLTADDRVNP CIGGVILFHE TLYQKADDG RPFPQVIKSK GGVVGIKVDK GVVPLAGTNG ETTTQGLDGL SERCAQYKKD GADFAKWRCV LKIGEHTPSA L AIMENANV ...文字列: HSHPALTPEQ KKELSDIAHR IVAPGKGILA ADESTGSIAK RLQSIGTENT EENRRFYRQL LLTADDRVNP CIGGVILFHE TLYQKADDG RPFPQVIKSK GGVVGIKVDK GVVPLAGTNG ETTTQGLDGL SERCAQYKKD GADFAKWRCV LKIGEHTPSA L AIMENANV LARYASICQQ NGIVPIVEPE ILPDGDHDLK RCQYVTEKVL AAVYKALSDH HIYLEGTLLK PNMVTPGHAC TQ KYSHEEI AMATVTALRR TVPPAVTGVT FLSGGQSEEE ASINLNAINK CPLLKPWALT FSYGRALQAS ALKAWGGKKE NLK AAQEEY VKRALANSLA CQGKYTP UniProtKB: Fructose-bisphosphate aldolase A |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 328 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.6 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 7 sec. / 前処理 - 雰囲気: OTHER / 詳細: 15 Watts | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: HOMEMADE PLUNGER 詳細: 3 uL of sample/grid was manually blotted for 4 seconds prior to immediate plunge-freezing in liquid nitrogen-cooled ethane.. | |||||||||
| 詳細 | Reconstituted from lyophilized form (Sigma Aldrich) |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - 画像ごとのフレーム数: 1-44 / 撮影したグリッド数: 1 / 実像数: 3905 / 平均露光時間: 11.0 sec. / 平均電子線量: 67.0 e/Å2 詳細: Images were collected using stage position navigation to target exposure. |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 73000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Starting model was generated by stripping PDB entry 5VY5 of all ligands and alternate conformations, then refining into the EM density using imposed symmetry while adjusting weighting/scoring according to estimated map resolution. The top 10 generated models (ranked based on quality metrics) were real-space refined using Phenix software. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 30 |
| 得られたモデル |  PDB-6v20: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)