+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20261 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single Particle Electron Cryo-Microscopy Reconstruction of BG505 SOSIP-I53-50NP | |||||||||||||||
 マップデータ マップデータ | BG505 SOSIP-I53-50NP | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / apoptotic process / host cell plasma membrane / structural molecule activity / virion membrane / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 | synthetic construct (人工物) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.5 Å | |||||||||||||||
 データ登録者 データ登録者 | Berndsen ZT / Nieusma T / Ward AB | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Enhancing and shaping the immunogenicity of native-like HIV-1 envelope trimers with a two-component protein nanoparticle. 著者: Philip J M Brouwer / Aleksandar Antanasijevic / Zachary Berndsen / Anila Yasmeen / Brooke Fiala / Tom P L Bijl / Ilja Bontjer / Jacob B Bale / William Sheffler / Joel D Allen / Anna Schorcht ...著者: Philip J M Brouwer / Aleksandar Antanasijevic / Zachary Berndsen / Anila Yasmeen / Brooke Fiala / Tom P L Bijl / Ilja Bontjer / Jacob B Bale / William Sheffler / Joel D Allen / Anna Schorcht / Judith A Burger / Miguel Camacho / Daniel Ellis / Christopher A Cottrell / Anna-Janina Behrens / Marco Catalano / Iván Del Moral-Sánchez / Thomas J Ketas / Celia LaBranche / Marit J van Gils / Kwinten Sliepen / Lance J Stewart / Max Crispin / David C Montefiori / David Baker / John P Moore / Per Johan Klasse / Andrew B Ward / Neil P King / Rogier W Sanders /    要旨: The development of native-like HIV-1 envelope (Env) trimer antigens has enabled the induction of neutralizing antibody (NAb) responses against neutralization-resistant HIV-1 strains in animal models. ...The development of native-like HIV-1 envelope (Env) trimer antigens has enabled the induction of neutralizing antibody (NAb) responses against neutralization-resistant HIV-1 strains in animal models. However, NAb responses are relatively weak and narrow in specificity. Displaying antigens in a multivalent fashion on nanoparticles (NPs) is an established strategy to increase their immunogenicity. Here we present the design and characterization of two-component protein NPs displaying 20 stabilized SOSIP trimers from various HIV-1 strains. The two-component nature permits the incorporation of exclusively well-folded, native-like Env trimers into NPs that self-assemble in vitro with high efficiency. Immunization studies show that the NPs are particularly efficacious as priming immunogens, improve the quality of the Ab response over a conventional one-component nanoparticle system, and are most effective when SOSIP trimers with an apex-proximate neutralizing epitope are displayed. Their ability to enhance and shape the immunogenicity of SOSIP trimers make these NPs a promising immunogen platform. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20261.map.gz emd_20261.map.gz | 772 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20261-v30.xml emd-20261-v30.xml emd-20261.xml emd-20261.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20261_fsc.xml emd_20261_fsc.xml | 21.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20261.png emd_20261.png | 132 KB | ||
| マスクデータ |  emd_20261_msk_1.map emd_20261_msk_1.map | 824 MB |  マスクマップ マスクマップ | |
| その他 |  emd_20261_half_map_1.map.gz emd_20261_half_map_1.map.gz emd_20261_half_map_2.map.gz emd_20261_half_map_2.map.gz | 666.4 MB 669.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20261 http://ftp.pdbj.org/pub/emdb/structures/EMD-20261 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20261 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20261 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20261_validation.pdf.gz emd_20261_validation.pdf.gz | 798.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20261_full_validation.pdf.gz emd_20261_full_validation.pdf.gz | 798.5 KB | 表示 | |
| XML形式データ |  emd_20261_validation.xml.gz emd_20261_validation.xml.gz | 29.3 KB | 表示 | |
| CIF形式データ |  emd_20261_validation.cif.gz emd_20261_validation.cif.gz | 39.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20261 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20261 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20261 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20261 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20261.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20261.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BG505 SOSIP-I53-50NP | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_20261_msk_1.map emd_20261_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
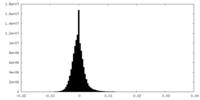
| 密度ヒストグラム |
-ハーフマップ: BG505 SOSIP-I53-50NP, half map 1
| ファイル | emd_20261_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BG505 SOSIP-I53-50NP, half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
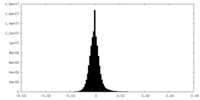
| 密度ヒストグラム |
-ハーフマップ: BG505 SOSIP-I53-50NP, half map 2
| ファイル | emd_20261_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BG505 SOSIP-I53-50NP, half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Two-component self-assembling nanoparticle consisting of BG505 SO...
| 全体 | 名称: Two-component self-assembling nanoparticle consisting of BG505 SOSIP-I53-50A.1NT1 and I53-50B.4PT1 |
|---|---|
| 要素 |
|
-超分子 #1: Two-component self-assembling nanoparticle consisting of BG505 SO...
| 超分子 | 名称: Two-component self-assembling nanoparticle consisting of BG505 SOSIP-I53-50A.1NT1 and I53-50B.4PT1 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: I53-50A.1NT1
| 超分子 | 名称: I53-50A.1NT1 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293F Homo sapiens (ヒト) / 組換細胞: HEK293F |
-超分子 #3: I53-50B.4PT1
| 超分子 | 名称: I53-50B.4PT1 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種: |
-分子 #1: I53-50A.1NT1
| 分子 | 名称: I53-50A.1NT1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 99.386711 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARAENLW VTVYYGVPVW KDAETTLFCA SDAKAYETKK HNVWATHCCV PTDPNPQEI HLENVTEEFN MWKNNMVEQM HTDIISLWDQ SLKPCVKLTP LCVTLQCTNV TNNITDDMRG ELKNCSFNMT T ELRDKKQK ...文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARAENLW VTVYYGVPVW KDAETTLFCA SDAKAYETKK HNVWATHCCV PTDPNPQEI HLENVTEEFN MWKNNMVEQM HTDIISLWDQ SLKPCVKLTP LCVTLQCTNV TNNITDDMRG ELKNCSFNMT T ELRDKKQK VYSLFYRLDV VQINENQGNR SNNSNKEYRL INCNTSAITQ ACPKVSFEPI PIHYCAPAGF AILKCKDKKF NG TGPCPSV STVQCTHGIK PVVSTQLLLN GSLAEEEVMI RSENITNNAK NILVQFNTPV QINCTRPNNN TRKSIRIGPG QWF YATGDI IGDIRQAHCN VSKATWNETL GKVVKQLRKH FGNNTIIRFA NSSGGDLEVT THSFNCGGEF FYCNTSGLFN STWI SNTSV QGSNSTGSND SITLPCRIKQ IINMWQRIGQ AMYAPPIQGV IRCVSNITGL ILTRDGGSTN STTETFRPGG GDMRD NWRS ELYKYKVVKI EPLGVAPTRC KRRVVGRRRR RRAVGIGAVF LGFLGAAGST MGAASMTLTV QARNLLSGIV QQQSNL LRA PECQQHLLKL TVWGIKQLQA RVLAVERYLR DQQLLGIWGC SGKLICCTNV PWNSSWSNRN LSEIWDNMTW LQWDKEI SN YTQIIYGLLE ESQNQQEKNE QDLLALDGSG GSGGSGGSGG SEKAAKAEEA ARKMEELFKK HKIVAVLRAN SVEEAIEK A VAVFAGGVHL IEITFTVPDA DTVIKALSVL KEKGAIIGAG TVTSVEQCRK AVESGAEFIV SPHLDEEISQ FCKEKGVFY MPGVMTPTEL VKAMKLGHDI LKLFPGEVVG PEFVKAMKGP FPNVKFVPTG GVDLDNVCEW FDAGVLAVGV GDALVEGDPD EVREKAKEF VEKIRGCTE |
-分子 #2: I53-50B.4PT1
| 分子 | 名称: I53-50B.4PT1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 18.236877 KDa |
| 組換発現 | 生物種: |
| 配列 | 文字列: MNQHSHKDHE TVRIAVVRAR WHAEIVDACV SAFEAAMRDI GGDRFAVDVF DVPGAYEIPL HARTLAETGR YGAVLGTAFV VNGGIYRHE FVASAVINGM MNVQLNTGVP VLSAVLTPHN YDKSKAHTLL FLALFAVKGM EAARACVEIL AAREKIAAGS L EHHHHHH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 平均露光時間: 8.0 sec. / 平均電子線量: 10.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-6p6f: |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X