[日本語] English
 万見
万見- EMDB-19962: Archaellum filament from the Halobacterium salinarum deltaAgl27 strain -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Archaellum filament from the Halobacterium salinarum deltaAgl27 strain | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | archaellum / haloarcheon / archaellin / STRUCTURAL PROTEIN | |||||||||
| 生物種 |  Halobacterium salinarum (好塩性) Halobacterium salinarum (好塩性) | |||||||||
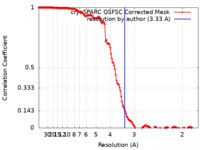
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.33 Å | |||||||||
 データ登録者 データ登録者 | Grossman-Haham I / Shahar A | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Perturbed N-glycosylation of Halobacterium salinarum archaellum filaments leads to filament bundling and compromised cell motility. 著者: Shahar Sofer / Zlata Vershinin / Leen Mashni / Ran Zalk / Anat Shahar / Jerry Eichler / Iris Grossman-Haham /  要旨: The swimming device of archaea-the archaellum-presents asparagine (N)-linked glycans. While N-glycosylation serves numerous roles in archaea, including enabling their survival in extreme ...The swimming device of archaea-the archaellum-presents asparagine (N)-linked glycans. While N-glycosylation serves numerous roles in archaea, including enabling their survival in extreme environments, how this post-translational modification contributes to cell motility remains under-explored. Here, we report the cryo-EM structure of archaellum filaments from the haloarchaeon Halobacterium salinarum, where archaellins, the building blocks of the archaellum, are N-glycosylated, and the N-glycosylation pathway is well-resolved. We further determined structures of archaellum filaments from two N-glycosylation mutant strains that generate truncated glycans and analyzed their motility. While cells from the parent strain exhibited unidirectional motility, the N-glycosylation mutant strain cells swam in ever-changing directions within a limited area. Although these mutant strain cells presented archaellum filaments that were highly similar in architecture to those of the parent strain, N-linked glycan truncation greatly affected interactions between archaellum filaments, leading to dramatic clustering of both isolated and cell-attached filaments. We propose that the N-linked tetrasaccharides decorating archaellins act as physical spacers that minimize the archaellum filament aggregation that limits cell motility. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19962.map.gz emd_19962.map.gz | 141.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19962-v30.xml emd-19962-v30.xml emd-19962.xml emd-19962.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19962_fsc.xml emd_19962_fsc.xml | 13.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19962.png emd_19962.png | 162.3 KB | ||
| Filedesc metadata |  emd-19962.cif.gz emd-19962.cif.gz | 5.8 KB | ||
| その他 |  emd_19962_additional_1.map.gz emd_19962_additional_1.map.gz emd_19962_half_map_1.map.gz emd_19962_half_map_1.map.gz emd_19962_half_map_2.map.gz emd_19962_half_map_2.map.gz | 233.9 MB 255.2 MB 255.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19962 http://ftp.pdbj.org/pub/emdb/structures/EMD-19962 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19962 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19962 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19962_validation.pdf.gz emd_19962_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19962_full_validation.pdf.gz emd_19962_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_19962_validation.xml.gz emd_19962_validation.xml.gz | 22.9 KB | 表示 | |
| CIF形式データ |  emd_19962_validation.cif.gz emd_19962_validation.cif.gz | 29.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19962 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19962 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19962 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19962 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19962.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19962.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.89 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_19962_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_19962_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_19962_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Archaellum filament
| 全体 | 名称: Archaellum filament |
|---|---|
| 要素 |
|
-超分子 #1: Archaellum filament
| 超分子 | 名称: Archaellum filament / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Halobacterium salinarum (好塩性) Halobacterium salinarum (好塩性) |
| 分子量 | 理論値: 20.466 KDa |
-分子 #1: Archaellin
| 分子 | 名称: Archaellin / タイプ: protein_or_peptide / ID: 1 詳細: Since Hbt. salinarum encodes five archaellins (i.e., FlaA1, FlaA2, FlaB1, FlaB2, and FlaB3) and their arrangement within the archaellum filaments is unknown, we further refined the cryo-EM ...詳細: Since Hbt. salinarum encodes five archaellins (i.e., FlaA1, FlaA2, FlaB1, FlaB2, and FlaB3) and their arrangement within the archaellum filaments is unknown, we further refined the cryo-EM map without applying symmetry, in an attempt to resolve the positions of these archaellins within the filament, as done previously with reconstruction of the Methanocaldococcus villosus archaellum. Symmetry-free refinement improved the overall resolution map to 3.1 A and revealed differences in density among archaellin subunits. Nonetheless, we were unable to identify features that would allow us to unambiguously assign specific archaellins into the density, perhaps because the regions that distinguish each archaellin are few, short, and mostly predicted to lack defined secondary structure, or because the five archaellins are not organized in a repeating pattern. Consequently, we built a model into the central region of the cryo-EM map comprising 26 archaellin subunits that share a consensus sequence, in which identical residues among the five archaellins are explicitly modelled, with those variable residues usually being modelled as alanine residues (UNK). コピー数: 25 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Halobacterium salinarum (好塩性) Halobacterium salinarum (好塩性) |
| 分子量 | 理論値: 20.107971 KDa |
| 配列 | 文字列: MFEFITDEDE RGQVGIGTLI VFIAMVLVAA IAAGVLINTA GYLQSKGSAT GEEASAQVSN RINIVSAYGN V(UNK) (UNK)(UNK)(UNK)VDYV NLTVRQAAGA DNINL(UNK)KSTI QWIGPD(UNK)ATT LTY(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK) ...文字列: MFEFITDEDE RGQVGIGTLI VFIAMVLVAA IAAGVLINTA GYLQSKGSAT GEEASAQVSN RINIVSAYGN V(UNK) (UNK)(UNK)(UNK)VDYV NLTVRQAAGA DNINL(UNK)KSTI QWIGPD(UNK)ATT LTY(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)ENFTT(UNK)S IKG(UNK)(UNK)(UNK)(UNK)VLV DQSDRIKVIM YA(UNK) (UNK)V(UNK)(UNK)(UNK)L (UNK)(UNK)G(UNK)EVQLTV TTQYGSKTTY WAQVPESLKD KNAV(UNK)L |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: OTHER / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)