+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E.coli DNA gyrase in complex with 217 bp substrate DNA and LEI-800 | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | TOPOISOMERASE / GYRASE / ALLOSTERIC INHIBITOR / ANTIBIOTIC / DNA BINDING PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA negative supercoiling activity / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) complex / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) activity / DNA topoisomerase (ATP-hydrolysing) / DNA topological change / ATP-dependent activity, acting on DNA / DNA-templated DNA replication / chromosome / response to xenobiotic stimulus / response to antibiotic ...DNA negative supercoiling activity / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) complex / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) activity / DNA topoisomerase (ATP-hydrolysing) / DNA topological change / ATP-dependent activity, acting on DNA / DNA-templated DNA replication / chromosome / response to xenobiotic stimulus / response to antibiotic / DNA-templated transcription / DNA binding / ATP binding / metal ion binding / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) | |||||||||||||||
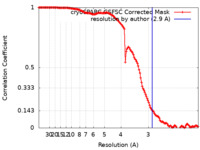
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||||||||
 データ登録者 データ登録者 | Ghilarov D / Martin NI / van der Stelt M | |||||||||||||||
| 資金援助 |  英国, European Union, 英国, European Union,  オランダ, 4件 オランダ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Chem / 年: 2024 ジャーナル: Nat Chem / 年: 2024タイトル: Discovery of isoquinoline sulfonamides as allosteric gyrase inhibitors with activity against fluoroquinolone-resistant bacteria. 著者: Alexander T Bakker / Ioli Kotsogianni / Mariana Avalos / Jeroen M Punt / Bing Liu / Diana Piermarini / Berend Gagestein / Cornelis J Slingerland / Le Zhang / Joost J Willemse / Leela B ...著者: Alexander T Bakker / Ioli Kotsogianni / Mariana Avalos / Jeroen M Punt / Bing Liu / Diana Piermarini / Berend Gagestein / Cornelis J Slingerland / Le Zhang / Joost J Willemse / Leela B Ghimire / Richard J H B N van den Berg / Antonius P A Janssen / Tom H M Ottenhoff / Constant A A van Boeckel / Gilles P van Wezel / Dmitry Ghilarov / Nathaniel I Martin / Mario van der Stelt /   要旨: Bacteria have evolved resistance to nearly all known antibacterials, emphasizing the need to identify antibiotics that operate via novel mechanisms. Here we report a class of allosteric inhibitors of ...Bacteria have evolved resistance to nearly all known antibacterials, emphasizing the need to identify antibiotics that operate via novel mechanisms. Here we report a class of allosteric inhibitors of DNA gyrase with antibacterial activity against fluoroquinolone-resistant clinical isolates of Escherichia coli. Screening of a small-molecule library revealed an initial isoquinoline sulfonamide hit, which was optimized via medicinal chemistry efforts to afford the more potent antibacterial LEI-800. Target identification studies, including whole-genome sequencing of in vitro selected mutants with resistance to isoquinoline sulfonamides, unanimously pointed to the DNA gyrase complex, an essential bacterial topoisomerase and an established antibacterial target. Using single-particle cryogenic electron microscopy, we determined the structure of the gyrase-LEI-800-DNA complex. The compound occupies an allosteric, hydrophobic pocket in the GyrA subunit and has a mode of action that is distinct from the clinically used fluoroquinolones or any other gyrase inhibitor reported to date. LEI-800 provides a chemotype suitable for development to counter the increasingly widespread bacterial resistance to fluoroquinolones. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18592.map.gz emd_18592.map.gz | 230.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18592-v30.xml emd-18592-v30.xml emd-18592.xml emd-18592.xml | 23.2 KB 23.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18592_fsc.xml emd_18592_fsc.xml | 13.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18592.png emd_18592.png | 47.9 KB | ||
| Filedesc metadata |  emd-18592.cif.gz emd-18592.cif.gz | 7.5 KB | ||
| その他 |  emd_18592_additional_1.map.gz emd_18592_additional_1.map.gz emd_18592_half_map_1.map.gz emd_18592_half_map_1.map.gz emd_18592_half_map_2.map.gz emd_18592_half_map_2.map.gz | 204.7 MB 226.3 MB 226.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18592 http://ftp.pdbj.org/pub/emdb/structures/EMD-18592 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18592 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18592 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18592_validation.pdf.gz emd_18592_validation.pdf.gz | 985.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18592_full_validation.pdf.gz emd_18592_full_validation.pdf.gz | 985.5 KB | 表示 | |
| XML形式データ |  emd_18592_validation.xml.gz emd_18592_validation.xml.gz | 22.2 KB | 表示 | |
| CIF形式データ |  emd_18592_validation.cif.gz emd_18592_validation.cif.gz | 28.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18592 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18592 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18592 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18592 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8qqiMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18592.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18592.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.02 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_18592_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18592_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18592_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of E.coli DNA gyrase with 217 bp linear dsDNA fra...
| 全体 | 名称: Ternary complex of E.coli DNA gyrase with 217 bp linear dsDNA fragment and LEI-800 |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of E.coli DNA gyrase with 217 bp linear dsDNA fra...
| 超分子 | 名称: Ternary complex of E.coli DNA gyrase with 217 bp linear dsDNA fragment and LEI-800 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: DNA gyrase subunit A
| 分子 | 名称: DNA gyrase subunit A / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 58.824207 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SSDLAREITP VNIEEELKSS YLDYAMSVIV GRALPDVRDG LKPVHRRVLY AMNVLGNDWN KAYKKSARVV GDVIGKYHPH GDSAVYDTI VRMAQPFSLR YMLVDGQGNF GSIDGDSAAA MRYTEIRLAK IAHELMADLE KETVDFVDNY DGTEKIPDVM P TKIPNLLV ...文字列: SSDLAREITP VNIEEELKSS YLDYAMSVIV GRALPDVRDG LKPVHRRVLY AMNVLGNDWN KAYKKSARVV GDVIGKYHPH GDSAVYDTI VRMAQPFSLR YMLVDGQGNF GSIDGDSAAA MRYTEIRLAK IAHELMADLE KETVDFVDNY DGTEKIPDVM P TKIPNLLV NGSSGIAVGM ATNIPPHNLT EVINGCLAYI DDEDISIEGL MEHIPGPDFP TAAIINGRRG IEEAYRTGRG KV YIRARAE VEVDAKTGRE TIIVHEIPYQ VNKARLIEKI AELVKEKRVE GISALRDESD KDGMRIVIEV KRDAVGEVVL NNL YSQTQL QVSFGINMVA LHHGQPKIMN LKDIIAAFVR HRREVVTRRT IFELRKARDR AHILEALAVA LANIDPIIEL IRHA PTPAE AKTALVANPW QLGNVAAMLE RAGDDAARPE WLEPEFGVRD GLYYLTEQQA QAILDLRLQK LTGLEHEKLL DEYKE LLDQ IAELLRILGS ADRLMEVIRE ELELVREQFG DKRRTEITAN UniProtKB: DNA gyrase subunit A |
-分子 #2: DNA gyrase subunit B
| 分子 | 名称: DNA gyrase subunit B / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO / EC番号: DNA topoisomerase (ATP-hydrolysing) |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 89.641391 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPSNSYDSSS IKVLKGLDAV RKRPGMYIGD TDDGTGLHHM VFEVVDNAID EALAGHCKEI IVTIHADNSV SVQDDGRGIP TGIHPEEGV SAAEVIMTVL HAGGKFDDNS YKVSGGLHGV GVSVVNALSQ KLELVIQREG KIHRQIYEHG VPQAPLAVTG E TEKTGTMV ...文字列: GPSNSYDSSS IKVLKGLDAV RKRPGMYIGD TDDGTGLHHM VFEVVDNAID EALAGHCKEI IVTIHADNSV SVQDDGRGIP TGIHPEEGV SAAEVIMTVL HAGGKFDDNS YKVSGGLHGV GVSVVNALSQ KLELVIQREG KIHRQIYEHG VPQAPLAVTG E TEKTGTMV RFWPSLETFT NVTEFEYEIL AKRLRELSFL NSGVSIRLRD KRDGKEDHFH YEGGIKAFVE YLNKNKTPIH PN IFYFSTE KDGIGVEVAL QWNDGFQENI YCFTNNIPQR DGGTHLAGFR AAMTRTLNAY MDKEGYSKKA KVSATGDDAR EGL IAVVSV KVPDPKFSSQ TKDKLVSSEV KSAVEQQMNE LLAEYLLENP TDAKIVVGKI IDAARAREAA RRAREMTRRK GALD LAGLP GKLADCQERD PALSELYLVE GDSAGGSAKQ GRNRKNQAIL PLKGKILNVE KARFDKMLSS QEVATLITAL GCGIG RDEY NPDKLRYHSI IIMTDADVDG SHIRTLLLTF FYRQMPEIVE RGHVYIAQPP LYKVKKGKQE QYIKDDEAMD QYQISI ALD GATLHTNASA PALAGEALEK LVSEYNATQK MINRMERRYP KAMLKELIYQ PTLTEADLSD EQTVTRWVNA LVSELND KE QHGSQWKFDV HTNAEQNLFE PIVRVRTHGV DTDYPLDHEF ITGGEYRRIC TLGEKLRGLL EEDAFIERGE RRQPVASF E QALDWLVKES RRGLSIQRYK GLGEMNPEQL WETTMDPESR RMLRVTVKDA IAADQLFTTL MGDAVEPRRA FIEENALKA A UniProtKB: DNA gyrase subunit B |
-分子 #3: Mu217 chain E
| 分子 | 名称: Mu217 chain E / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) |
| 分子量 | 理論値: 7.943173 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DA)(DG)(DG)(DC)(DA)(DT)(DA) (DA)(DA)(DA)(DT)(DC)(DA)(DC)(DC)(DC)(DG) (DC)(DA)(DC)(DA)(DG)(DA) |
-分子 #4: Mu217 chain F
| 分子 | 名称: Mu217 chain F / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage Mu (ファージ) Escherichia phage Mu (ファージ) |
| 分子量 | 理論値: 8.031157 KDa |
| 配列 | 文字列: (DT)(DC)(DT)(DG)(DT)(DG)(DC)(DG)(DG)(DG) (DT)(DG)(DA)(DT)(DT)(DT)(DT)(DA)(DT)(DG) (DC)(DC)(DT)(DG)(DA)(DT) |
-分子 #5: ~{N}-[[(2~{S},5~{R})-5-(4-pyridin-3-ylphenyl)pyrrolidin-2-yl]meth...
| 分子 | 名称: ~{N}-[[(2~{S},5~{R})-5-(4-pyridin-3-ylphenyl)pyrrolidin-2-yl]methyl]isoquinoline-5-sulfonamide タイプ: ligand / ID: 5 / コピー数: 2 / 式: LRL |
|---|---|
| 分子量 | 理論値: 444.549 Da |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 12 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 25 mM Na-HEPES pH 8 KOAc 30 mM MgOAc 2.5 mM TCEP 0.5 mM CHAPSO 8 mM |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | Monodisperse sample |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 39.58 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X