+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Sulfolobus acidocaldarius AAP filament. | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | cell surface appendage / N-glycosylation / PROTEIN FIBRIL | |||||||||
| Function / homology | Immunoglobulin-like fold / membrane / DUF4352 domain-containing protein Function and homology information Function and homology information | |||||||||
| Biological species |   Sulfolobus acidocaldarius (acidophilic) Sulfolobus acidocaldarius (acidophilic) | |||||||||
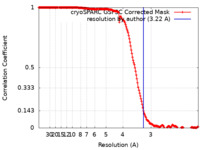
| Method | helical reconstruction / cryo EM / Resolution: 3.22 Å | |||||||||
 Authors Authors | Isupov MN / Gaines M / Daum B / McLaren M | |||||||||
| Funding support | European Union, 1 items
| |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2024 Journal: Nat Commun / Year: 2024Title: CryoEM reveals the structure of an archaeal pilus involved in twitching motility. Authors: Matthew C Gaines / Shamphavi Sivabalasarma / Michail N Isupov / Risat Ul Haque / Mathew McLaren / Cyril Hanus / Vicki A M Gold / Sonja-Verena Albers / Bertram Daum /    Abstract: Amongst the major types of archaeal filaments, several have been shown to closely resemble bacterial homologues of the Type IV pili (T4P). Within Sulfolobales, member species encode for three types ...Amongst the major types of archaeal filaments, several have been shown to closely resemble bacterial homologues of the Type IV pili (T4P). Within Sulfolobales, member species encode for three types of T4P, namely the archaellum, the UV-inducible pilus system (Ups) and the archaeal adhesive pilus (Aap). Whereas the archaellum functions primarily in swimming motility, and the Ups in UV-induced cell aggregation and DNA-exchange, the Aap plays an important role in adhesion and twitching motility. Here, we present a cryoEM structure of the Aap of the archaeal model organism Sulfolobus acidocaldarius. We identify the component subunit as AapB and find that while its structure follows the canonical T4P blueprint, it adopts three distinct conformations within the pilus. The tri-conformer Aap structure that we describe challenges our current understanding of pilus structure and sheds new light on the principles of twitching motility. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_18119.map.gz emd_18119.map.gz | 24.7 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-18119-v30.xml emd-18119-v30.xml emd-18119.xml emd-18119.xml | 16.1 KB 16.1 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_18119_fsc.xml emd_18119_fsc.xml | 16.9 KB | Display |  FSC data file FSC data file |
| Images |  emd_18119.png emd_18119.png | 82.2 KB | ||
| Filedesc metadata |  emd-18119.cif.gz emd-18119.cif.gz | 5.3 KB | ||
| Others |  emd_18119_additional_1.map.gz emd_18119_additional_1.map.gz emd_18119_half_map_1.map.gz emd_18119_half_map_1.map.gz emd_18119_half_map_2.map.gz emd_18119_half_map_2.map.gz | 443.5 MB 475.9 MB 475.9 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18119 http://ftp.pdbj.org/pub/emdb/structures/EMD-18119 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18119 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18119 | HTTPS FTP |
-Related structure data
| Related structure data |  8q30MC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_18119.map.gz / Format: CCP4 / Size: 512 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_18119.map.gz / Format: CCP4 / Size: 512 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.047 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Additional map: #1
| File | emd_18119_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #1
| File | emd_18119_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: DeepEMhance map
| File | emd_18119_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | DeepEMhance map | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : Filament
| Entire | Name: Filament |
|---|---|
| Components |
|
-Supramolecule #1: Filament
| Supramolecule | Name: Filament / type: organelle_or_cellular_component / ID: 1 / Parent: 0 / Macromolecule list: #1 |
|---|---|
| Source (natural) | Organism:   Sulfolobus acidocaldarius (acidophilic) Sulfolobus acidocaldarius (acidophilic) |
-Macromolecule #1: DUF4352 domain-containing protein
| Macromolecule | Name: DUF4352 domain-containing protein / type: protein_or_peptide / ID: 1 / Number of copies: 72 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:   Sulfolobus acidocaldarius (acidophilic) Sulfolobus acidocaldarius (acidophilic) |
| Molecular weight | Theoretical: 14.177022 KDa |
| Sequence | String: LSGAIVALIL VIAGVIIAIA VVLFAFGLIP GISNQGSIQV LGSGTITNST ASGSSRTIYN ITITVKNTGT TSISVTSINI NGQPFNING TAPSIPAGRT QPITFEVTPA SGKPNFSPGA SYTATIYFSN GQGAPATLIY QG UniProtKB: DUF4352 domain-containing protein |
-Macromolecule #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| Macromolecule | Name: 2-acetamido-2-deoxy-beta-D-glucopyranose / type: ligand / ID: 4 / Number of copies: 72 / Formula: NAG |
|---|---|
| Molecular weight | Theoretical: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | helical reconstruction |
| Aggregation state | filament |
- Sample preparation
Sample preparation
| Buffer | pH: 3 |
|---|---|
| Vitrification | Cryogen name: ETHANE |
| Details | Mixed population of filaments isolated from Sulfolobus acidocaldarius |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K2 SUMMIT (4k x 4k) / Detector mode: COUNTING / Number real images: 6272 / Average electron dose: 42.33 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Nominal defocus max: 2.5 µm / Nominal defocus min: 1.0 µm |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
- Image processing
Image processing
-Atomic model buiding 1
| Details | Jligand was used for preparing dictionaries for an unusual sugars |
|---|---|
| Refinement | Space: RECIPROCAL / Protocol: AB INITIO MODEL |
| Output model |  PDB-8q30: |
 Movie
Movie Controller
Controller




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)