[日本語] English
 万見
万見- EMDB-17645: Structure of a heteropolymeric type 4 pilus from a monoderm bacterium -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a heteropolymeric type 4 pilus from a monoderm bacterium | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bacterial pilus Type 4 pilus / PROTEIN FIBRIL | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報establishment of competence for transformation / protein secretion by the type II secretion system / type II protein secretion system complex / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptococcus sanguinis (バクテリア) Streptococcus sanguinis (バクテリア) | |||||||||
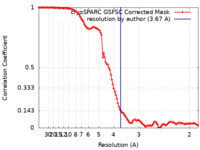
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.67 Å | |||||||||
 データ登録者 データ登録者 | Anger R / Pieulle L / Shahin M / Valette O / Le Guenno H / Kosta A / Pelicic V / Fronzes R | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structure of a heteropolymeric type 4 pilus from a monoderm bacterium. 著者: Robin Anger / Laetitia Pieulle / Meriam Shahin / Odile Valette / Hugo Le Guenno / Artemis Kosta / Vladimir Pelicic / Rémi Fronzes /   要旨: Type 4 pili (T4P) are important virulence factors, which belong to a superfamily of nanomachines ubiquitous in prokaryotes, called type 4 filaments (T4F). T4F are defined as helical polymers of type ...Type 4 pili (T4P) are important virulence factors, which belong to a superfamily of nanomachines ubiquitous in prokaryotes, called type 4 filaments (T4F). T4F are defined as helical polymers of type 4 pilins. Recent advances in cryo-electron microscopy (cryo-EM) led to structures of several T4F, revealing that the long N-terminal α-helix (α1) - the trademark of pilins - packs in the centre of the filaments to form a hydrophobic core. In diderm bacteria - all available bacterial T4F structures are from diderm species - a portion of α1 is melted (unfolded). Here we report that this architecture is conserved in phylogenetically distant monoderm species by determining the structure of Streptococcus sanguinis T4P. Our 3.7 Å resolution cryo-EM structure of S. sanguinis heteropolymeric T4P and the resulting full atomic model including all minor pilins highlight universal features of bacterial T4F and have widespread implications in understanding T4F biology. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17645.map.gz emd_17645.map.gz | 62.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17645-v30.xml emd-17645-v30.xml emd-17645.xml emd-17645.xml | 13 KB 13 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17645_fsc.xml emd_17645_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17645.png emd_17645.png | 58.4 KB | ||
| Filedesc metadata |  emd-17645.cif.gz emd-17645.cif.gz | 4.7 KB | ||
| その他 |  emd_17645_half_map_1.map.gz emd_17645_half_map_1.map.gz emd_17645_half_map_2.map.gz emd_17645_half_map_2.map.gz | 115.9 MB 115.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17645 http://ftp.pdbj.org/pub/emdb/structures/EMD-17645 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17645 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17645 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17645_validation.pdf.gz emd_17645_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17645_full_validation.pdf.gz emd_17645_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_17645_validation.xml.gz emd_17645_validation.xml.gz | 19 KB | 表示 | |
| CIF形式データ |  emd_17645_validation.cif.gz emd_17645_validation.cif.gz | 24.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17645 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17645 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17645 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17645 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8pfbMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17645.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17645.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.93 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_17645_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
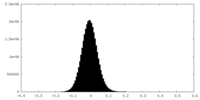
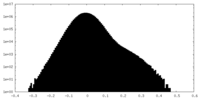
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17645_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
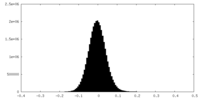
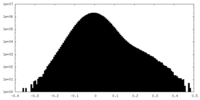
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Type 4 pilus of Streptococcus sanguinis
| 全体 | 名称: Type 4 pilus of Streptococcus sanguinis |
|---|---|
| 要素 |
|
-超分子 #1: Type 4 pilus of Streptococcus sanguinis
| 超分子 | 名称: Type 4 pilus of Streptococcus sanguinis / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Streptococcus sanguinis (バクテリア) Streptococcus sanguinis (バクテリア) |
-分子 #1: Type IV pilin PilE1
| 分子 | 名称: Type IV pilin PilE1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus sanguinis (バクテリア) Streptococcus sanguinis (バクテリア) |
| 分子量 | 理論値: 14.752422 KDa |
| 配列 | 文字列: FTLVELIVVI IIIAIIAAVA IPAITSFQDN ARKSRIQSEH RELVSAIQSY IGAQDDPTNP SEITLAKLAP YMSKNAKNED GIVNSLAKD KSGNSSTSAP GSAHQIDTTN HKLISTFTPS NGGQATVLTY DWSANGVNSN UniProtKB: Type IV pilin PilE1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 50.7 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.0460000000000003 µm 最小 デフォーカス(公称値): 0.492 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X