+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryo-EM structure of human SLC15A4 in outward-open state | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | transporter / lysosome / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報histidine transport / mast cell homeostasis / L-histidine transmembrane export from vacuole / L-histidine transmembrane transporter activity / peptidoglycan transmembrane transporter activity / Proton/oligopeptide cotransporters / regulation of isotype switching to IgG isotypes / positive regulation of toll-like receptor 8 signaling pathway / peptidoglycan transport / positive regulation of toll-like receptor 7 signaling pathway ...histidine transport / mast cell homeostasis / L-histidine transmembrane export from vacuole / L-histidine transmembrane transporter activity / peptidoglycan transmembrane transporter activity / Proton/oligopeptide cotransporters / regulation of isotype switching to IgG isotypes / positive regulation of toll-like receptor 8 signaling pathway / peptidoglycan transport / positive regulation of toll-like receptor 7 signaling pathway / SLC15A4:TASL-dependent IRF5 activation / positive regulation of nucleotide-binding oligomerization domain containing 1 signaling pathway / dipeptide import across plasma membrane / peptide:proton symporter activity / positive regulation of toll-like receptor 9 signaling pathway / dipeptide transmembrane transporter activity / regulation of nucleotide-binding domain, leucine rich repeat containing receptor signaling pathway / positive regulation of nucleotide-binding oligomerization domain containing 2 signaling pathway / endolysosome membrane / positive regulation of innate immune response / specific granule membrane / monoatomic ion transport / protein transport / early endosome membrane / lysosomal membrane / innate immune response / Neutrophil degranulation / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
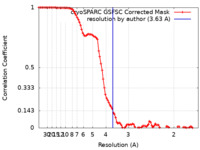
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.63 Å | |||||||||
 データ登録者 データ登録者 | Parker JL / Kato T / Newstead S | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: To be published ジャーナル: To be publishedタイトル: cryo-EM structure of human SLC15A4 PHT1 in outward-open state 著者: Parker JL / Kato T / Newstead S | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17462.map.gz emd_17462.map.gz | 85.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17462-v30.xml emd-17462-v30.xml emd-17462.xml emd-17462.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17462_fsc.xml emd_17462_fsc.xml | 9.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17462.png emd_17462.png | 129.7 KB | ||
| マスクデータ |  emd_17462_msk_1.map emd_17462_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17462.cif.gz emd-17462.cif.gz | 5.4 KB | ||
| その他 |  emd_17462_half_map_1.map.gz emd_17462_half_map_1.map.gz emd_17462_half_map_2.map.gz emd_17462_half_map_2.map.gz | 84.5 MB 84.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17462 http://ftp.pdbj.org/pub/emdb/structures/EMD-17462 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17462 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17462 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17462_validation.pdf.gz emd_17462_validation.pdf.gz | 955.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17462_full_validation.pdf.gz emd_17462_full_validation.pdf.gz | 954.7 KB | 表示 | |
| XML形式データ |  emd_17462_validation.xml.gz emd_17462_validation.xml.gz | 17.8 KB | 表示 | |
| CIF形式データ |  emd_17462_validation.cif.gz emd_17462_validation.cif.gz | 22.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17462 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17462 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17462 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17462 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8p6aMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17462.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17462.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||||||||||||||||||
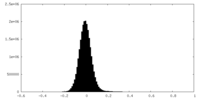
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17462_msk_1.map emd_17462_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
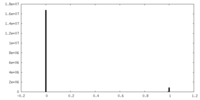
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17462_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
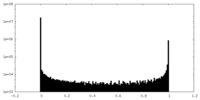
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17462_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
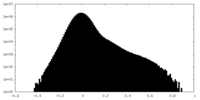
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SLC15A4
| 全体 | 名称: SLC15A4 |
|---|---|
| 要素 |
|
-超分子 #1: SLC15A4
| 超分子 | 名称: SLC15A4 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Solute carrier family 15 member 4
| 分子 | 名称: Solute carrier family 15 member 4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 63.32743 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEGSGGGAGE RAPLLGARRA AAAAAAAGAF AGRRAACGAV LLTELLERAA FYGITSNLVL FLNGAPFCWE GAQASEALLL FMGLTYLGS PFGGWLADAR LGRARAILLS LALYLLGMLA FPLLAAPATR AALCGSARLL NCTAPGPDAA ARCCSPATFA G LVLVGLGV ...文字列: MEGSGGGAGE RAPLLGARRA AAAAAAAGAF AGRRAACGAV LLTELLERAA FYGITSNLVL FLNGAPFCWE GAQASEALLL FMGLTYLGS PFGGWLADAR LGRARAILLS LALYLLGMLA FPLLAAPATR AALCGSARLL NCTAPGPDAA ARCCSPATFA G LVLVGLGV ATVKANITPF GADQVKDRGP EATRRFFNWF YWSINLGAIL SLGGIAYIQQ NVSFVTGYAI PTVCVGLAFV VF LCGQSVF ITKPPDGSAF TDMFKILTYS CCSQKRSGER QSNGEGIGVF QQSSKQSLFD SCKMSHGGPF TEEKVEDVKA LVK IVPVFL ALIPYWTVYF QMQTTYVLQS LHLRIPEISN ITTTPHTLPA AWLTMFDAVL ILLLIPLKDK LVDPILRRHG LLPS SLKRI AVGMFFVMCS AFAAGILESK RLNLVKEKTI NQTIGNVVYH AADLSLWWQV PQYLLIGISE IFASIAGLEF AYSAA PKSM QSAIMGLFFF FSGVGSFVGS GLLALVSIKA IGWMSSHTDF GNINGCYLNY YFFLLAAIQG ATLLLFLIIS VKYDHH RDH QRSRANGVPT SRRALEDYKD DDDK UniProtKB: Solute carrier family 15 member 4 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 44.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-8p6a: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)