+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Bipartite interaction of TOPBP1 with the GINS complex | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Scaffold / Replisome / Recruitment / REPLICATION | |||||||||
| Function / homology |  Function and homology information Function and homology informationUnwinding of DNA / DNA strand elongation involved in mitotic DNA replication / GINS complex / CMG complex / double-strand break repair via break-induced replication / mitotic DNA replication initiation / inner cell mass cell proliferation / isomerase activity / DNA-templated DNA replication / multicellular organism growth ...Unwinding of DNA / DNA strand elongation involved in mitotic DNA replication / GINS complex / CMG complex / double-strand break repair via break-induced replication / mitotic DNA replication initiation / inner cell mass cell proliferation / isomerase activity / DNA-templated DNA replication / multicellular organism growth / spindle pole / cellular senescence / chromosome / DNA replication / DNA repair / centrosome / DNA binding / nucleoplasm / nucleus / cytoplasm Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 4.1 Å | |||||||||
 Authors Authors | Day M / Oliver AW / Pearl LH | |||||||||
| Funding support |  United Kingdom, 1 items United Kingdom, 1 items
| |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2024 Journal: Nat Commun / Year: 2024Title: TopBP1 utilises a bipartite GINS binding mode to support genome replication. Authors: Matthew Day / Bilal Tetik / Milena Parlak / Yasser Almeida-Hernández / Markus Räschle / Farnusch Kaschani / Heike Siegert / Anika Marko / Elsa Sanchez-Garcia / Markus Kaiser / Isabel A ...Authors: Matthew Day / Bilal Tetik / Milena Parlak / Yasser Almeida-Hernández / Markus Räschle / Farnusch Kaschani / Heike Siegert / Anika Marko / Elsa Sanchez-Garcia / Markus Kaiser / Isabel A Barker / Laurence H Pearl / Antony W Oliver / Dominik Boos /   Abstract: Activation of the replicative Mcm2-7 helicase by loading GINS and Cdc45 is crucial for replication origin firing, and as such for faithful genetic inheritance. Our biochemical and structural studies ...Activation of the replicative Mcm2-7 helicase by loading GINS and Cdc45 is crucial for replication origin firing, and as such for faithful genetic inheritance. Our biochemical and structural studies demonstrate that the helicase activator GINS interacts with TopBP1 through two separate binding surfaces, the first involving a stretch of highly conserved amino acids in the TopBP1-GINI region, the second a surface on TopBP1-BRCT4. The two surfaces bind to opposite ends of the A domain of the GINS subunit Psf1. Mutation analysis reveals that either surface is individually able to support TopBP1-GINS interaction, albeit with reduced affinity. Consistently, either surface is sufficient for replication origin firing in Xenopus egg extracts and becomes essential in the absence of the other. The TopBP1-GINS interaction appears sterically incompatible with simultaneous binding of DNA polymerase epsilon (Polε) to GINS when bound to Mcm2-7-Cdc45, although TopBP1-BRCT4 and the Polε subunit PolE2 show only partial competitivity in binding to Psf1. Our TopBP1-GINS model improves the understanding of the recently characterised metazoan pre-loading complex. It further predicts the coordination of three molecular origin firing processes, DNA polymerase epsilon arrival, TopBP1 ejection and GINS integration into Mcm2-7-Cdc45. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_16916.map.gz emd_16916.map.gz | 78.4 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-16916-v30.xml emd-16916-v30.xml emd-16916.xml emd-16916.xml | 22.8 KB 22.8 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_16916.png emd_16916.png | 75.8 KB | ||
| Filedesc metadata |  emd-16916.cif.gz emd-16916.cif.gz | 6.5 KB | ||
| Others |  emd_16916_additional_1.map.gz emd_16916_additional_1.map.gz emd_16916_additional_2.map.gz emd_16916_additional_2.map.gz emd_16916_half_map_1.map.gz emd_16916_half_map_1.map.gz emd_16916_half_map_2.map.gz emd_16916_half_map_2.map.gz | 49.4 MB 65.4 MB 65.4 MB 65.4 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16916 http://ftp.pdbj.org/pub/emdb/structures/EMD-16916 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16916 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16916 | HTTPS FTP |
-Related structure data
| Related structure data |  8ok2MC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_16916.map.gz / Format: CCP4 / Size: 83.7 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_16916.map.gz / Format: CCP4 / Size: 83.7 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.74 Å | ||||||||||||||||||||||||||||||||||||
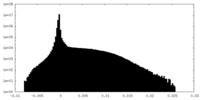
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Additional map: #2
| File | emd_16916_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
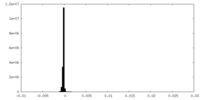
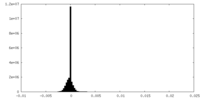
| Density Histograms |
-Additional map: #1
| File | emd_16916_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
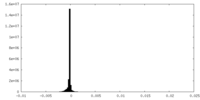
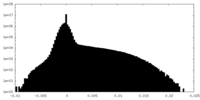
| Density Histograms |
-Half map: #2
| File | emd_16916_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
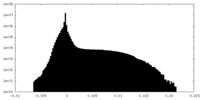
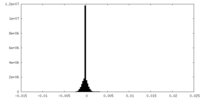
| Density Histograms |
-Half map: #1
| File | emd_16916_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
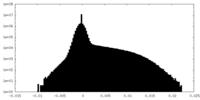
| Density Histograms |
- Sample components
Sample components
-Entire : GINS complex bound to TOPBP1
| Entire | Name: GINS complex bound to TOPBP1 |
|---|---|
| Components |
|
-Supramolecule #1: GINS complex bound to TOPBP1
| Supramolecule | Name: GINS complex bound to TOPBP1 / type: complex / ID: 1 / Parent: 0 / Macromolecule list: all |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
-Macromolecule #1: DNA replication complex GINS protein PSF1
| Macromolecule | Name: DNA replication complex GINS protein PSF1 / type: protein_or_peptide / ID: 1 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 17.772438 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MFCEKAMELI RELHRAPEGQ LPAFNEDGLR QVLEEMKALY EQNQSDVNEA KSGGRSDLIP TIKFRHCSLL RNRRCTVAYL YDRLLRIRA LRWEYGSILP NALRFHMAAE EMEWFNNYKR SLATYMRSLG GDEGLDITQD MKPPKSLYIE VR UniProtKB: DNA replication complex GINS protein PSF1 |
-Macromolecule #2: DNA replication complex GINS protein PSF2
| Macromolecule | Name: DNA replication complex GINS protein PSF2 / type: protein_or_peptide / ID: 2 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 21.453713 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MDAAEVEFLA EKELVTIIPN FSLDKIYLIG GDLGPFNPGL PVEVPLWLAI NLKQRQKCRL LPPEWMDVEK LEKMRDHERK EETFTPMPS PYYMELTKLL LNHASDNIPK ADEIRTLVKD MWDTRIAKLR VSADSFVRQQ EAHAKLDNLT LMEINTSGTF L TQALNHMY KLRTNLQPLE STQSQDF UniProtKB: DNA replication complex GINS protein PSF2 |
-Macromolecule #3: DNA replication complex GINS protein PSF3
| Macromolecule | Name: DNA replication complex GINS protein PSF3 / type: protein_or_peptide / ID: 3 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 24.562611 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MSEAYFRVES GALGPEENFL SLDDILMSHE KLPVRTETAM PRLGAFFLER SAGAETDNAV PQGSKLELPL WLAKGLFDNK RRILSVELP KIYQEGWRTV FSADPNVVDL HKMGPHFYGF GSQLLHFDSP ENADISQSLL QTFIGRFRRI MDSSQNAYNE D TSALVARL ...String: MSEAYFRVES GALGPEENFL SLDDILMSHE KLPVRTETAM PRLGAFFLER SAGAETDNAV PQGSKLELPL WLAKGLFDNK RRILSVELP KIYQEGWRTV FSADPNVVDL HKMGPHFYGF GSQLLHFDSP ENADISQSLL QTFIGRFRRI MDSSQNAYNE D TSALVARL DEMERGLFQT GQKGLNDFQC WEKGQASQIT ASNLVQNYKK RKFTDMED UniProtKB: DNA replication complex GINS protein PSF3 |
-Macromolecule #4: DNA replication complex GINS protein SLD5
| Macromolecule | Name: DNA replication complex GINS protein SLD5 / type: protein_or_peptide / ID: 4 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 30.831814 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MHHHHHHGRM DYKDDDDKAD YKDDDDKADY KDDDDKGRPM TEEVDFLGQD SDGGSEEVVL TPAELIERLE QAWMNEKFAP ELLESKPEI VECVMEQLEH MEENLRRAKR EDLKVSIHQM EMERIRYVLS SYLRCRLMKI EKFFPHVLEK EKTRPEGEPS S LSPEELAF ...String: MHHHHHHGRM DYKDDDDKAD YKDDDDKADY KDDDDKGRPM TEEVDFLGQD SDGGSEEVVL TPAELIERLE QAWMNEKFAP ELLESKPEI VECVMEQLEH MEENLRRAKR EDLKVSIHQM EMERIRYVLS SYLRCRLMKI EKFFPHVLEK EKTRPEGEPS S LSPEELAF AREFMANTES YLKNVALKHM PPNLQKVDLF RAVPKPDLDS YVFLRVRERQ ENILVEPDTD EQRDYVIDLE KG SQHLIRY KTIAPLVASG AVQLI UniProtKB: DNA replication complex GINS protein SLD5 |
-Macromolecule #5: Topoisomerase (DNA) II binding protein 1
| Macromolecule | Name: Topoisomerase (DNA) II binding protein 1 / type: protein_or_peptide / ID: 5 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 52.397535 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MGSHHHHHHG SLEVLFQGPH MASNSLNSKL EPTLENLENL DVSAFQAPED LLDGCRIYLC GFSGRKLDKL RRLINSGGGV RFNQLNEDV THVIVGDYDD ELKQFWNKSA HRPHVVGAKW LLECFSKGYM LSEEPYIHAN YQPVEIPVSH QPESKAALLK K KNSSFSKK ...String: MGSHHHHHHG SLEVLFQGPH MASNSLNSKL EPTLENLENL DVSAFQAPED LLDGCRIYLC GFSGRKLDKL RRLINSGGGV RFNQLNEDV THVIVGDYDD ELKQFWNKSA HRPHVVGAKW LLECFSKGYM LSEEPYIHAN YQPVEIPVSH QPESKAALLK K KNSSFSKK DFAPSEKHEQ ADEDLLSQYE NGSSTVVEAK TSEARPFNDS THAEPLNDST HISLQEENQS SVSHCVPDVS TI TEEGLFS QKSFLVLGFS NENESNIANI IKENAGKIMS LLSRTVADYA VVPLLGCEVE ATVGEVVTNT WLVTCIDYQT LFD PKSNPL FTPVPVMTGM TPLEDCVISF SQCAGAEKES LTFLANLLGA SVQEYFVRKS NAKKGMFAST HLILKERGGS KYEA AKKWN LPAVTIAWLL ETARTGKRAD ESHFLIENST KEERSLETEI TNGINLNSDT AEHPRRAWSH PQFEK UniProtKB: DNA topoisomerase 2-binding protein 1 |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 7.5 |
|---|---|
| Vitrification | Cryogen name: ETHANE-PROPANE |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: FEI FALCON IV (4k x 4k) / Average electron dose: 39.69290657 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Nominal defocus max: 2.2 µm / Nominal defocus min: 1.2 µm |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
- Image processing
Image processing
| Startup model | Type of model: NONE |
|---|---|
| Final reconstruction | Resolution.type: BY AUTHOR / Resolution: 4.1 Å / Resolution method: FSC 0.143 CUT-OFF / Number images used: 154278 |
| Initial angle assignment | Type: PROJECTION MATCHING |
| Final angle assignment | Type: MAXIMUM LIKELIHOOD |
 Movie
Movie Controller
Controller






 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)