+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM reconstruction of the native 24-mer E2o core of the 2-oxoglutarate dehydrogenase complex of C. thermophilum at 3.35 A resolution | |||||||||
 マップデータ マップデータ | Cryo-EM reconstruction of the native 24-mer E2o core of the 2-oxoglutarate dehydrogenase complex of C. thermophilum. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Dihydrolipoyl Succinyltransferase / E2 / Oxoglutarate / a-Ketoglutarate / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報L-lysine catabolic process to acetyl-CoA via saccharopine / dihydrolipoyllysine-residue succinyltransferase / dihydrolipoyllysine-residue succinyltransferase activity / oxoglutarate dehydrogenase complex / tricarboxylic acid cycle / mitochondrion / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Thermochaetoides thermophila DSM 1495 (菌類) Thermochaetoides thermophila DSM 1495 (菌類) | |||||||||
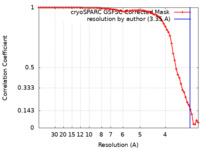
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.35 Å | |||||||||
 データ登録者 データ登録者 | Skalidis I / Tueting C / Kyrilis FL / Hamdi F / Kastritis PL | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2023 ジャーナル: Commun Biol / 年: 2023タイトル: Structural analysis of an endogenous 4-megadalton succinyl-CoA-generating metabolon. 著者: Ioannis Skalidis / Fotis L Kyrilis / Christian Tüting / Farzad Hamdi / Toni K Träger / Jaydeep Belapure / Gerd Hause / Marta Fratini / Francis J O'Reilly / Ingo Heilmann / Juri Rappsilber / ...著者: Ioannis Skalidis / Fotis L Kyrilis / Christian Tüting / Farzad Hamdi / Toni K Träger / Jaydeep Belapure / Gerd Hause / Marta Fratini / Francis J O'Reilly / Ingo Heilmann / Juri Rappsilber / Panagiotis L Kastritis /     要旨: The oxoglutarate dehydrogenase complex (OGDHc) participates in the tricarboxylic acid cycle and, in a multi-step reaction, decarboxylates α-ketoglutarate, transfers succinyl to CoA, and reduces NAD+. ...The oxoglutarate dehydrogenase complex (OGDHc) participates in the tricarboxylic acid cycle and, in a multi-step reaction, decarboxylates α-ketoglutarate, transfers succinyl to CoA, and reduces NAD+. Due to its pivotal role in metabolism, OGDHc enzymatic components have been studied in isolation; however, their interactions within the endogenous OGDHc remain elusive. Here, we discern the organization of a thermophilic, eukaryotic, native OGDHc in its active state. By combining biochemical, biophysical, and bioinformatic methods, we resolve its composition, 3D architecture, and molecular function at 3.35 Å resolution. We further report the high-resolution cryo-EM structure of the OGDHc core (E2o), which displays various structural adaptations. These include hydrogen bonding patterns confining interactions of OGDHc participating enzymes (E1o-E2o-E3), electrostatic tunneling that drives inter-subunit communication, and the presence of a flexible subunit (E3BPo), connecting E2o and E3. This multi-scale analysis of a succinyl-CoA-producing native cell extract provides a blueprint for structure-function studies of complex mixtures of medical and biotechnological value. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16900.map.gz emd_16900.map.gz | 32.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16900-v30.xml emd-16900-v30.xml emd-16900.xml emd-16900.xml | 21.9 KB 21.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16900_fsc.xml emd_16900_fsc.xml | 7.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16900.png emd_16900.png | 84.2 KB | ||
| マスクデータ |  emd_16900_msk_1.map emd_16900_msk_1.map | 34.3 MB |  マスクマップ マスクマップ | |
| その他 |  emd_16900_additional_1.map.gz emd_16900_additional_1.map.gz emd_16900_half_map_1.map.gz emd_16900_half_map_1.map.gz emd_16900_half_map_2.map.gz emd_16900_half_map_2.map.gz | 2.5 MB 31.8 MB 31.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16900 http://ftp.pdbj.org/pub/emdb/structures/EMD-16900 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16900 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16900 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16900_validation.pdf.gz emd_16900_validation.pdf.gz | 823.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16900_full_validation.pdf.gz emd_16900_full_validation.pdf.gz | 823.4 KB | 表示 | |
| XML形式データ |  emd_16900_validation.xml.gz emd_16900_validation.xml.gz | 14.5 KB | 表示 | |
| CIF形式データ |  emd_16900_validation.cif.gz emd_16900_validation.cif.gz | 18.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16900 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16900 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16900 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16900 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8oiuMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16900.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16900.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of the native 24-mer E2o core of the 2-oxoglutarate dehydrogenase complex of C. thermophilum. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.5678 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16900_msk_1.map emd_16900_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Cryo-EM reconstruction of the native asymmetric complex of...
| ファイル | emd_16900_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of the native asymmetric complex of the 2-oxoglutarate dehydrogenase complex of C. thermophilum. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM reconstruction of the native 24-mer E2o core...
| ファイル | emd_16900_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of the native 24-mer E2o core of the 2-oxoglutarate dehydrogenase complex of C. thermophilum, Half-Map A. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Cryo-EM reconstruction of the native 24-mer E2o core...
| ファイル | emd_16900_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of the native 24-mer E2o core of the 2-oxoglutarate dehydrogenase complex of C. thermophilum, Half-Map B. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Native 24-mer core of Oxoglutarate Dehydrogenase Complex
| 全体 | 名称: Native 24-mer core of Oxoglutarate Dehydrogenase Complex |
|---|---|
| 要素 |
|
-超分子 #1: Native 24-mer core of Oxoglutarate Dehydrogenase Complex
| 超分子 | 名称: Native 24-mer core of Oxoglutarate Dehydrogenase Complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila DSM 1495 (菌類) Thermochaetoides thermophila DSM 1495 (菌類) |
| 分子量 | 理論値: 3 MDa |
-分子 #1: Dihydrolipoyllysine-residue succinyltransferase
| 分子 | 名称: Dihydrolipoyllysine-residue succinyltransferase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: dihydrolipoyllysine-residue succinyltransferase |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila DSM 1495 (菌類) Thermochaetoides thermophila DSM 1495 (菌類) |
| 分子量 | 理論値: 45.987004 KDa |
| 配列 | 文字列: MLYRGLRMAA RAAPKFAVLN THAALRQLPL QFQHVRTYAD KIIKVPQMAE SITEGTLKQW NKAVGDYVEA DEEIATIETD KIDVAVNAP EAGVIKEFFV NEEDTVLVGQ DLVRLEVGGE KPAEAAKEQP KAAAPEPKVE EKKVPEAPAP EPSKTAAPAP A PPKQEAPA ...文字列: MLYRGLRMAA RAAPKFAVLN THAALRQLPL QFQHVRTYAD KIIKVPQMAE SITEGTLKQW NKAVGDYVEA DEEIATIETD KIDVAVNAP EAGVIKEFFV NEEDTVLVGQ DLVRLEVGGE KPAEAAKEQP KAAAPEPKVE EKKVPEAPAP EPSKTAAPAP A PPKQEAPA SPKPASKPAE TPAVTLGNRE ERRVKMNRMR LRIAERLKQS QNTAASLTTF NEVDMSALIE FRNKYKDEVL KK TGVKLGF MSAFSRAVVL AIRDLPVVNA SIEGPNGGDT IVYRDYVDIS VAVATEKGLV TPVVRNAETM DLITIEKTIA ELG KKARDG KLTIEDMAGG TFTISNGGVF GSLMGTPIIN LPQSAVLGLH AIKERPVAVN GKVEIRPMMY LALTYDHRLL DGRE AVQFL VKVKEYIEDP RKMLL |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 濃度: 200.0 mM / 構成要素 - 式: NH4CH2COOH / 構成要素 - 名称: Ammonium acetate |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 25 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: For plunging, blot force 0 and blotting time of 4 sec were applied.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 温度 | 最低: 77.15 K / 最高: 103.15 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 実像数: 21381 / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 92000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Initial fitting was performed in ChimeraX v1.2, then real-space refined in PHENIX v1.19 |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-8oiu: |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X