+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Deinococcus radidurans HPI S-layer | |||||||||
 マップデータ マップデータ | PostProcessed map with B-factor sharpening. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | S-layer / cell wall organization / Prokaryotic membrane lipoprotein lipid attachment site profile. / extracellular region / Hexagonally packed intermediate-layer surface protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Deinococcus radiodurans (放射線耐性) / Deinococcus radiodurans (放射線耐性) /  Deinococcus radiodurans (strain ATCC 13939 / DSM 20539 / JCM 16871 / LMG 4051 / NBRC 15346 / NCIMB 9279 / R1 / VKM B-1422) (放射線耐性) Deinococcus radiodurans (strain ATCC 13939 / DSM 20539 / JCM 16871 / LMG 4051 / NBRC 15346 / NCIMB 9279 / R1 / VKM B-1422) (放射線耐性) | |||||||||
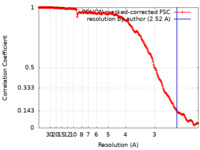
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.52 Å | |||||||||
 データ登録者 データ登録者 | von Kuegelgen A / Yamashita K / Murshudov G / Bharat T | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Interdigitated immunoglobulin arrays form the hyperstable surface layer of the extremophilic bacterium . 著者: Andriko von Kügelgen / Sofie van Dorst / Keitaro Yamashita / Danielle L Sexton / Elitza I Tocheva / Garib Murshudov / Vikram Alva / Tanmay A M Bharat /    要旨: is an atypical diderm bacterium with a remarkable ability to tolerate various environmental stresses, due in part to its complex cell envelope encapsulated within a hyperstable surface layer (S- ... is an atypical diderm bacterium with a remarkable ability to tolerate various environmental stresses, due in part to its complex cell envelope encapsulated within a hyperstable surface layer (S-layer). Despite decades of research on this cell envelope, atomic structural details of the S-layer have remained obscure. In this study, we report the electron cryomicroscopy structure of the S-layer, showing how it is formed by the Hexagonally Packed Intermediate-layer (HPI) protein arranged in a planar hexagonal lattice. The HPI protein forms an array of immunoglobulin-like folds within the S-layer, with each monomer extending into the adjacent hexamer, resulting in a highly interconnected, stable, sheet-like arrangement. Using electron cryotomography and subtomogram averaging from focused ion beam-milled cells, we have obtained a structure of the cellular S-layer, showing how this HPI S-layer coats native membranes on the surface of cells. Our S-layer structure from the diderm bacterium shows similarities to immunoglobulin-like domain-containing S-layers from monoderm bacteria and archaea, highlighting common features in cell surface organization across different domains of life, with connotations on the evolution of immunoglobulin-based molecular recognition systems in eukaryotes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16694.map.gz emd_16694.map.gz | 227.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16694-v30.xml emd-16694-v30.xml emd-16694.xml emd-16694.xml | 24 KB 24 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16694_fsc.xml emd_16694_fsc.xml | 17.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16694.png emd_16694.png | 253.4 KB | ||
| マスクデータ |  emd_16694_msk_1.map emd_16694_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_16694_half_map_1.map.gz emd_16694_half_map_1.map.gz emd_16694_half_map_2.map.gz emd_16694_half_map_2.map.gz | 217.3 MB 217.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16694 http://ftp.pdbj.org/pub/emdb/structures/EMD-16694 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16694 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16694 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16694_validation.pdf.gz emd_16694_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16694_full_validation.pdf.gz emd_16694_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_16694_validation.xml.gz emd_16694_validation.xml.gz | 23.4 KB | 表示 | |
| CIF形式データ |  emd_16694_validation.cif.gz emd_16694_validation.cif.gz | 31.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16694 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16694 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16694 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16694 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ckaMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16694.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16694.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PostProcessed map with B-factor sharpening. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.092 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
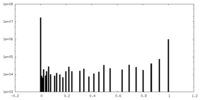
| ファイル |  emd_16694_msk_1.map emd_16694_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
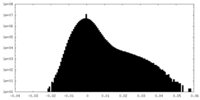
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_16694_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
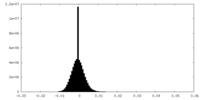
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_16694_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of Hexagonally Packed Intermediate-layer (HPI) protein
| 全体 | 名称: Structure of Hexagonally Packed Intermediate-layer (HPI) protein |
|---|---|
| 要素 |
|
-超分子 #1: Structure of Hexagonally Packed Intermediate-layer (HPI) protein
| 超分子 | 名称: Structure of Hexagonally Packed Intermediate-layer (HPI) protein タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Structure of Hexagonally Packed Intermediate-layer (HPI) protein |
|---|---|
| 由来(天然) | 生物種:  Deinococcus radiodurans (放射線耐性) / 細胞中の位置: extracellular Deinococcus radiodurans (放射線耐性) / 細胞中の位置: extracellular |
-分子 #1: Hexagonally packed intermediate-layer surface protein
| 分子 | 名称: Hexagonally packed intermediate-layer surface protein タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Deinococcus radiodurans (strain ATCC 13939 / DSM 20539 / JCM 16871 / LMG 4051 / NBRC 15346 / NCIMB 9279 / R1 / VKM B-1422) (放射線耐性) Deinococcus radiodurans (strain ATCC 13939 / DSM 20539 / JCM 16871 / LMG 4051 / NBRC 15346 / NCIMB 9279 / R1 / VKM B-1422) (放射線耐性) |
| 分子量 | 理論値: 99.424 KDa |
| 配列 | 文字列: MKKNIALMAL TGILTLASCG QNGTGTTPTA DACATANTCS VTVNISGVSS ADFDVTMDGK TTSMTLSNGQ KLPVAKTGTV TLTPKAKDG YTTPAAQSTT ISSTNLTPSV NFAYTTVPST GNGNGNGGTT PTQPFTLNIT SPTNGAAATT GTPIRVVFTS S VALSSATC ...文字列: MKKNIALMAL TGILTLASCG QNGTGTTPTA DACATANTCS VTVNISGVSS ADFDVTMDGK TTSMTLSNGQ KLPVAKTGTV TLTPKAKDG YTTPAAQSTT ISSTNLTPSV NFAYTTVPST GNGNGNGGTT PTQPFTLNIT SPTNGAAATT GTPIRVVFTS S VALSSATC KIGNSAAVNA QVSSTGGYCD VTPTTAGGGL ITVTGTANGQ TVSSTVTVDV KAPVVDNRYG TVTPAGDQEL TL TNEGIVK DADNGWRRLG QGVSTPSDPN GNVDIYVKGT VNFSVNAAAG SKVEVFLART TGSDVPTNDD VQAGDVLRSV AST SGTETF SLDSRRLAEF DGVRKWIVVR INGTQVTYQP VIADNKGPQQ PDPELNGVQN AYSNILNNYN NSGLTYVRGD VNVF TGNPS LQDREFGQAP LGSSFVQRRP SGFESIRYYL VPETAFGNKA LQESDEMLRA KAIKSVATVV SAPVLEPGTV KATSF SRVI GSGATSTVTP KAQDNVTYRV YAISRDQLGN ETASATYELV RFDNVGPTIT GSVIRDTSDL PFASQEPERC LSDIAT ITL GGITDNAGGV GLNPGQGLTF TLGGRQIQAG QFDTNQLADG EYTIGFNSLT DALGNPVVSA PTNAKVYIDN TDPTVNF NR AVMQGTFASG ERVSVESDAS DGGCGVYETR LFWDTDNGVV DDATTTPAIG HPVQFARQRV TDGAKADSLN AGWNALQL P NGAGAVYLRA LVVDRAGNAT ISTTPIVVNA KITNQARPLL GGFDAFKRNA SAQFMSNSNA ISGVNGTAVT PNTTANSAL DNILSLDSVG TLTTNAYLPR GATETAITEK IRNVGAYGRF DATQWNRIRD YQLNTDPTLR SAYVNAGNLA NQRGNNWRIR TPWVELGSS DTANTQQKFD FNSDLLNDFY FGRTFGNNDN VNLFSYDQFN GIVSGTAGAY SFYGETVQK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
詳細: 50 mM HEPES/NaOH pH=7.5, 150 mM NaCl, 5 mM MgCl2, 1 mM CaCl2 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER/RHODIUM / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR / 詳細: 15 mA | |||||||||||||||
| 凍結 | 凍結剤: NITROGEN / チャンバー内湿度: 100 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV 詳細: absorption for 60 sec and blotted for 5 sec with blot force -10. | |||||||||||||||
| 詳細 | In vitro isolate Hexagonally Packed Intermediate-layer (HPI) layer |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 特殊光学系 | 球面収差補正装置: not used / 色収差補正装置: not used / エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 1002 / 平均露光時間: 3.43 sec. / 平均電子線量: 53.245 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 5.0 µm / 最小 デフォーカス(補正後): 2.0 µm / 倍率(補正後): 81000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: AB INITIO MODEL / 温度因子: 19.48 / 当てはまり具合の基準: Best Fit |
|---|---|
| 得られたモデル |  PDB-8cka: |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X