+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human heparan sulfate N-deacetylase-N-sulfotransferase 1 in complex with calcium, 3'-phosphoadenosine-5'-phosphosulfate, and nanobody nAb7 (composite map and model) | |||||||||||||||||||||
 マップデータ マップデータ | Composite map after local refinements. Used for model building and refinement. | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | Deacetylase / Sulfotransferase / Heparan Sulfate / Carbohydrate / Glycosaminoglycan / Nanobody | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報[heparan sulfate]-glucosamine N-sulfotransferase / [heparan sulfate]-glucosamine N-sulfotransferase activity / heparan sulfate N-deacetylase activity / N-acetylglucosamine deacetylase activity / heparan sulfate proteoglycan biosynthetic process, enzymatic modification / heparan sulfate proteoglycan biosynthetic process, polysaccharide chain biosynthetic process / heparin biosynthetic process / embryonic neurocranium morphogenesis / embryonic viscerocranium morphogenesis / HS-GAG biosynthesis ...[heparan sulfate]-glucosamine N-sulfotransferase / [heparan sulfate]-glucosamine N-sulfotransferase activity / heparan sulfate N-deacetylase activity / N-acetylglucosamine deacetylase activity / heparan sulfate proteoglycan biosynthetic process, enzymatic modification / heparan sulfate proteoglycan biosynthetic process, polysaccharide chain biosynthetic process / heparin biosynthetic process / embryonic neurocranium morphogenesis / embryonic viscerocranium morphogenesis / HS-GAG biosynthesis / deacetylase activity / cardiac septum development / respiratory gaseous exchange by respiratory system / coronary vasculature development / positive regulation of smoothened signaling pathway / aorta development / 加水分解酵素; ペプチド以外のCN結合加水分解酵素; 鎖状アミドに作用 / midbrain development / fibroblast growth factor receptor signaling pathway / forebrain development / trans-Golgi network membrane / cell population proliferation / positive regulation of MAPK cascade / inflammatory response / Golgi membrane / Golgi apparatus 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.42 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Mycroft-West CJ / Wu L | |||||||||||||||||||||
| 資金援助 |  英国, 6件 英国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural and mechanistic characterization of bifunctional heparan sulfate N-deacetylase-N-sulfotransferase 1. 著者: Courtney J Mycroft-West / Sahar Abdelkarim / Helen M E Duyvesteyn / Neha S Gandhi / Mark A Skidmore / Raymond J Owens / Liang Wu /    要旨: Heparan sulfate (HS) polysaccharides are major constituents of the extracellular matrix, which are involved in myriad structural and signaling processes. Mature HS polysaccharides contain complex, ...Heparan sulfate (HS) polysaccharides are major constituents of the extracellular matrix, which are involved in myriad structural and signaling processes. Mature HS polysaccharides contain complex, non-templated patterns of sulfation and epimerization, which mediate interactions with diverse protein partners. Complex HS modifications form around initial clusters of glucosamine-N-sulfate (GlcNS) on nascent polysaccharide chains, but the mechanistic basis underpinning incorporation of GlcNS itself into HS remains unclear. Here, we determine cryo-electron microscopy structures of human N-deacetylase-N-sulfotransferase (NDST)1, the bifunctional enzyme primarily responsible for initial GlcNS modification of HS. Our structures reveal the architecture of both NDST1 deacetylase and sulfotransferase catalytic domains, alongside a non-catalytic N-terminal domain. The two catalytic domains of NDST1 adopt a distinct back-to-back topology that limits direct cooperativity. Binding analyses, aided by activity-modulating nanobodies, suggest that anchoring of the substrate at the sulfotransferase domain initiates the NDST1 catalytic cycle, providing a plausible mechanism for cooperativity despite spatial domain separation. Our data shed light on key determinants of NDST1 activity, and describe tools to probe NDST1 function in vitro and in vivo. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16565.map.gz emd_16565.map.gz | 300 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16565-v30.xml emd-16565-v30.xml emd-16565.xml emd-16565.xml | 14.4 KB 14.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16565.png emd_16565.png | 62.3 KB | ||
| Filedesc metadata |  emd-16565.cif.gz emd-16565.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16565 http://ftp.pdbj.org/pub/emdb/structures/EMD-16565 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16565 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16565 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16565_validation.pdf.gz emd_16565_validation.pdf.gz | 639.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16565_full_validation.pdf.gz emd_16565_full_validation.pdf.gz | 638.8 KB | 表示 | |
| XML形式データ |  emd_16565_validation.xml.gz emd_16565_validation.xml.gz | 7.3 KB | 表示 | |
| CIF形式データ |  emd_16565_validation.cif.gz emd_16565_validation.cif.gz | 8.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16565 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16565 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16565 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16565 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8cd0MC  8ccyC  8chsC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16565.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16565.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite map after local refinements. Used for model building and refinement. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.73 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex of heparan sulfate N-deacetylase-N-sulfotransferase 1 (wi...
| 全体 | 名称: Complex of heparan sulfate N-deacetylase-N-sulfotransferase 1 (with ligands PAP and Mg2+) and nanobody nAb7 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of heparan sulfate N-deacetylase-N-sulfotransferase 1 (wi...
| 超分子 | 名称: Complex of heparan sulfate N-deacetylase-N-sulfotransferase 1 (with ligands PAP and Mg2+) and nanobody nAb7 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 100 KDa |
-分子 #1: Bifunctional heparan sulfate N-deacetylase/N-sulfotransferase 1
| 分子 | 名称: Bifunctional heparan sulfate N-deacetylase/N-sulfotransferase 1 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; ペプチド以外のCN結合加水分解酵素; 鎖状アミドに作用 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 100.997617 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MPALACLRRL CRHVSPQAVL FLLFIFCLFS VFISAYYLYG WKRGLEPSAD APEPDCGDPP PVAPSRLLPL KPVQAATPSR TDPLVLVFV ESLYSQLGQE VVAILESSRF KYRTEIAPGK GDMPTLTDKG RGRFALIIYE NILKYVNLDA WNRELLDKYC V AYGVGIIG ...文字列: MPALACLRRL CRHVSPQAVL FLLFIFCLFS VFISAYYLYG WKRGLEPSAD APEPDCGDPP PVAPSRLLPL KPVQAATPSR TDPLVLVFV ESLYSQLGQE VVAILESSRF KYRTEIAPGK GDMPTLTDKG RGRFALIIYE NILKYVNLDA WNRELLDKYC V AYGVGIIG FFKANENSLL SAQLKGFPLF LHSNLGLKDC SINPKSPLLY VTRPSEVEKG VLPGEDWTVF QSNHSTYEPV LL AKTRSSE SIPHLGADAG LHAALHATVV QDLGLHDGIQ RVLFGNNLNF WLHKLVFVDA VAFLTGKRLS LPLDRYILVD IDD IFVGKE GTRMKVEDVK ALFDTQNELR AHIPNFTFNL GYSGKFFHTG TNAEDAGDDL LLSYVKEFWW FPHMWSHMQP HLFH NQSVL AEQMALNKKF AVEHGIPTDM GYAVAPHHSG VYPVHVQLYE AWKQVWSIRV TSTEEYPHLK PARYRRGFIH NGIMV LPRQ TCGLFTHTIF YNEYPGGSSE LDKIINGGEL FLTVLLNPIS IFMTHLSNYG NDRLGLYTFK HLVRFLHSWT NLRLQT LPP VQLAQKYFQI FSEEKDPLWQ DPCEDKRHKD IWSKEKTCDR FPKLLIIGPQ KTGTTALYLF LGMHPDLSSN YPSSETF EE IQFFNGHNYH KGIDWYMEFF PIPSNTTSDF YFEKSANYFD SEVAPRRAAA LLPKAKVLTI LINPADRAYS WYQHQRAH D DPVALKYTFH EVITAGSDAS SKLRALQNRC LVPGWYATHI ERWLSAYHAN QILVLDGKLL RTEPAKVMDM VQKFLGVTN TIDYHKTLAF DPKKGFWCQL LEGGKTKCLG KSKGRKYPEM DLDSRAFLKD YYRDHNIELS KLLYKMGQTL PTWLREDLQN TR UniProtKB: Bifunctional heparan sulfate N-deacetylase/N-sulfotransferase 1 |
-分子 #2: Nanobody nAb7
| 分子 | 名称: Nanobody nAb7 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 17.656316 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLVESGGG SVQAGGSLRL SCAASGFNVD DYAIGWFRQS PGKEREGVSC IGGDGTTYYE NSVKGRFTVS SDKRDNTVYL QMNNLRPED TAIYFCAADR SKYCVGKYFS TPSQYDFWGR GTHVTVSSEP KTPKPQPAAG PGGQHHHHHH GAEQKLISEE D LS |
-分子 #3: ADENOSINE-3'-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-3'-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: A3P |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information | 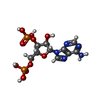 ChemComp-A3P: |
-分子 #4: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 4 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.42 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 614944 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー













