+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of NADH bound SLA dehydrogenase RlGabD from Rhizobium leguminosarum bv. trifolii SRD1565 | |||||||||||||||||||||
 マップデータ マップデータ | Postprocess map | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | Sulfolactaldehyde dehydrogenase NADH GabD / OXIDOREDUCTASE | |||||||||||||||||||||
| 生物種 |  Rhizobium leguminosarum bv. trifolii SRDI565 (根粒菌) Rhizobium leguminosarum bv. trifolii SRDI565 (根粒菌) | |||||||||||||||||||||
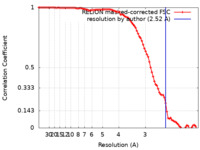
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.52 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Sharma M / Meek RW / Armstrong Z / Blaza JN / Alhifthi A / Li J / Goddard-Borger ED / Williams SJ / Davies GJ | |||||||||||||||||||||
| 資金援助 |  英国, 英国,  オーストラリア, 6件 オーストラリア, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Chem Sci / 年: 2023 ジャーナル: Chem Sci / 年: 2023タイトル: Molecular basis of sulfolactate synthesis by sulfolactaldehyde dehydrogenase from . 著者: Jinling Li / Mahima Sharma / Richard Meek / Amani Alhifthi / Zachary Armstrong / Niccolay Madiedo Soler / Mihwa Lee / Ethan D Goddard-Borger / James N Blaza / Gideon J Davies / Spencer J Williams /    要旨: Sulfolactate (SL) is a short-chain organosulfonate that is an important reservoir of sulfur in the biosphere. SL is produced by oxidation of sulfolactaldehyde (SLA), which in turn derives from ...Sulfolactate (SL) is a short-chain organosulfonate that is an important reservoir of sulfur in the biosphere. SL is produced by oxidation of sulfolactaldehyde (SLA), which in turn derives from sulfoglycolysis of the sulfosugar sulfoquinovose, or through oxidation of 2,3-dihydroxypropanesulfonate. Oxidation of SLA is catalyzed by SLA dehydrogenases belonging to the aldehyde dehydrogenase superfamily. We report that SLA dehydrogenase GabD from the sulfoglycolytic bacterium SRDI565 can use both NAD and NADP as cofactor to oxidize SLA, and indicatively operates through a rapid equilibrium ordered mechanism. We report the cryo-EM structure of GabD bound to NADH, revealing a tetrameric quaternary structure and supporting proposal of organosulfonate binding residues in the active site, and a catalytic mechanism. Sequence based homology searches identified SLA dehydrogenase homologs in a range of putative sulfoglycolytic gene clusters in bacteria predominantly from the phyla Actinobacteria, Firmicutes, and Proteobacteria. This work provides a structural and biochemical view of SLA dehydrogenases to complement our knowledge of SLA reductases, and provide detailed insights into a critical step in the organosulfur cycle. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16433.map.gz emd_16433.map.gz | 60 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16433-v30.xml emd-16433-v30.xml emd-16433.xml emd-16433.xml | 18.3 KB 18.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16433_fsc.xml emd_16433_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16433.png emd_16433.png | 202.7 KB | ||
| マスクデータ |  emd_16433_msk_1.map emd_16433_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16433.cif.gz emd-16433.cif.gz | 6.3 KB | ||
| その他 |  emd_16433_half_map_1.map.gz emd_16433_half_map_1.map.gz emd_16433_half_map_2.map.gz emd_16433_half_map_2.map.gz | 48.4 MB 48.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16433 http://ftp.pdbj.org/pub/emdb/structures/EMD-16433 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16433 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16433 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16433_validation.pdf.gz emd_16433_validation.pdf.gz | 1005.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16433_full_validation.pdf.gz emd_16433_full_validation.pdf.gz | 1005 KB | 表示 | |
| XML形式データ |  emd_16433_validation.xml.gz emd_16433_validation.xml.gz | 15.3 KB | 表示 | |
| CIF形式データ |  emd_16433_validation.cif.gz emd_16433_validation.cif.gz | 20.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16433 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16433 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16433 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16433 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16433.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16433.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocess map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0045 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16433_msk_1.map emd_16433_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: HalfMap2
| ファイル | emd_16433_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HalfMap2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: HalfMap1
| ファイル | emd_16433_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HalfMap1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tetrameric assembly of RlGabD
| 全体 | 名称: Tetrameric assembly of RlGabD |
|---|---|
| 要素 |
|
-超分子 #1: Tetrameric assembly of RlGabD
| 超分子 | 名称: Tetrameric assembly of RlGabD / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Rhizobium leguminosarum bv. trifolii SRDI565 (根粒菌) Rhizobium leguminosarum bv. trifolii SRDI565 (根粒菌) |
| 分子量 | 理論値: 210 KDa |
-分子 #1: Succinate semialdehyde dehydrogenase
| 分子 | 名称: Succinate semialdehyde dehydrogenase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rhizobium leguminosarum bv. trifolii SRDI565 (根粒菌) Rhizobium leguminosarum bv. trifolii SRDI565 (根粒菌) |
| 分子量 | 理論値: 52.697355 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTERRTLALK DPALLTDKAF VAGAWIGAPD GKSIAVTDPF DGALIANVPD LGQAVVAQAI DAAAEAQKLW ERRTAKERAQ VLKRWYDLI VENADDLALI LTTEQGKPLA EARGEVVSNA AYLEWFAEEA KRIDGDIIPG PNAGQRIMVL KQPVGVCAAI T PWNFPNGM ...文字列: MTERRTLALK DPALLTDKAF VAGAWIGAPD GKSIAVTDPF DGALIANVPD LGQAVVAQAI DAAAEAQKLW ERRTAKERAQ VLKRWYDLI VENADDLALI LTTEQGKPLA EARGEVVSNA AYLEWFAEEA KRIDGDIIPG PNAGQRIMVL KQPVGVCAAI T PWNFPNGM ITRKAGPALA AGCSMILKPA SQTPLSALAL AVLAERAGVP KGVFSVVTGE AKPIGLEFCH NPRIAKITFT GS TGVGRWL MKEAAGGIKR LSLELGGNAP FIVFDDADLD AAVEGAMISK FRNAGQTCVC ANRIYVQESV AAAFTERLLA KVS GLSLGR GTDAGVSQGP LIDEKAVAKM EEHIADAMAN GGTVLTGGKR SVLGGTFFEP TVVTGVTQTM KVAKEETFAP LAPI ISFKH ENDVIAMAND SEFGLASYFY AKDMARIWRV AEALEAGMVG VNTGMIANEM APFGGIKQSG TGREGSKYGI EGFLE LKYV ALGGMLEHHH HHH |
-分子 #2: 1,4-DIHYDRONICOTINAMIDE ADENINE DINUCLEOTIDE
| 分子 | 名称: 1,4-DIHYDRONICOTINAMIDE ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 2 / コピー数: 4 / 式: NAI |
|---|---|
| 分子量 | 理論値: 665.441 Da |
| Chemical component information |  ChemComp-NAI: |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 330 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 50mM Tris, 300mM NaCl, pH7.5 |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 30.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 240000 |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)