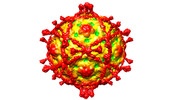
登録情報 データベース : EMDB / ID : EMD-15930タイトル Structure of Echovirus 11 complexed with DAF (CD55) calculated from symmetry expansion 複合体 : Complex of Echovirus 11 with DAFタンパク質・ペプチド : Complement decay-accelerating factorタンパク質・ペプチド : Genome polyproteinタンパク質・ペプチド : Genome polyproteinタンパク質・ペプチド : Genome polyproteinリガンド : SPHINGOSINE機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 / Homo sapiens (ヒト)手法 / / 解像度 : 3.93 Å Stuart DI / Ren J / Qin L / Zhou D 資金援助 Organization Grant number 国 Medical Research Council (MRC, United Kingdom)
ジャーナル : Viruses / 年 : 2022タイトル : Switching of Receptor Binding Poses between Closely Related Enteroviruses.著者 : Daming Zhou / Ling Qin / Helen M E Duyvesteyn / Yuguang Zhao / Tzou-Yien Lin / Elizabeth E Fry / Jingshan Ren / Kuan-Ying A Huang / David I Stuart / 要旨 : Echoviruses, for which there are currently no approved vaccines or drugs, are responsible for a range of human diseases, for example echovirus 11 (E11) is a major cause of serious neonatal morbidity ... Echoviruses, for which there are currently no approved vaccines or drugs, are responsible for a range of human diseases, for example echovirus 11 (E11) is a major cause of serious neonatal morbidity and mortality. Decay-accelerating factor (DAF, also known as CD55) is an attachment receptor for E11. Here, we report the structure of the complex of E11 and the full-length ectodomain of DAF (short consensus repeats, SCRs, 1-4) at 3.1 Å determined by cryo-electron microscopy (cryo-EM). SCRs 3 and 4 of DAF interact with E11 at the southern rim of the canyon via the VP2 EF and VP3 BC loops. We also observe an unexpected interaction between the N-linked glycan (residue 95 of DAF) and the VP2 BC loop of E11. DAF is a receptor for at least 20 enteroviruses and we classify its binding patterns from reported DAF/virus complexes into two distinct positions and orientations, named as E6 and E11 poses. Whilst 60 DAF molecules can attach to the virion in the E6 pose, no more than 30 can attach to E11 due to steric restrictions. Analysis of the distinct modes of interaction and structure and sequence-based phylogenies suggests that the two modes evolved independently, with the E6 mode likely found earlier. 履歴 登録 2022年10月5日 - ヘッダ(付随情報) 公開 2022年12月7日 - マップ公開 2022年12月7日 - 更新 2023年1月4日 - 現状 2023年1月4日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報
 Echovirus E11 (ウイルス) /
Echovirus E11 (ウイルス) /  Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 英国, 1件
英国, 1件  引用
引用 ジャーナル: Viruses / 年: 2022
ジャーナル: Viruses / 年: 2022

 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_15930.map.gz
emd_15930.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-15930-v30.xml
emd-15930-v30.xml emd-15930.xml
emd-15930.xml EMDBヘッダ
EMDBヘッダ emd_15930.png
emd_15930.png emd_15930_half_map_1.map.gz
emd_15930_half_map_1.map.gz emd_15930_half_map_2.map.gz
emd_15930_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-15930
http://ftp.pdbj.org/pub/emdb/structures/EMD-15930 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15930
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15930 emd_15930_validation.pdf.gz
emd_15930_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_15930_full_validation.pdf.gz
emd_15930_full_validation.pdf.gz emd_15930_validation.xml.gz
emd_15930_validation.xml.gz emd_15930_validation.cif.gz
emd_15930_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15930
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15930 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15930
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15930

 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_15930.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_15930.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素
 Echovirus E11 (ウイルス)
Echovirus E11 (ウイルス) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Echovirus E11 (ウイルス)
Echovirus E11 (ウイルス) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Echovirus E11 (ウイルス)
Echovirus E11 (ウイルス) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Echovirus E11 (ウイルス)
Echovirus E11 (ウイルス) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X


















