+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CRL4CSA-E2-Ub (state 2) | |||||||||
 マップデータ マップデータ | The main map. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA repair / transcription / ubiqutin / cryo-EM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of transcription-coupled nucleotide-excision repair / nucleotide-excision repair complex / negative regulation of granulocyte differentiation / response to auditory stimulus / cullin-RING-type E3 NEDD8 transferase / NEDD8 transferase activity / (E3-independent) E2 ubiquitin-conjugating enzyme / cullin-RING ubiquitin ligase complex / single strand break repair / cellular response to chemical stress ...regulation of transcription-coupled nucleotide-excision repair / nucleotide-excision repair complex / negative regulation of granulocyte differentiation / response to auditory stimulus / cullin-RING-type E3 NEDD8 transferase / NEDD8 transferase activity / (E3-independent) E2 ubiquitin-conjugating enzyme / cullin-RING ubiquitin ligase complex / single strand break repair / cellular response to chemical stress / Cul7-RING ubiquitin ligase complex / regulation of DNA damage checkpoint / ubiquitin-dependent protein catabolic process via the C-end degron rule pathway / positive regulation by virus of viral protein levels in host cell / Loss of Function of FBXW7 in Cancer and NOTCH1 Signaling / double-strand break repair via classical nonhomologous end joining / positive regulation of protein autoubiquitination / spindle assembly involved in female meiosis / RNA polymerase II transcription initiation surveillance / protein neddylation / regulation of nucleotide-excision repair / epigenetic programming in the zygotic pronuclei / UV-damage excision repair / NEDD8 ligase activity / VCB complex / negative regulation of response to oxidative stress / Cul5-RING ubiquitin ligase complex / biological process involved in interaction with symbiont / regulation of mitotic cell cycle phase transition / SCF ubiquitin ligase complex / Cul2-RING ubiquitin ligase complex / negative regulation of type I interferon production / WD40-repeat domain binding / ubiquitin-ubiquitin ligase activity / E2 ubiquitin-conjugating enzyme / SCF-dependent proteasomal ubiquitin-dependent protein catabolic process / Cul4A-RING E3 ubiquitin ligase complex / Cul4-RING E3 ubiquitin ligase complex / Cul3-RING ubiquitin ligase complex / Cul4B-RING E3 ubiquitin ligase complex / ubiquitin ligase complex scaffold activity / negative regulation of mitophagy / Prolactin receptor signaling / negative regulation of reproductive process / negative regulation of developmental process / ubiquitin conjugating enzyme activity / TGF-beta receptor signaling activates SMADs / cullin family protein binding / viral release from host cell / hemopoiesis / site of DNA damage / regulation of proteolysis / regulation of postsynapse assembly / somatic stem cell population maintenance / anatomical structure morphogenesis / protein monoubiquitination / response to X-ray / positive regulation of G1/S transition of mitotic cell cycle / ectopic germ cell programmed cell death / positive regulation of viral genome replication / ubiquitin-like ligase-substrate adaptor activity / protein K48-linked ubiquitination / response to UV / proteasomal protein catabolic process / protein autoubiquitination / Nuclear events stimulated by ALK signaling in cancer / Maturation of protein E / Maturation of protein E / transcription-coupled nucleotide-excision repair / ER Quality Control Compartment (ERQC) / Myoclonic epilepsy of Lafora / FLT3 signaling by CBL mutants / Prevention of phagosomal-lysosomal fusion / IRAK2 mediated activation of TAK1 complex / Alpha-protein kinase 1 signaling pathway / Glycogen synthesis / IRAK1 recruits IKK complex / IRAK1 recruits IKK complex upon TLR7/8 or 9 stimulation / Endosomal Sorting Complex Required For Transport (ESCRT) / Membrane binding and targetting of GAG proteins / positive regulation of gluconeogenesis / Negative regulation of FLT3 / Regulation of TBK1, IKKε (IKBKE)-mediated activation of IRF3, IRF7 / positive regulation of TORC1 signaling / PTK6 Regulates RTKs and Their Effectors AKT1 and DOK1 / Regulation of TBK1, IKKε-mediated activation of IRF3, IRF7 upon TLR3 ligation / Constitutive Signaling by NOTCH1 HD Domain Mutants / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / NOTCH2 Activation and Transmission of Signal to the Nucleus / TICAM1,TRAF6-dependent induction of TAK1 complex / regulation of cellular response to insulin stimulus / TICAM1-dependent activation of IRF3/IRF7 / APC/C:Cdc20 mediated degradation of Cyclin B / Downregulation of ERBB4 signaling / Regulation of FZD by ubiquitination / APC-Cdc20 mediated degradation of Nek2A / p75NTR recruits signalling complexes / intrinsic apoptotic signaling pathway / InlA-mediated entry of Listeria monocytogenes into host cells / TRAF6 mediated IRF7 activation in TLR7/8 or 9 signaling 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
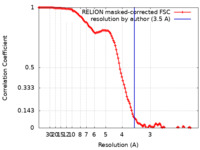
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Kokic G / Cramer P | |||||||||
| 資金援助 |  ドイツ, European Union, 2件 ドイツ, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: C(N)RL4CSA-E2-Ub (state 2) 著者: Kokic G / Cramer P | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15829.map.gz emd_15829.map.gz | 141.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15829-v30.xml emd-15829-v30.xml emd-15829.xml emd-15829.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15829_fsc.xml emd_15829_fsc.xml | 12.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15829.png emd_15829.png | 129.7 KB | ||
| Filedesc metadata |  emd-15829.cif.gz emd-15829.cif.gz | 7.7 KB | ||
| その他 |  emd_15829_half_map_1.map.gz emd_15829_half_map_1.map.gz emd_15829_half_map_2.map.gz emd_15829_half_map_2.map.gz | 139.3 MB 139.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15829 http://ftp.pdbj.org/pub/emdb/structures/EMD-15829 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15829 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15829 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15829_validation.pdf.gz emd_15829_validation.pdf.gz | 940.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15829_full_validation.pdf.gz emd_15829_full_validation.pdf.gz | 939.9 KB | 表示 | |
| XML形式データ |  emd_15829_validation.xml.gz emd_15829_validation.xml.gz | 19.6 KB | 表示 | |
| CIF形式データ |  emd_15829_validation.cif.gz emd_15829_validation.cif.gz | 25.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15829 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15829 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15829 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15829 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8b3iMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15829.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15829.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The main map. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map corresponding to the main map.
| ファイル | emd_15829_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map corresponding to the main map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map corresponding to the main map.
| ファイル | emd_15829_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map corresponding to the main map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : C(N)RL4CSA-E2-Ub complex.
| 全体 | 名称: C(N)RL4CSA-E2-Ub complex. |
|---|---|
| 要素 |
|
-超分子 #1: C(N)RL4CSA-E2-Ub complex.
| 超分子 | 名称: C(N)RL4CSA-E2-Ub complex. / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#7 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Ubiquitin-conjugating enzyme E2 D2
| 分子 | 名称: Ubiquitin-conjugating enzyme E2 D2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: E2 ubiquitin-conjugating enzyme |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 16.755227 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MALKRIHKEL NDLARDPPAQ CSAGPVGDDM FHWQATIMGP NDSPYQGGVF FLTIHFPTDY PFKPPKVAFT TRIYHPNINS NGSICLDIL RSQWSPALTI SKVLLSICSL LCDPNPDDPL VPEIARIYKT DREKYNRIAR EWTQKYAM UniProtKB: Ubiquitin-conjugating enzyme E2 D2 |
-分子 #2: NEDD8
| 分子 | 名称: NEDD8 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 8.573978 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLIKVKTLTG KEIEIDIEPT DKVERIKERV EEKEGIPPQQ QRLIYSGKQM NDEKTAADYK ILGGSVLHLV LALRGG UniProtKB: Ubiquitin-like protein NEDD8 |
-分子 #3: E3 ubiquitin-protein ligase RBX1
| 分子 | 名称: E3 ubiquitin-protein ligase RBX1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: RING-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.289977 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAAAMDVDTP SGTNSGAGKK RFEVKKWNAV ALWAWDIVVD NCAICRNHIM DLCIECQANQ ASATSEECTV AWGVCNHAFH FHCISRWLK TRQVCPLDNR EWEFQKYGH UniProtKB: E3 ubiquitin-protein ligase RBX1 |
-分子 #4: Ubiquitin
| 分子 | 名称: Ubiquitin / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 8.576831 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQIFVKTLTG KTITLEVEPS DTIENVKAKI QDKEGIPPDQ QRLIFAGKQL EDGRTLSDYN IQKESTLHLV LRLRGG UniProtKB: Polyubiquitin-C |
-分子 #5: DNA excision repair protein ERCC-8
| 分子 | 名称: DNA excision repair protein ERCC-8 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 44.10716 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MLGFLSARQT GLEDPLRLRR AESTRRVLGL ELNKDRDVER IHGGGINTLD IEPVEGRYML SGGSDGVIVL YDLENSSRQS YYTCKAVCS IGRDHPDVHR YSVETVQWYP HDTGMFTSSS FDKTLKVWDT NTLQTADVFN FEETVYSHHM SPVSTKHCLV A VGTRGPKV ...文字列: MLGFLSARQT GLEDPLRLRR AESTRRVLGL ELNKDRDVER IHGGGINTLD IEPVEGRYML SGGSDGVIVL YDLENSSRQS YYTCKAVCS IGRDHPDVHR YSVETVQWYP HDTGMFTSSS FDKTLKVWDT NTLQTADVFN FEETVYSHHM SPVSTKHCLV A VGTRGPKV QLCDLKSGSC SHILQGHRQE ILAVSWSPRY DYILATASAD SRVKLWDVRR ASGCLITLDQ HNGKKSQAVE SA NTAHNGK VNGLCFTSDG LHLLTVGTDN RMRLWNSSNG ENTLVNYGKV CNNSKKGLKF TVSCGCSSEF VFVPYGSTIA VYT VYSGEQ ITMLKGHYKT VDCCVFQSNF QELYSGSRDC NILAWVPSLY EPVPDDDETT TKSQLNPAFE DAWSSSDEEG UniProtKB: DNA excision repair protein ERCC-8 |
-分子 #6: DNA damage-binding protein 1
| 分子 | 名称: DNA damage-binding protein 1 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 127.097469 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSYNYVVTAQ KPTAVNGCVT GHFTSAEDLN LLIAKNTRLE IYVVTAEGLR PVKEVGMYGK IAVMELFRPK GESKDLLFIL TAKYNACIL EYKQSGESID IITRAHGNVQ DRIGRPSETG IIGIIDPECR MIGLRLYDGL FKVIPLDRDN KELKAFNIRL E ELHVIDVK ...文字列: MSYNYVVTAQ KPTAVNGCVT GHFTSAEDLN LLIAKNTRLE IYVVTAEGLR PVKEVGMYGK IAVMELFRPK GESKDLLFIL TAKYNACIL EYKQSGESID IITRAHGNVQ DRIGRPSETG IIGIIDPECR MIGLRLYDGL FKVIPLDRDN KELKAFNIRL E ELHVIDVK FLYGCQAPTI CFVYQDPQGR HVKTYEVSLR EKEFNKGPWK QENVEAEASM VIAVPEPFGG AIIIGQESIT YH NGDKYLA IAPPIIKQST IVCHNRVDPN GSRYLLGDME GRLFMLLLEK EEQMDGTVTL KDLRVELLGE TSIAECLTYL DNG VVFVGS RLGDSQLVKL NVDSNEQGSY VVAMETFTNL GPIVDMCVVD LERQGQGQLV TCSGAFKEGS LRIIRNGIGI HEHA SIDLP GIKGLWPLRS DPNRETDDTL VLSFVGQTRV LMLNGEEVEE TELMGFVDDQ QTFFCGNVAH QQLIQITSAS VRLVS QEPK ALVSEWKEPQ AKNISVASCN SSQVVVAVGR ALYYLQIHPQ ELRQISHTEM EHEVACLDIT PLGDSNGLSP LCAIGL WTD ISARILKLPS FELLHKEMLG GEIIPRSILM TTFESSHYLL CALGDGALFY FGLNIETGLL SDRKKVTLGT QPTVLRT FR SLSTTNVFAC SDRPTVIYSS NHKLVFSNVN LKEVNYMCPL NSDGYPDSLA LANNSTLTIG TIDEIQKLHI RTVPLYES P RKICYQEVSQ CFGVLSSRIE VQDTSGGTTA LRPSASTQAL SSSVSSSKLF SSSTAPHETS FGEEVEVHNL LIIDQHTFE VLHAHQFLQN EYALSLVSCK LGKDPNTYFI VGTAMVYPEE AEPKQGRIVV FQYSDGKLQT VAEKEVKGAV YSMVEFNGKL LASINSTVR LYEWTTEKEL RTECNHYNNI MALYLKTKGD FILVGDLMRS VLLLAYKPME GNFEEIARDF NPNWMSAVEI L DDDNFLGA ENAFNLFVCQ KDSAATTDEE RQHLQEVGLF HLGEFVNVFC HGSLVMQNLG ETSTPTQGSV LFGTVNGMIG LV TSLSESW YNLLLDMQNR LNKVIKSVGK IEHSFWRSFH TERKTEPATG FIDGDLIESF LDISRPKMQE VVANLQYDDG SGM KREATA DDLIKVVEEL TRIH UniProtKB: DNA damage-binding protein 1 |
-分子 #7: Cullin-4A
| 分子 | 名称: Cullin-4A / タイプ: protein_or_peptide / ID: 7 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 87.814297 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MADEAPRKGS FSALVGRTNG LTKPAALAAA PAKPGGAGGS KKLVIKNFRD RPRLPDNYTQ DTWRKLHEAV RAVQSSTSIR YNLEELYQA VENLCSHKVS PMLYKQLRQA CEDHVQAQIL PFREDSLDSV LFLKKINTCW QDHCRQMIMI RSIFLFLDRT Y VLQNSTLP ...文字列: MADEAPRKGS FSALVGRTNG LTKPAALAAA PAKPGGAGGS KKLVIKNFRD RPRLPDNYTQ DTWRKLHEAV RAVQSSTSIR YNLEELYQA VENLCSHKVS PMLYKQLRQA CEDHVQAQIL PFREDSLDSV LFLKKINTCW QDHCRQMIMI RSIFLFLDRT Y VLQNSTLP SIWDMGLELF RTHIISDKMV QSKTIDGILL LIERERSGEA VDRSLLRSLL GMLSDLQVYK DSFELKFLEE TN CLYAAEG QRLMQEREVP EYLNHVSKRL EEEGDRVITY LDHSTQKPLI ACVEKQLLGE HLTAILQKGL DHLLDENRVP DLA QMYQLF SRVRGGQQAL LQHWSEYIKT FGTAIVINPE KDKDMVQDLL DFKDKVDHVI EVCFQKNERF VNLMKESFET FINK RPNKP AELIAKHVDS KLRAGNKEAT DEELERTLDK IMILFRFIHG KDVFEAFYKK DLAKRLLVGK SASVDAEKSM LSKLK HECG AAFTSKLEGM FKDMELSKDI MVHFKQHMQN QSDSGPIDLT VNILTMGYWP TYTPMEVHLT PEMIKLQEVF KAFYLG KHS GRKLQWQTTL GHAVLKAEFK EGKKEFQVSL FQTLVLLMFN EGDGFSFEEI KMATGIEDSE LRRTLQSLAC GKARVLI KS PKGKEVEDGD KFIFNGEFKH KLFRIKINQI QMKETVEEQV STTERVFQDR QYQIDAAIVR IMKMRKTLGH NLLVSELY N QLKFPVKPGD LKKRIESLID RDYMERDKDN PNQYHYVA UniProtKB: Cullin-4A |
-分子 #8: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 8 / コピー数: 3 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 42.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.15 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)