[English] 日本語
 Yorodumi
Yorodumi- EMDB-15482: Structure of the Ancestral Scaffold Antigen-6 of Coronavirus Spik... -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Structure of the Ancestral Scaffold Antigen-6 of Coronavirus Spike protein | ||||||||||||||||||
 Map data Map data | |||||||||||||||||||
 Sample Sample |
| ||||||||||||||||||
 Keywords Keywords | Ancestor / Spike / Coronavirus / Scaffold / S-protein / Protein Engineering / BIOSYNTHETIC PROTEIN | ||||||||||||||||||
| Function / homology |  Function and homology information Function and homology informationvirion component / symbiont-mediated disruption of host tissue / Maturation of spike protein / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / viral translation / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion ...virion component / symbiont-mediated disruption of host tissue / Maturation of spike protein / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / viral translation / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / membrane fusion / entry receptor-mediated virion attachment to host cell / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / host cell surface receptor binding / symbiont-mediated suppression of host innate immune response / receptor ligand activity / endocytosis involved in viral entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / symbiont entry into host cell / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane Similarity search - Function | ||||||||||||||||||
| Biological species |  Severe acute respiratory syndrome coronavirus / Severe acute respiratory syndrome coronavirus /  Enterobacteria phage T4 (Bacteriophage T4) (virus) Enterobacteria phage T4 (Bacteriophage T4) (virus) | ||||||||||||||||||
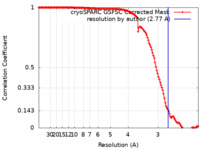
| Method | single particle reconstruction / cryo EM / Resolution: 2.77 Å | ||||||||||||||||||
 Authors Authors | Hueting D / Schriever K / Wallden K / Andrell J / Syren PO | ||||||||||||||||||
| Funding support |  Sweden, European Union, 5 items Sweden, European Union, 5 items
| ||||||||||||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2023 Journal: Nat Commun / Year: 2023Title: Design, structure and plasma binding of ancestral β-CoV scaffold antigens. Authors: David Hueting / Karen Schriever / Rui Sun / Stelios Vlachiotis / Fanglei Zuo / Likun Du / Helena Persson / Camilla Hofström / Mats Ohlin / Karin Walldén / Marcus Buggert / Lennart ...Authors: David Hueting / Karen Schriever / Rui Sun / Stelios Vlachiotis / Fanglei Zuo / Likun Du / Helena Persson / Camilla Hofström / Mats Ohlin / Karin Walldén / Marcus Buggert / Lennart Hammarström / Harold Marcotte / Qiang Pan-Hammarström / Juni Andréll / Per-Olof Syrén /  Abstract: We report the application of ancestral sequence reconstruction on coronavirus spike protein, resulting in stable and highly soluble ancestral scaffold antigens (AnSAs). The AnSAs interact with plasma ...We report the application of ancestral sequence reconstruction on coronavirus spike protein, resulting in stable and highly soluble ancestral scaffold antigens (AnSAs). The AnSAs interact with plasma of patients recovered from COVID-19 but do not bind to the human angiotensin-converting enzyme 2 (ACE2) receptor. Cryo-EM analysis of the AnSAs yield high resolution structures (2.6-2.8 Å) indicating a closed pre-fusion conformation in which all three receptor-binding domains (RBDs) are facing downwards. The structures reveal an intricate hydrogen-bonding network mediated by well-resolved loops, both within and across monomers, tethering the N-terminal domain and RBD together. We show that AnSA-5 can induce and boost a broad-spectrum immune response against the wild-type RBD as well as circulating variants of concern in an immune organoid model derived from tonsils. Finally, we highlight how AnSAs are potent scaffolds by replacing the ancestral RBD with the wild-type sequence, which restores ACE2 binding and increases the interaction with convalescent plasma. #1:  Journal: Res Sq / Year: 2023 Journal: Res Sq / Year: 2023Title: Design, structure and plasma binding of ancestral beta-CoV scaffold antigens Authors: Hueting D / Schriever K / Zuo F / Du L / Persson H / Hofstrom C / Ohlin M / Wallden K / Hammarstrom L / Marcotte H / Pan-Hammarstrom Q / Andrell J / Syren PO | ||||||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_15482.map.gz emd_15482.map.gz | 204.1 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-15482-v30.xml emd-15482-v30.xml emd-15482.xml emd-15482.xml | 20.8 KB 20.8 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_15482_fsc.xml emd_15482_fsc.xml | 12.6 KB | Display |  FSC data file FSC data file |
| Images |  emd_15482.png emd_15482.png | 27 KB | ||
| Masks |  emd_15482_msk_1.map emd_15482_msk_1.map | 216 MB |  Mask map Mask map | |
| Filedesc metadata |  emd-15482.cif.gz emd-15482.cif.gz | 7.3 KB | ||
| Others |  emd_15482_half_map_1.map.gz emd_15482_half_map_1.map.gz emd_15482_half_map_2.map.gz emd_15482_half_map_2.map.gz | 200.2 MB 200.2 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15482 http://ftp.pdbj.org/pub/emdb/structures/EMD-15482 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15482 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15482 | HTTPS FTP |
-Related structure data
| Related structure data |  8ajlMC  8ajaC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_15482.map.gz / Format: CCP4 / Size: 216 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_15482.map.gz / Format: CCP4 / Size: 216 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.11 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Mask #1
| File |  emd_15482_msk_1.map emd_15482_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #2
| File | emd_15482_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #1
| File | emd_15482_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : Ancestral S-protein of coronaviruses related to SARS-CoV-2
| Entire | Name: Ancestral S-protein of coronaviruses related to SARS-CoV-2 |
|---|---|
| Components |
|
-Supramolecule #1: Ancestral S-protein of coronaviruses related to SARS-CoV-2
| Supramolecule | Name: Ancestral S-protein of coronaviruses related to SARS-CoV-2 type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1 Details: Ancestral coronavirus generated by Ancestral sequence reconstruction expressed in human cells. |
|---|---|
| Source (natural) | Organism:  Severe acute respiratory syndrome coronavirus Severe acute respiratory syndrome coronavirus |
-Macromolecule #1: Spike glycoprotein,Fibritin
| Macromolecule | Name: Spike glycoprotein,Fibritin / type: protein_or_peptide / ID: 1 Details: AnSA-6 full sequence 1-19 WT SARS-CoV-2 S protein signaling peptide 672-679 No density. 1139 onwards, No density. Expression tags.,AnSA-6 full sequence 1-19 WT SARS-CoV-2 S protein signaling ...Details: AnSA-6 full sequence 1-19 WT SARS-CoV-2 S protein signaling peptide 672-679 No density. 1139 onwards, No density. Expression tags.,AnSA-6 full sequence 1-19 WT SARS-CoV-2 S protein signaling peptide 672-679 No density. 1139 onwards, No density. Expression tags. Number of copies: 3 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Enterobacteria phage T4 (Bacteriophage T4) (virus) Enterobacteria phage T4 (Bacteriophage T4) (virus) |
| Molecular weight | Theoretical: 138.993469 KDa |
| Recombinant expression | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Sequence | String: TCGTLSNKSP PNMTQFSSSR RGVYYPDDIF RSDVLHLTQD YFLPFNSNVT RYLSLNSDSD RIVRFDNPII PFGDGVYFAA TEKSNVIRG WIFGSTLDNT SQSAIIMNNS THIVIRVCNF QLCDDPMFAV SRPTGQHYKT WIYTNARNCT YEYVSKSFQL D VSEKPGNF ...String: TCGTLSNKSP PNMTQFSSSR RGVYYPDDIF RSDVLHLTQD YFLPFNSNVT RYLSLNSDSD RIVRFDNPII PFGDGVYFAA TEKSNVIRG WIFGSTLDNT SQSAIIMNNS THIVIRVCNF QLCDDPMFAV SRPTGQHYKT WIYTNARNCT YEYVSKSFQL D VSEKPGNF KHLREFVFKN VDGFLHVYSG YEPIDVARGL PSGFSVLKPI FKLPLGINIT NFRVIMTMFS PTTSNWGAEA AA YFVGYLK PTTFMLKFDE NGTITDAVDC SQDPLSELKC TVKSFNVEKG IYQTSNFRVS PTKEVVRFPN ITNLCPFGEV FNA TTFPSV YAWERTRISD CVADYSVLYN STSFSTFKCY GVSPTKLNDL CFSSVYADSF VVKGDDVRQI APGQTGVIAD YNYK LPDDF TGCVIAWNTA NLDATSTGNY NYYYRSLRHG KLKPFERDIS NVPFSPEGKP CTPPAFNCYR PLNTYGFNPT VGIGY QPYR VVVLSFELLN APATVCGPKL STELVKNQCV NFNFNGLTGT GVLTDSSKRF QPFQQFGRDV SDFTDSVRDP KTLEIL DIS PCSFGGVSVI TPGTNTSSEV AVLYQDVNCT DVPTAIHADQ LTPAWRVYST GVNVFQTQAG CLIGAEHVNA SYECDIP IG AGICASYHTA STLRSTGQKS IVAYTMSLGA ENSIAYSNNT IAIPTNFSIS VTTEVMPVSM AKTSVDCTMY ICGDSTEC S NLLLQYGSFC TQLNRALSGI AVEQDKNTRE VFAQVKQMYK TPAIKDFGGF NFSQILPDPS KPTKRSFIED LLFNKVTLA DAGFMKQYGE CLGDISARDL ICAQKFNGLT VLPPLLTDEM IAAYTAALVS GTATAGWTFG AGAALQIPFA MQMAYRFNGI GVTQNVLYE NQKQIANQFN KAISQIQESL TTTSTALGKL QDVVNQNAQA LNTLVKQLSS NFGAISSVLN DILSRLDKVE A EVQIDRLI TGRLQSLQTY VTQQLIRAAE IRASANLAAT KMSECVLGQS KRVDFCGKGY HLMSFPQAAP HGVVFLHVTY VP SQERNFT TAPAICHEGK AYFPREGVFV SNGTSWFITQ RNFYSPQIIT TDNTFVSGNC DVVIGIINNT VYDPLQPELD SFK EELDKY FKNHTSPDVD LGDISGINAS VVNIQKEIDR LNEVAKNLNE SLIDLQELGK YEQGSGYIPE APRDGQAYVR KDGE WVLLS TFLGTSLEVL FQGPGHHHHH HHHSAWSHPQ FEKGGGSGGG GSGGSAWSHP QFEK UniProtKB: Spike glycoprotein, Fibritin |
-Macromolecule #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| Macromolecule | Name: 2-acetamido-2-deoxy-beta-D-glucopyranose / type: ligand / ID: 3 / Number of copies: 45 / Formula: NAG |
|---|---|
| Molecular weight | Theoretical: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 1.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 7.5 Component:
| |||||||||
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 277 K / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | TFS KRIOS |
|---|---|
| Specialist optics | Energy filter - Name: GIF Bioquantum / Energy filter - Slit width: 20 eV |
| Image recording | Film or detector model: GATAN K3 BIOQUANTUM (6k x 4k) / Average electron dose: 1.11 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 50.0 µm / Illumination mode: OTHER / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.0 µm / Nominal defocus min: 0.6 µm / Nominal magnification: 105000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)