+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the canine distemper virus tetrameric attachment glycoprotein | |||||||||
 マップデータ マップデータ | final sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell membrane / host cell surface receptor binding / symbiont entry into host cell / viral envelope / virion attachment to host cell / virion membrane / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Canine morbillivirus (ウイルス) Canine morbillivirus (ウイルス) | |||||||||
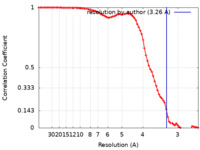
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.26 Å | |||||||||
 データ登録者 データ登録者 | Kalbermatter D / Jeckelmann J-M / Wyss M / Plattet P / Fotiadis D | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Structure and supramolecular organization of the canine distemper virus attachment glycoprotein. 著者: David Kalbermatter / Jean-Marc Jeckelmann / Marianne Wyss / Neeta Shrestha / Dimanthi Pliatsika / Rainer Riedl / Thomas Lemmin / Philippe Plattet / Dimitrios Fotiadis /  要旨: Canine distemper virus (CDV) is an enveloped RNA morbillivirus that triggers respiratory, enteric, and high incidence of severe neurological disorders. CDV induces devastating outbreaks in wild and ...Canine distemper virus (CDV) is an enveloped RNA morbillivirus that triggers respiratory, enteric, and high incidence of severe neurological disorders. CDV induces devastating outbreaks in wild and endangered animals as well as in domestic dogs in countries associated with suboptimal vaccination programs. The receptor-binding tetrameric attachment (H)-protein is part of the morbilliviral cell entry machinery. Here, we present the cryo-electron microscopy (cryo-EM) structure and supramolecular organization of the tetrameric CDV H-protein ectodomain. The structure reveals that the morbilliviral H-protein is composed of three main domains: stalk, neck, and heads. The most unexpected feature was the inherent asymmetric architecture of the CDV H-tetramer being shaped by the neck, which folds into an almost 90° bent conformation with respect to the stalk. Consequently, two non-contacting receptor-binding H-head dimers, which are also tilted toward each other, are located on one side of an intertwined four helical bundle stalk domain. Positioning of the four protomer polypeptide chains within the neck domain is guided by a glycine residue (G158), which forms a hinge point exclusively in two protomer polypeptide chains. Molecular dynamics simulations validated the stability of the asymmetric structure under near physiological conditions and molecular docking showed that two receptor-binding sites are fully accessible. Thus, this spatial organization of the CDV H-tetramer would allow for concomitant protein interactions with the stalk and head domains without steric clashes. In summary, the structure of the CDV H-protein ectodomain provides new insights into the morbilliviral cell entry system and offers a blueprint for next-generation structure-based antiviral drug discovery. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
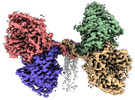
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14842.map.gz emd_14842.map.gz | 40.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14842-v30.xml emd-14842-v30.xml emd-14842.xml emd-14842.xml | 20.7 KB 20.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14842_fsc.xml emd_14842_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14842.png emd_14842.png | 185.6 KB | ||
| マスクデータ |  emd_14842_msk_1.map emd_14842_msk_1.map | 42.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_14842_additional_1.map.gz emd_14842_additional_1.map.gz emd_14842_half_map_1.map.gz emd_14842_half_map_1.map.gz emd_14842_half_map_2.map.gz emd_14842_half_map_2.map.gz | 21.5 MB 39.8 MB 39.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14842 http://ftp.pdbj.org/pub/emdb/structures/EMD-14842 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14842 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14842 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14842_validation.pdf.gz emd_14842_validation.pdf.gz | 698 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14842_full_validation.pdf.gz emd_14842_full_validation.pdf.gz | 697.6 KB | 表示 | |
| XML形式データ |  emd_14842_validation.xml.gz emd_14842_validation.xml.gz | 15.3 KB | 表示 | |
| CIF形式データ |  emd_14842_validation.cif.gz emd_14842_validation.cif.gz | 19.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14842 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14842 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14842 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14842 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7znyMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14842.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14842.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | final sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.29171 Å | ||||||||||||||||||||||||||||||||||||
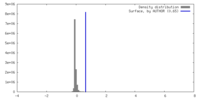
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14842_msk_1.map emd_14842_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
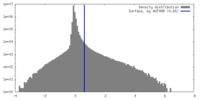
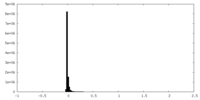
| 密度ヒストグラム |
-追加マップ: full map
| ファイル | emd_14842_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
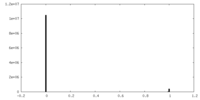
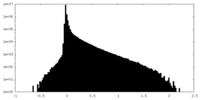
| 密度ヒストグラム |
-ハーフマップ: half map1
| ファイル | emd_14842_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
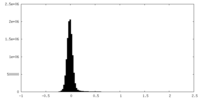
| 密度ヒストグラム |
-ハーフマップ: half map2
| ファイル | emd_14842_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
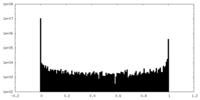
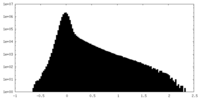
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Canine morbillivirus
| 全体 | 名称:  Canine morbillivirus (ウイルス) Canine morbillivirus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Canine morbillivirus
| 超分子 | 名称: Canine morbillivirus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 11232 / 生物種: Canine morbillivirus / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: Yes / ウイルス・中空状態: Yes |
|---|---|
| 分子量 | 理論値: 272 KDa |
-分子 #1: Hemagglutinin glycoprotein
| 分子 | 名称: Hemagglutinin glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Canine morbillivirus (ウイルス) Canine morbillivirus (ウイルス) |
| 分子量 | 理論値: 68.311031 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MLSYQDKVSA FYKDNARANS SKLSLVTEEQ GGRRPPYLLF VLLILLVGIM ALLAITGVRF HQVSTSNMEF SRLLKEDMEK SEAVHHQVI DVLTPLFKII GDEIGLRLPQ KLNEIKQFIL QKTNFFNPNR EFDFRDLHWC INPPSKIKVN FTNYCDTIGI R KSIASAAN ...文字列: MLSYQDKVSA FYKDNARANS SKLSLVTEEQ GGRRPPYLLF VLLILLVGIM ALLAITGVRF HQVSTSNMEF SRLLKEDMEK SEAVHHQVI DVLTPLFKII GDEIGLRLPQ KLNEIKQFIL QKTNFFNPNR EFDFRDLHWC INPPSKIKVN FTNYCDTIGI R KSIASAAN PILLSALSGG RGDIFPPYRC SGATTSVGRV FPLSVSLSMS LISRTSEIIN MLTAISDGVY GKTYLLVPDY IE GGFDTQK IRVFEIGFIK RWLNDMPLLQ TTNYMVLPEN SKAKVCTIAV GELTLASLCV DESTVLLYHD SDGSQDGILV VTL GIFGAT PMDQVEEVIP VAHPSVEKIH ITNHRGFIKD SIATWMVPAL VSEKQEEQKN CLESACQRKS YPMCNQTSWE PFGG GQLPS YGRLTLPLDP SIDLQLNISF TYGPVILNGD GMDYYESPLL DSGWLTIPPK NGTVLGLINK ASRGDQFTVI PHVLT FAPR ESSGNCYLPI QTSQIMDKDV LTESNLVVLP TQNFRYVIAT YDISRGDHAI VYYVYDPIRA ISYTYPFRLT TKGRPD FLR IECFVWDDDL WCHQFYRFEA DSTNSTTSVE NLVRIRFSCN RSKP |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.025 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: Blot Time: 3.5 sec Blot Force: -6 Drain Time: 0 Wait Time: 0. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 実像数: 26835 / 平均露光時間: 1.5 sec. / 平均電子線量: 50.0 e/Å2 詳細: Two Datasets from two different grids were recorded: 13,760 and 13,075 movies |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 20.0 µm / 最小 デフォーカス(公称値): 8.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7zny: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)