+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Amyloid fibril (in vitro) from full-length hnRNPA1 protein | |||||||||
 マップデータ マップデータ | Final map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Amyloidosis / Misfolding disease / Inflammation / Prion / Protein fibril / NUCLEAR PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to sodium arsenite / SARS-CoV-1-host interactions / import into nucleus / telomeric repeat-containing RNA binding / G-rich strand telomeric DNA binding / pre-mRNA binding / nuclear export / RNA export from nucleus / miRNA binding / FGFR2 alternative splicing ...cellular response to sodium arsenite / SARS-CoV-1-host interactions / import into nucleus / telomeric repeat-containing RNA binding / G-rich strand telomeric DNA binding / pre-mRNA binding / nuclear export / RNA export from nucleus / miRNA binding / FGFR2 alternative splicing / regulation of alternative mRNA splicing, via spliceosome / SARS-CoV-1 modulates host translation machinery / regulation of RNA splicing / negative regulation of telomere maintenance via telomerase / Processing of Capped Intron-Containing Pre-mRNA / mRNA transport / cellular response to glucose starvation / positive regulation of telomere maintenance via telomerase / catalytic step 2 spliceosome / mRNA Splicing - Major Pathway / mRNA 3'-UTR binding / spliceosomal complex / mRNA splicing, via spliceosome / single-stranded DNA binding / single-stranded RNA binding / ribonucleoprotein complex / protein domain specific binding / DNA binding / RNA binding / extracellular exosome / nucleoplasm / identical protein binding / membrane / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
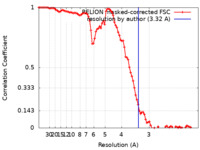
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.32 Å | |||||||||
 データ登録者 データ登録者 | Sharma K / Banerjee S / Schmidt M / Faendrich M | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2023 ジャーナル: J Mol Biol / 年: 2023タイトル: Cryo-EM Structure of the Full-length hnRNPA1 Amyloid Fibril. 著者: Kartikay Sharma / Sambhasan Banerjee / Dilan Savran / Cedric Rajes / Sebastian Wiese / Amandeep Girdhar / Nadine Schwierz / Christopher Lee / James Shorter / Matthias Schmidt / Lin Guo / Marcus Fändrich /   要旨: Heterogeneous nuclear ribonucleoprotein A1 (hnRNPA1) is a multifunctional RNA-binding protein that is associated with neurodegenerative diseases, such as amyotrophic lateral sclerosis and multisystem ...Heterogeneous nuclear ribonucleoprotein A1 (hnRNPA1) is a multifunctional RNA-binding protein that is associated with neurodegenerative diseases, such as amyotrophic lateral sclerosis and multisystem proteinopathy. In this study, we have used cryo-electron microscopy to investigate the three-dimensional structure of amyloid fibrils from full-length hnRNPA1 protein. We find that the fibril core is formed by a 45-residue segment of the prion-like low-complexity domain of the protein, whereas the remaining parts of the protein (275 residues) form a fuzzy coat around the fibril core. The fibril consists of two fibril protein stacks that are arranged into a pseudo-2 screw symmetry. The ordered core harbors several of the positions that are known to be affected by disease-associated mutations, but does not encompass the most aggregation-prone segments of the protein. These data indicate that the structures of amyloid fibrils from full-length proteins may be more complex than anticipated by current theories on protein misfolding. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14739.map.gz emd_14739.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14739-v30.xml emd-14739-v30.xml emd-14739.xml emd-14739.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14739_fsc.xml emd_14739_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14739.png emd_14739.png | 71.2 KB | ||
| Filedesc metadata |  emd-14739.cif.gz emd-14739.cif.gz | 5.8 KB | ||
| その他 |  emd_14739_half_map_1.map.gz emd_14739_half_map_1.map.gz emd_14739_half_map_2.map.gz emd_14739_half_map_2.map.gz | 49.2 MB 49.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14739 http://ftp.pdbj.org/pub/emdb/structures/EMD-14739 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14739 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14739 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zj2MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14739.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14739.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Relion half map-1
| ファイル | emd_14739_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion half map-1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Relion half map-2
| ファイル | emd_14739_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion half map-2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Amyloid fibril (in vitro) from full-length hnRNPA1 protein
| 全体 | 名称: Amyloid fibril (in vitro) from full-length hnRNPA1 protein |
|---|---|
| 要素 |
|
-超分子 #1: Amyloid fibril (in vitro) from full-length hnRNPA1 protein
| 超分子 | 名称: Amyloid fibril (in vitro) from full-length hnRNPA1 protein タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: In vitro full-length hnRNPA1 amyloid fibril |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Isoform A1-A of Heterogeneous nuclear ribonucleoprotein A1
| 分子 | 名称: Isoform A1-A of Heterogeneous nuclear ribonucleoprotein A1 タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 34.246227 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSKSESPKEP EQLRKLFIGG LSFETTDESL RSHFEQWGTL TDCVVMRDPN TKRSRGFGFV TYATVEEVDA AMNARPHKVD GRVVEPKRA VSREDSQRPG AHLTVKKIFV GGIKEDTEEH HLRDYFEQYG KIEVIEIMTD RGSGKKRGFA FVTFDDHDSV D KIVIQKYH ...文字列: MSKSESPKEP EQLRKLFIGG LSFETTDESL RSHFEQWGTL TDCVVMRDPN TKRSRGFGFV TYATVEEVDA AMNARPHKVD GRVVEPKRA VSREDSQRPG AHLTVKKIFV GGIKEDTEEH HLRDYFEQYG KIEVIEIMTD RGSGKKRGFA FVTFDDHDSV D KIVIQKYH TVNGHNCEVR KALSKQEMAS ASSSQRGRSG SGNFGGGRGG GFGGNDNFGR GGNFSGRGGF GGSRGGGGYG GS GDGYNGF GNDGSNFGGG GSYNDFGNYN NQSSNFGPMK GGNFGGRSSG PYGGGGQYFA KPRNQGGYGG SSSSSSYGSG RRF UniProtKB: Heterogeneous nuclear ribonucleoprotein A1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 2624 / 平均露光時間: 10.0 sec. / 平均電子線量: 42.64 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X