+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Endonuclease state of the E. coli Mre11-Rad50 (SbcCD) head complex bound to ADP and dsDNA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ABC-type ATPase / Nuclease / DNA repair / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報double-stranded DNA endonuclease activity / DNA exonuclease activity / single-stranded DNA endodeoxyribonuclease activity / 3'-5'-DNA exonuclease activity / DNA repair complex / 3'-5' exonuclease activity / double-strand break repair / DNA recombination / DNA replication / DNA repair ...double-stranded DNA endonuclease activity / DNA exonuclease activity / single-stranded DNA endodeoxyribonuclease activity / 3'-5'-DNA exonuclease activity / DNA repair complex / 3'-5' exonuclease activity / double-strand break repair / DNA recombination / DNA replication / DNA repair / ATP hydrolysis activity / DNA binding / ATP binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
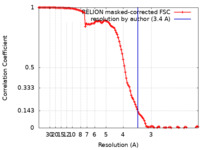
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Gut F / Kaeshammer L | |||||||||
| 資金援助 | European Union,  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2022 ジャーナル: Mol Cell / 年: 2022タイトル: Structural mechanism of endonucleolytic processing of blocked DNA ends and hairpins by Mre11-Rad50. 著者: Fabian Gut / Lisa Käshammer / Katja Lammens / Joseph D Bartho / Anna-Maria Boggusch / Erik van de Logt / Brigitte Kessler / Karl-Peter Hopfner /  要旨: DNA double-strand breaks (DSBs) threaten genome stability and are linked to tumorigenesis in humans. Repair of DSBs requires the removal of attached proteins and hairpins through a poorly understood ...DNA double-strand breaks (DSBs) threaten genome stability and are linked to tumorigenesis in humans. Repair of DSBs requires the removal of attached proteins and hairpins through a poorly understood but physiologically critical endonuclease activity by the Mre11-Rad50 complex. Here, we report cryoelectron microscopy (cryo-EM) structures of the bacterial Mre11-Rad50 homolog SbcCD bound to a protein-blocked DNA end and a DNA hairpin. The structures reveal that Mre11-Rad50 bends internal DNA for endonucleolytic cleavage and show how internal DNA, DNA ends, and hairpins are processed through a similar ATP-regulated conformational state. Furthermore, Mre11-Rad50 is loaded onto blocked DNA ends with Mre11 pointing away from the block, explaining the distinct biochemistries of 3' → 5' exonucleolytic and endonucleolytic incision through the way Mre11-Rad50 interacts with diverse DNA ends. In summary, our results unify Mre11-Rad50's enigmatic nuclease diversity within a single structural framework and reveal how blocked DNA ends and hairpins are processed. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14391.map.gz emd_14391.map.gz | 7.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14391-v30.xml emd-14391-v30.xml emd-14391.xml emd-14391.xml | 21.9 KB 21.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14391_fsc.xml emd_14391_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14391.png emd_14391.png | 123.8 KB | ||
| マスクデータ |  emd_14391_msk_1.map emd_14391_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14391.cif.gz emd-14391.cif.gz | 6.9 KB | ||
| その他 |  emd_14391_additional_1.map.gz emd_14391_additional_1.map.gz emd_14391_half_map_1.map.gz emd_14391_half_map_1.map.gz emd_14391_half_map_2.map.gz emd_14391_half_map_2.map.gz | 951.6 MB 98.4 MB 98.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14391 http://ftp.pdbj.org/pub/emdb/structures/EMD-14391 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14391 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14391 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14391_validation.pdf.gz emd_14391_validation.pdf.gz | 726.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14391_full_validation.pdf.gz emd_14391_full_validation.pdf.gz | 726.4 KB | 表示 | |
| XML形式データ |  emd_14391_validation.xml.gz emd_14391_validation.xml.gz | 18.5 KB | 表示 | |
| CIF形式データ |  emd_14391_validation.cif.gz emd_14391_validation.cif.gz | 24.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14391 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14391 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14391 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14391 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7yzoMC  7yzpC  7z03C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14391.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14391.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.059 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14391_msk_1.map emd_14391_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
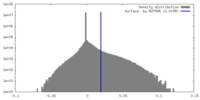
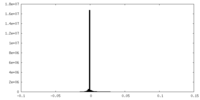
| 密度ヒストグラム |
-追加マップ: LAFTER-filtered cryo-EM map
| ファイル | emd_14391_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LAFTER-filtered cryo-EM map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
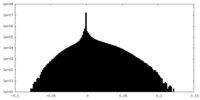
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_14391_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
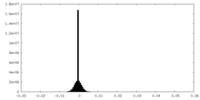
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14391_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
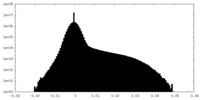
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of Mre11 dimer, Rad50 dimer and double-stranded DNA
| 全体 | 名称: Complex of Mre11 dimer, Rad50 dimer and double-stranded DNA |
|---|---|
| 要素 |
|
-超分子 #1: Complex of Mre11 dimer, Rad50 dimer and double-stranded DNA
| 超分子 | 名称: Complex of Mre11 dimer, Rad50 dimer and double-stranded DNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Nuclease SbcCD subunit C
| 分子 | 名称: Nuclease SbcCD subunit C / タイプ: protein_or_peptide / ID: 1 詳細: A large part of the structure in the middle of the sequence is missing. Therefore the alignment is not correct. The C-terminal part misaligns to the middle domain of the sequence. コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 118.851508 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKILSLRLKN LNSLKGEWKI DFTREPFASN GLFAITGPTG AGKTTLLDAI CLALYHETPR LSNVSQSQND LMTRDTAECL AEVEFEVKG EAYRAFWSQN RARNQPDGNL QVPRVELARC ADGKILADKV KDKLELTATL TGLDYGRFTR SMLLSQGQFA A FLNAKPKE ...文字列: MKILSLRLKN LNSLKGEWKI DFTREPFASN GLFAITGPTG AGKTTLLDAI CLALYHETPR LSNVSQSQND LMTRDTAECL AEVEFEVKG EAYRAFWSQN RARNQPDGNL QVPRVELARC ADGKILADKV KDKLELTATL TGLDYGRFTR SMLLSQGQFA A FLNAKPKE RAELLEELTG TEIYGQISAM VFEQHKSART ELEKLQAQAS GVTLLTPEQV QSLTASLQVL TDEEKQLITA QQ QEQQSLN WLTRQDELQQ EASRRQQALQ QALAEEEKAQ PQLAALSLAQ PARNLRPHWE RIAEHSAALA HIRQQIEEVN TRL QSTMAL RASIRHHAAK QSAELQQQQQ SLNTWLQEHD RFRQWNNEPA GWRAQFSQQT SDREHLRQWQ QQLTHAEQKL NALA AITLT LTADEVATAL AQHAEQRPLR QHLVALHGQI VPQQKRLAQL QVAIQNVTQE QTQRNAALNE MRQRYKEKTQ QLADV KTIC EQEARIKTLE AQRAQLQAGQ PCPLCGSTSH PAVEAYQALE PGVNQSRLLA LENEVKKLGE EGATLRGQLD AITKQL QRD ENEAQSLRQD EQALTQQWQA VTASLNITLQ PLDDIQPWLD AQDEHERQLR LLSQRHELQG QIAAHNQQII QYQQQIE QR QQLLLTTLTG YALTLPQEDE EESWLATRQQ EAQSWQQRQN ELTALQNRIQ QLTPILETLP QSDELPHCEE TVVLENWR Q VHEQCLALHS QQQTLQQQDV LAAQSLQKAQ AQFDTALQAS VFDDQQAFLA ALMDEQTLTQ LEQLKQNLEN QRRQAQTLV TQTAETLAQH QQHRPDDGLA LTVTVEQIQQ ELAQTHQKLR ENTTSQGEIR QQLKQDADNR QQQQTLMQQI AQMTQQVEDW GYLNSLIGS KEGDKFRKFA QGLTLDNLVH LANQQLTRLH GRYLLQRKAS EALEVEVVDT WQADAVRDTR TLSGGESFLV S LALALALS DLVSHKTRID SLFLDEGFGT LDSETLDTAL DALDALNASG KTIGVISHVE AMKERIPVQI KVKKINGLGY SK LESTFAV K UniProtKB: Nuclease SbcCD subunit C |
-分子 #2: Nuclease SbcCD subunit D
| 分子 | 名称: Nuclease SbcCD subunit D / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 45.640277 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRILHTSDWH LGQNFYSKSR EAEHQAFLDW LLETAQTHQV DAIIVAGDVF DTGSPPSYAR TLYNRFVVNL QQTGCHLVVL AGNQDSVAT LNESRDIMAF LNTTVVASAG HAPQILPRRD GTPGAVLCPI PFLRPRDIIT SQAGLNGIEK QQHLLAAITD Y YQQHYADA ...文字列: MRILHTSDWH LGQNFYSKSR EAEHQAFLDW LLETAQTHQV DAIIVAGDVF DTGSPPSYAR TLYNRFVVNL QQTGCHLVVL AGNQDSVAT LNESRDIMAF LNTTVVASAG HAPQILPRRD GTPGAVLCPI PFLRPRDIIT SQAGLNGIEK QQHLLAAITD Y YQQHYADA CKLRGDQPLP IIATGHLTTV GASKSDAVRD IYIGTLDAFP AQNFPPADYI ALGHIHRAQI IGGMEHVRYC GS PIPLSFD ECGKSKYVHL VTFSNGKLES VENLNVPVTQ PMAVLKGDLA SITAQLEQWR DVSQEPPVWL DIEITTDEYL HDI QRKIQA LTESLPVEVL LVRRSREQRE RVLASQQRET LSELSVEEVF NRRLALEELD ESQQQRLQHL FTTTLHTLAG EHEA GHHHH HH UniProtKB: Nuclease SbcCD subunit D |
-分子 #3: DNA (31-MER)
| 分子 | 名称: DNA (31-MER) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 37.099617 KDa |
| 配列 | 文字列: (DC)(DC)(DA)(DG)(DT)(DT)(DG)(DA)(DT)(DC) (DG)(DG)(DC)(DG)(DC)(DG)(DA)(DG)(DA)(DT) (DT)(DT)(DA)(DA)(DT)(DC)(DG)(DC)(DC) (DG)(DC)(DG)(DA)(DC)(DA)(DA)(DT)(DT)(DT) (DG) (DC)(DG)(DA)(DC)(DG) ...文字列: (DC)(DC)(DA)(DG)(DT)(DT)(DG)(DA)(DT)(DC) (DG)(DG)(DC)(DG)(DC)(DG)(DA)(DG)(DA)(DT) (DT)(DT)(DA)(DA)(DT)(DC)(DG)(DC)(DC) (DG)(DC)(DG)(DA)(DC)(DA)(DA)(DT)(DT)(DT) (DG) (DC)(DG)(DA)(DC)(DG)(DG)(DC)(DG) (DC)(DG)(DT)(DG)(DC)(DA)(DG)(DG)(DG)(DC) (DC)(DA) (DG)(DA)(DC)(DT)(DG)(DG)(DA) (DG)(DG)(DT)(DG)(DG)(DC)(DA)(DA)(DC)(DG) (DC)(DC)(DA) (DA)(DT)(DC)(DA)(DG)(DC) (DA)(DA)(DC)(DG)(DA)(DC)(DT)(DG)(DT)(DT) (DT)(DG)(DC)(DC) (DC)(DG)(DC)(DC)(DA) (DG)(DT)(DT)(DG)(DT)(DT)(DG)(DT)(DG)(DC) (DC)(DA)(DC)(DG)(DC) GENBANK: GENBANK: MN982376.1 |
-分子 #4: DNA (31-MER)
| 分子 | 名称: DNA (31-MER) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 36.970527 KDa |
| 配列 | 文字列: (DG)(DC)(DG)(DT)(DG)(DG)(DC)(DA)(DC)(DA) (DA)(DC)(DA)(DA)(DC)(DT)(DG)(DG)(DC)(DG) (DG)(DG)(DC)(DA)(DA)(DA)(DC)(DA)(DG) (DT)(DC)(DG)(DT)(DT)(DG)(DC)(DT)(DG)(DA) (DT) (DT)(DG)(DG)(DC)(DG) ...文字列: (DG)(DC)(DG)(DT)(DG)(DG)(DC)(DA)(DC)(DA) (DA)(DC)(DA)(DA)(DC)(DT)(DG)(DG)(DC)(DG) (DG)(DG)(DC)(DA)(DA)(DA)(DC)(DA)(DG) (DT)(DC)(DG)(DT)(DT)(DG)(DC)(DT)(DG)(DA) (DT) (DT)(DG)(DG)(DC)(DG)(DT)(DT)(DG) (DC)(DC)(DA)(DC)(DC)(DT)(DC)(DC)(DA)(DG) (DT)(DC) (DT)(DG)(DG)(DC)(DC)(DC)(DT) (DG)(DC)(DA)(DC)(DG)(DC)(DG)(DC)(DC)(DG) (DT)(DC)(DG) (DC)(DA)(DA)(DA)(DT)(DT) (DG)(DT)(DC)(DG)(DC)(DG)(DG)(DC)(DG)(DA) (DT)(DT)(DA)(DA) (DA)(DT)(DC)(DT)(DC) (DG)(DC)(DG)(DC)(DC)(DG)(DA)(DT)(DC)(DA) (DA)(DC)(DT)(DG)(DG) GENBANK: GENBANK: MN982376.1 |
-分子 #5: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 2 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #7: MANGANESE (II) ION
| 分子 | 名称: MANGANESE (II) ION / タイプ: ligand / ID: 7 / コピー数: 4 / 式: MN |
|---|---|
| 分子量 | 理論値: 54.938 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 43.19 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)