+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Mimivirus genomic fibre in its compact 5-start helix form | ||||||||||||||||||||||||
 マップデータ マップデータ | Masked postprocessed map | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報oxidoreductase activity, acting on CH-OH group of donors / flavin adenine dinucleotide binding 類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |   Acanthamoeba polyphaga mimivirus (ウイルス) Acanthamoeba polyphaga mimivirus (ウイルス) | ||||||||||||||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Villalta A / Schmitt A / Estrozi LF / Quemin ERJ / Alempic JM / Lartigue A / Prazak V / Belmudes L / Vasishtan D / Colmant AMG ...Villalta A / Schmitt A / Estrozi LF / Quemin ERJ / Alempic JM / Lartigue A / Prazak V / Belmudes L / Vasishtan D / Colmant AMG / Honore FA / Coute Y / Grunewald K / Abergel C | ||||||||||||||||||||||||
| 資金援助 |  フランス, 7件 フランス, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: The giant mimivirus 1.2 Mb genome is elegantly organized into a 30-nm diameter helical protein shield. 著者: Alejandro Villalta / Alain Schmitt / Leandro F Estrozi / Emmanuelle R J Quemin / Jean-Marie Alempic / Audrey Lartigue / Vojtěch Pražák / Lucid Belmudes / Daven Vasishtan / Agathe M G ...著者: Alejandro Villalta / Alain Schmitt / Leandro F Estrozi / Emmanuelle R J Quemin / Jean-Marie Alempic / Audrey Lartigue / Vojtěch Pražák / Lucid Belmudes / Daven Vasishtan / Agathe M G Colmant / Flora A Honoré / Yohann Couté / Kay Grünewald / Chantal Abergel /    要旨: Mimivirus is the prototype of the family of giant dsDNA viruses. Little is known about the organization of the 1.2 Mb genome inside the membrane-limited nucleoid filling the ~0.5 µm icosahedral ...Mimivirus is the prototype of the family of giant dsDNA viruses. Little is known about the organization of the 1.2 Mb genome inside the membrane-limited nucleoid filling the ~0.5 µm icosahedral capsids. Cryo-electron microscopy, cryo-electron tomography, and proteomics revealed that it is encased into a ~30-nm diameter helical protein shell surprisingly composed of two GMC-type oxidoreductases, which also form the glycosylated fibrils decorating the capsid. The genome is arranged in 5- or 6-start left-handed super-helices, with each DNA-strand lining the central channel. This luminal channel of the nucleoprotein fiber is wide enough to accommodate oxidative stress proteins and RNA polymerase subunits identified by proteomics. Such elegant supramolecular organization would represent a remarkable evolutionary strategy for packaging and protecting the genome, in a state ready for immediate transcription upon unwinding in the host cytoplasm. The parsimonious use of the same protein in two unrelated substructures of the virion is unexpected for a giant virus with thousand genes at its disposal. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14354.map.gz emd_14354.map.gz | 30.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14354-v30.xml emd-14354-v30.xml emd-14354.xml emd-14354.xml | 19.6 KB 19.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14354_fsc.xml emd_14354_fsc.xml | 13.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14354.png emd_14354.png | 76.5 KB | ||
| その他 |  emd_14354_additional_1.map.gz emd_14354_additional_1.map.gz emd_14354_half_map_1.map.gz emd_14354_half_map_1.map.gz emd_14354_half_map_2.map.gz emd_14354_half_map_2.map.gz | 164.2 MB 164.9 MB 164.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14354 http://ftp.pdbj.org/pub/emdb/structures/EMD-14354 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14354 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14354 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14354_validation.pdf.gz emd_14354_validation.pdf.gz | 788.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14354_full_validation.pdf.gz emd_14354_full_validation.pdf.gz | 788 KB | 表示 | |
| XML形式データ |  emd_14354_validation.xml.gz emd_14354_validation.xml.gz | 21.1 KB | 表示 | |
| CIF形式データ |  emd_14354_validation.cif.gz emd_14354_validation.cif.gz | 27.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14354 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14354 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14354 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14354 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14354.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14354.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Masked postprocessed map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09 Å | ||||||||||||||||||||||||||||||||||||
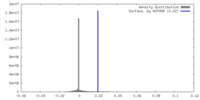
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unmasked map before postprocessing
| ファイル | emd_14354_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unmasked map before postprocessing | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unmasked half map
| ファイル | emd_14354_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unmasked half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unmasked half map
| ファイル | emd_14354_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unmasked half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mimivirus genomic fibre in its compact 5-start helix form
| 全体 | 名称: Mimivirus genomic fibre in its compact 5-start helix form |
|---|---|
| 要素 |
|
-超分子 #1: Mimivirus genomic fibre in its compact 5-start helix form
| 超分子 | 名称: Mimivirus genomic fibre in its compact 5-start helix form タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Acanthamoeba polyphaga mimivirus (ウイルス) / 株: Reunion Acanthamoeba polyphaga mimivirus (ウイルス) / 株: Reunion |
-分子 #1: Putative glucose-methanol-choline oxidoreductase protein
| 分子 | 名称: Putative glucose-methanol-choline oxidoreductase protein タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Acanthamoeba polyphaga mimivirus (ウイルス) Acanthamoeba polyphaga mimivirus (ウイルス) |
| 分子量 | 理論値: 76.431055 KDa |
| 配列 | 文字列: MAHRSRCNCN DTSNSNGSQH GINLPLRKID TYDPCVNCRV KPHLCPKPHP CPKPENLEAD IVIIGAGAAG CVLAYYLTKF SDLKIILLE AGHTHFNDPV VTDPMGFFGK YNPPNENIRM SQNPSYAWQP ALEPDTGAYS MRNVVAHGLA VGGSTAINQL N YIVGGRTV ...文字列: MAHRSRCNCN DTSNSNGSQH GINLPLRKID TYDPCVNCRV KPHLCPKPHP CPKPENLEAD IVIIGAGAAG CVLAYYLTKF SDLKIILLE AGHTHFNDPV VTDPMGFFGK YNPPNENIRM SQNPSYAWQP ALEPDTGAYS MRNVVAHGLA VGGSTAINQL N YIVGGRTV FDNDWPTGWK YDDIKKYFRR VLADISPIRD GTKVNLTNTI LESMRVLADQ QVSSGVPVDF LINKATGGLP NI EQTYQGA PIVNLNDYEG INSVCGFKSY YVGVNQLSDG SYIRKYAGNT YLNSYYVDSN GFGIGKFSNL RVISDAVVDR IHF EGQRAV SVTYIDKKGN LHSVKVHKEV EICSGSFFTP TILQRSGIGD FSYLSSIGVP DLVYNNPLVG QGLRNHYSPI TQVS VTGPD AAAFLSNTAA GPTNMSFRGA GMLGYHKLEP NKPSNAGSVT YRKYELLVTG GVAISADQQY LSGISSSTGN YFALI ADDI RFAPVGYIKI GTPNFPRDTP KIFFNTFVNY TPTTDPADQQ WPVAQKTLAP LISALLGYDA IYQIVQQMKV VAVNAG FNV TLQMAYPPND LLVELHNGLN TYGINWWHYF VPSLVNDDTP AGKLFASTLS KLSYYPRSGA HLDSHQSCSC SIGGTVD TE LKVIGVENVR VTDLSAAPHP PGGNTWCTAA MIGARATDLI LGKPLVANLP PEDVPVFTTS |
-分子 #2: FLAVIN-ADENINE DINUCLEOTIDE
| 分子 | 名称: FLAVIN-ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: FAD |
|---|---|
| 分子量 | 理論値: 785.55 Da |
| Chemical component information |  ChemComp-FAD: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 構成要素 - 濃度: 40.0 mM / 構成要素 - 式: (HOCH2)3CNH2 / 構成要素 - 名称: Tris buffer |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 50.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)