+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Tn4430 TnpA transposase from Tn3 family in complex with 100 bp long transposon end DNA | |||||||||||||||
 マップデータ マップデータ | composite map created in phenix_combine_phocused_map from consensus refinement of dimer and aligned body 2. The map was used for model refinement. | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 | Tn3 transposase DDE domain / Domain of unknown function DUF4158 / : / Tn3 transposase DDE domain / Domain of unknown function (DUF4158) / transposase activity / DNA transposition / DNA binding / Transposase for transposon Tn4430 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  | |||||||||||||||
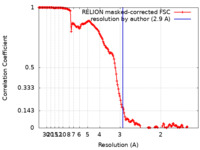
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||||||||
 データ登録者 データ登録者 | Shkumatov AV / Oger CA / Aryanpour N / Hallet BF / Efremov RG | |||||||||||||||
| 資金援助 |  ベルギー, 4件 ベルギー, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural insight into Tn3 family transposition mechanism. 著者: Alexander V Shkumatov / Nicolas Aryanpour / Cédric A Oger / Gérôme Goossens / Bernard F Hallet / Rouslan G Efremov /  要旨: Transposons are diverse mobile genetic elements that play the critical role as genome architects in all domains of life. Tn3 is a widespread family and among the first identified bacterial ...Transposons are diverse mobile genetic elements that play the critical role as genome architects in all domains of life. Tn3 is a widespread family and among the first identified bacterial transposons famed for their contribution to the dissemination of antibiotic resistance. Transposition within this family is mediated by a large TnpA transposase, which facilitates both transposition and target immunity. Howtever, a structural framework required for understanding the mechanism of TnpA transposition is lacking. Here, we describe the cryo-EM structures of TnpA from Tn4430 in the apo form and paired with transposon ends before and after DNA cleavage and strand transfer. We show that TnpA has an unusual architecture and exhibits a family specific regulatory mechanism involving metamorphic refolding of the RNase H-like catalytic domain. The TnpA structure, constrained by a double dimerization interface, creates a peculiar topology that suggests a specific role for the target DNA in transpososome assembly and activation. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13906.map.gz emd_13906.map.gz | 191.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13906-v30.xml emd-13906-v30.xml emd-13906.xml emd-13906.xml | 25.4 KB 25.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13906_fsc.xml emd_13906_fsc.xml | 15.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13906.png emd_13906.png | 102.2 KB | ||
| マスクデータ |  emd_13906_msk_1.map emd_13906_msk_1.map | 325 MB |  マスクマップ マスクマップ | |
| その他 |  emd_13906_additional_1.map.gz emd_13906_additional_1.map.gz emd_13906_half_map_1.map.gz emd_13906_half_map_1.map.gz emd_13906_half_map_2.map.gz emd_13906_half_map_2.map.gz | 6.4 MB 259.6 MB 259.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13906 http://ftp.pdbj.org/pub/emdb/structures/EMD-13906 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13906 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13906 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13906_validation.pdf.gz emd_13906_validation.pdf.gz | 695.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13906_full_validation.pdf.gz emd_13906_full_validation.pdf.gz | 694.8 KB | 表示 | |
| XML形式データ |  emd_13906_validation.xml.gz emd_13906_validation.xml.gz | 23.4 KB | 表示 | |
| CIF形式データ |  emd_13906_validation.cif.gz emd_13906_validation.cif.gz | 30.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13906 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13906 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13906 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13906 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13906.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13906.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | composite map created in phenix_combine_phocused_map from consensus refinement of dimer and aligned body 2. The map was used for model refinement. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.766 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_13906_msk_1.map emd_13906_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
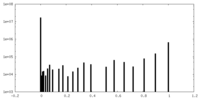
| 投影像・断面図 |
| ||||||||||||
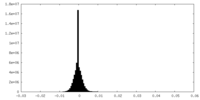
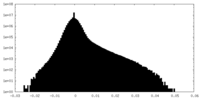
| 密度ヒストグラム |
-追加マップ: post processed map of body2 used for model...
| ファイル | emd_13906_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | post processed map of body2 used for model building. Not aligned to the map of the dimer | ||||||||||||
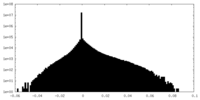
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_13906_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
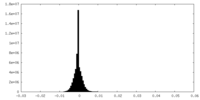
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_13906_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
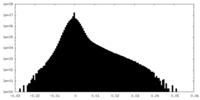
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TnpA-IR100 complex
| 全体 | 名称: TnpA-IR100 complex |
|---|---|
| 要素 |
|
-超分子 #1: TnpA-IR100 complex
| 超分子 | 名称: TnpA-IR100 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: hyperactive TnpA mutant S911R in complex with 100bp DNA containing trasnposone recognition sequence. |
|---|
-超分子 #2: hyperactive TnpA mutant S911R
| 超分子 | 名称: hyperactive TnpA mutant S911R / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 / 詳細: hyperactive TnpA mutant S911R |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-超分子 #3: DNA substrate
| 超分子 | 名称: DNA substrate / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 / 詳細: DNA substrate |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種: synthetic construct (人工物) |
-分子 #1: Transposase for transposon Tn4430
| 分子 | 名称: Transposase for transposon Tn4430 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 116.992117 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGVKQLLSEA QRNELMDLSR LTEWDLVTFH TFSKHDLHLI LKHRRGYNRL GFALQLVLIR YPGWSLTEYK DIPQYVVAYV ASQLQIPPE EFLVYAKRGN TLWEHLGEIR TEYGYQNFSS EYKETLLQFL VQQAMDNNNT LYLIEITIST LRKMKVILPA M YVIEDIVW ...文字列: MGVKQLLSEA QRNELMDLSR LTEWDLVTFH TFSKHDLHLI LKHRRGYNRL GFALQLVLIR YPGWSLTEYK DIPQYVVAYV ASQLQIPPE EFLVYAKRGN TLWEHLGEIR TEYGYQNFSS EYKETLLQFL VQQAMDNNNT LYLIEITIST LRKMKVILPA M YVIEDIVW EAKQQADQKV YSILHDGLVQ EQKDQLDALL LPTINGKSPL AWLKDVPAQP SPESFLKVID RLQFVQKIGL TI DTTKINT NRLRQLARLG SKYEPYAFRR FNEVKRYSML VSFLLEITQD LIDYAIEIHD RLMMNLQTKG KKEQDEIQQA NGK KLNEKI LQFITVCGTL IEAKETGKDA FAALDEVMSW NEMVESVEEA KQLSRPLNYD YLDLLNTRYS YVRRYAPTLL RSLH FRATK SGEPVLQALD TIHELNETGK RKVPHGAPLH FVSNRWQKHV YDDDGNINRH YYELAALTEL RNHIRSGDIF VSGSR HHKA FDDYLIPYDE WNEVSNIPNG LTAPLKAEDY ITDRINRLNE HLEWLSKNSE KLEGVDISQG KLHVERLDRG TPEEAK AFS KLLHSMLPRI KLTDLLIEVA SWTGFHDQFI HASTNQSPDQ EEQNIVLATL MAMGTNIGLT KMAEATPGIS YRQMANA SQ WRMYDDAMVR AQSILVNFQK EQKLSSYWGD GTTSSSDGMR LSIAVRSLHA DSNPHYGTGK GGTIYRFVSD QLSAYHVK V ITTNARDALH VLDGLLHHET DLKIEEHYTD TAGYTDQVFA LTHLLGFRFA PRIRDLADTK LFSIPGGEEY ENVQALLKG KINVKLIKEN YEDIRRLAYS VQTGKVSSAL IMGKLGSYAR QNKLATALGE MGRIEKTLFT LDYISNKAVR RRVQKGLNKG EAINALARI IFFGQRGEFR ERALQDQLQR ARALNIIINA ISVWNTVYME KAVEELKARG EFREDLMPYA WPLGWEHINF L GEYKFEGL HDTGQMNLRP LRIKEPFYSP IRSFLEQKLI SEEDLNSAVD HHHHHH |
-分子 #2: IR100 DNA substrate, none transferred strand
| 分子 | 名称: IR100 DNA substrate, none transferred strand / タイプ: dna / ID: 2 詳細: 100 base pairs internal DNA repeat containing recognition sequence for Tn4430 transposone. コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 30.848781 KDa |
| 配列 | 文字列: (DC)(DG)(DG)(DG)(DG)(DA)(DT)(DC)(DC)(DT) (DC)(DT)(DA)(DG)(DC)(DT)(DC)(DG)(DA)(DG) (DA)(DT)(DG)(DC)(DA)(DT)(DC)(DC)(DA) (DT)(DG)(DG)(DG)(DG)(DG)(DT)(DA)(DC)(DC) (DG) (DC)(DC)(DA)(DG)(DC) ...文字列: (DC)(DG)(DG)(DG)(DG)(DA)(DT)(DC)(DC)(DT) (DC)(DT)(DA)(DG)(DC)(DT)(DC)(DG)(DA)(DG) (DA)(DT)(DG)(DC)(DA)(DT)(DC)(DC)(DA) (DT)(DG)(DG)(DG)(DG)(DG)(DT)(DA)(DC)(DC) (DG) (DC)(DC)(DA)(DG)(DC)(DA)(DT)(DT) (DT)(DC)(DG)(DG)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DC) (DC)(DA)(DC)(DG)(DC)(DT)(DA) (DA)(DG)(DA)(DT)(DC)(DC)(DT)(DC)(DT)(DA) (DG)(DG)(DC) (DT)(DA)(DG)(DC)(DG)(DC) (DG)(DC)(DA)(DA)(DA)(DA)(DT)(DA)(DA)(DT) (DA)(DC)(DA)(DA) |
-分子 #3: IR100 DNA substrate, transferred strand
| 分子 | 名称: IR100 DNA substrate, transferred strand / タイプ: dna / ID: 3 詳細: 100 base pairs internal DNA repeat containing recognition sequence for Tn4430 transposone. コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 30.851658 KDa |
| 配列 | 文字列: (DT)(DT)(DG)(DT)(DA)(DT)(DT)(DA)(DT)(DT) (DT)(DT)(DG)(DC)(DG)(DC)(DG)(DC)(DT)(DA) (DG)(DC)(DC)(DT)(DA)(DG)(DA)(DG)(DG) (DA)(DT)(DC)(DT)(DT)(DA)(DG)(DC)(DG)(DT) (DG) (DG)(DT)(DT)(DT)(DT) ...文字列: (DT)(DT)(DG)(DT)(DA)(DT)(DT)(DA)(DT)(DT) (DT)(DT)(DG)(DC)(DG)(DC)(DG)(DC)(DT)(DA) (DG)(DC)(DC)(DT)(DA)(DG)(DA)(DG)(DG) (DA)(DT)(DC)(DT)(DT)(DA)(DG)(DC)(DG)(DT) (DG) (DG)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DC)(DC)(DG)(DA)(DA)(DA)(DT)(DG)(DC)(DT) (DG)(DG) (DC)(DG)(DG)(DT)(DA)(DC)(DC) (DC)(DC)(DC)(DA)(DT)(DG)(DG)(DA)(DT)(DG) (DC)(DA)(DT) (DC)(DT)(DC)(DG)(DA)(DG) (DC)(DT)(DA)(DG)(DA)(DG)(DG)(DA)(DT)(DC) (DC)(DC)(DC)(DG) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.15 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 50 mM HEPES (pH 7.5), 100 mM NaCl, 30 mM L-Arg HCL |
| グリッド | モデル: Quantifoil R2/1 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 4756 / 平均露光時間: 3.0 sec. / 平均電子線量: 62.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 6.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 28.8 |
|---|---|
| 得られたモデル |  PDB-7qd4: |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X