+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Pentameric ligand-gated ion channel, DeCLIC at pH 7 with 10 mM EDTA | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | ion channel / ligand-gated channel / pentameric channel / MEMBRANE PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報extracellular ligand-gated monoatomic ion channel activity / regulation of membrane potential / transmembrane signaling receptor activity / neuron projection / signal transduction / membrane / metal ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Desulfofustis sp. PB-SRB1 (バクテリア) Desulfofustis sp. PB-SRB1 (バクテリア) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | ||||||||||||
 データ登録者 データ登録者 | Lycksell M / Rovsnik U | ||||||||||||
| 資金援助 |  スウェーデン, 3件 スウェーデン, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Biophysical characterization of calcium-binding and modulatory-domain dynamics in a pentameric ligand-gated ion channel. 著者: Marie Lycksell / Urška Rovšnik / Anton Hanke / Anne Martel / Rebecca J Howard / Erik Lindahl /    要旨: Pentameric ligand-gated ion channels (pLGICs) perform electrochemical signal transduction in organisms ranging from bacteria to humans. Among the prokaryotic pLGICs, there is architectural diversity ...Pentameric ligand-gated ion channels (pLGICs) perform electrochemical signal transduction in organisms ranging from bacteria to humans. Among the prokaryotic pLGICs, there is architectural diversity involving N-terminal domains (NTDs) not found in eukaryotic relatives, exemplified by the calcium-sensitive channel (DeCLIC) from a deltaproteobacterium, which has an NTD in addition to the canonical pLGIC structure. Here, we have characterized the structure and dynamics of DeCLIC through cryoelectron microscopy (cryo-EM), small-angle neutron scattering (SANS), and molecular dynamics (MD) simulations. In the presence and absence of calcium, cryo-EM yielded structures with alternative conformations of the calcium-binding site. SANS profiles further revealed conformational diversity at room temperature beyond that observed in static structures, shown through MD to be largely attributable to rigid-body motions of the NTD relative to the protein core, with expanded and asymmetric conformations improving the fit of the SANS data. This work reveals the range of motion available to the DeCLIC NTD and calcium-binding site, expanding the conformational landscape of the pLGIC family. Further, these findings demonstrate the power of combining low-resolution scattering, high-resolution structural, and MD simulation data to elucidate interfacial interactions that are highly conserved in the pLGIC family. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13792.map.gz emd_13792.map.gz | 9.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13792-v30.xml emd-13792-v30.xml emd-13792.xml emd-13792.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_13792.png emd_13792.png | 76 KB | ||
| マスクデータ |  emd_13792_msk_1.map emd_13792_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-13792.cif.gz emd-13792.cif.gz | 5.6 KB | ||
| その他 |  emd_13792_additional_1.map.gz emd_13792_additional_1.map.gz emd_13792_half_map_1.map.gz emd_13792_half_map_1.map.gz emd_13792_half_map_2.map.gz emd_13792_half_map_2.map.gz | 49.1 MB 49.5 MB 49.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13792 http://ftp.pdbj.org/pub/emdb/structures/EMD-13792 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13792 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13792 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13792_validation.pdf.gz emd_13792_validation.pdf.gz | 863.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13792_full_validation.pdf.gz emd_13792_full_validation.pdf.gz | 863.1 KB | 表示 | |
| XML形式データ |  emd_13792_validation.xml.gz emd_13792_validation.xml.gz | 12.3 KB | 表示 | |
| CIF形式データ |  emd_13792_validation.cif.gz emd_13792_validation.cif.gz | 14.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13792 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13792 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13792 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13792 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7q3hMC  7q3gC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13792.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13792.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
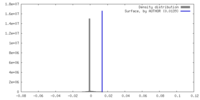
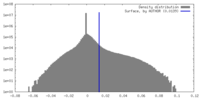
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_13792_msk_1.map emd_13792_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_13792_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13792_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_13792_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pentameric ligand-gated ion channel DeCLIC
| 全体 | 名称: Pentameric ligand-gated ion channel DeCLIC |
|---|---|
| 要素 |
|
-超分子 #1: Pentameric ligand-gated ion channel DeCLIC
| 超分子 | 名称: Pentameric ligand-gated ion channel DeCLIC / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Desulfofustis sp. PB-SRB1 (バクテリア) Desulfofustis sp. PB-SRB1 (バクテリア) |
| 分子量 | 理論値: 364.31 kDa/nm |
-分子 #1: Neur_chan_LBD domain-containing protein
| 分子 | 名称: Neur_chan_LBD domain-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Desulfofustis sp. PB-SRB1 (バクテリア) Desulfofustis sp. PB-SRB1 (バクテリア) |
| 分子量 | 理論値: 71.733992 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHNLQQLLPT RSLIWIFSFL TSISIWCTVA HAETEGRVQH FTGYIEDGRG IFYSLPDMKQ GDIIYASMQN TGGNLDPLVG IMAEEIDPA VSLGQVLEKA LASENDLISE LTAVADRIFL GWDDDGGKGY SASLEFTIPR DGTYHIFAGS TITNQRLDKF Q PTYTTGSF ...文字列: MHNLQQLLPT RSLIWIFSFL TSISIWCTVA HAETEGRVQH FTGYIEDGRG IFYSLPDMKQ GDIIYASMQN TGGNLDPLVG IMAEEIDPA VSLGQVLEKA LASENDLISE LTAVADRIFL GWDDDGGKGY SASLEFTIPR DGTYHIFAGS TITNQRLDKF Q PTYTTGSF QLILGLNAPQ VISGEGEPEG EVFASLASLE IKPEAHVQEL EIRLDKDTRY LTQHTRNLQP GDTFHALVEP IG EAPLPRL RLTDSGGKPL AFGLIDQPGE SVELNYTCDQ DICELVVHVD GTDGQKDSGE AVYRLLVGIN APNLRESGQT PVG SSVFLE SDLVTVGLAV DQIVGVDQRS ENFSVVGTLK LSWHDPKLGF SPDQCGCTVK SFEDASIRAV AGEINLPLPS FSFY NQQGN RWSQNQVIFV TPDGRASYFE RFTVTLQAPD FDFLAYPFDR QKFSIKVDLA VPTNMFIFNE IERFQQVVGD QLGEE EWVV TSYSQEITEV PFERGSTNSR FTTTLLVKRN LEYYILRIFV PLFLIISVSW VIFFLKDYGR QLEVASGNLL VFVAFN FTI SGDLPRLGYL TVLDRFMIVS FCLTAIVVLI SVCQKRLGAV GKQAVAAQID TWVLVIYPLV YSLYIIWVYL RFFTDHI GW UniProtKB: Neurotransmitter-gated ion-channel ligand-binding domain-containing protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 平均露光時間: 6.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 11000 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)