+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
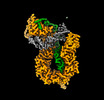
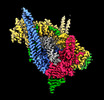
| タイトル | Cryo-EM structure of S.cerevisiae condensin Ycg1-Brn1-DNA complex | ||||||||||||
 マップデータ マップデータ | cropped box=162pix | ||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of meiotic DNA double-strand break formation / meiotic chromosome condensation / Condensation of Prometaphase Chromosomes / tRNA gene clustering / meiotic chromosome separation / condensin complex / rDNA chromatin condensation / synaptonemal complex assembly / mitotic chromosome condensation / chromosome condensation ...negative regulation of meiotic DNA double-strand break formation / meiotic chromosome condensation / Condensation of Prometaphase Chromosomes / tRNA gene clustering / meiotic chromosome separation / condensin complex / rDNA chromatin condensation / synaptonemal complex assembly / mitotic chromosome condensation / chromosome condensation / mitotic sister chromatid segregation / condensed chromosome / cell division / chromatin binding / nucleus / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | ||||||||||||
 データ登録者 データ登録者 | Lee B-G / Rhodes J / Lowe J | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Clamping of DNA shuts the condensin neck gate. 著者: Byung-Gil Lee / James Rhodes / Jan Löwe /  要旨: SignificanceDNA needs to be compacted to fit into nuclei and during cell division, when dense chromatids are formed for their mechanical segregation, a process that depends on the protein complex ...SignificanceDNA needs to be compacted to fit into nuclei and during cell division, when dense chromatids are formed for their mechanical segregation, a process that depends on the protein complex condensin. It forms and enlarges loops in DNA through loop extrusion. Our work resolves the atomic structure of a DNA-bound state of condensin in which ATP has not been hydrolyzed. The DNA is clamped within a compartment that has been reported previously in other structural maintenance of chromosomes (SMC) complexes, including Rad50, cohesin, and MukBEF. With the caveat of important differences, it means that all SMC complexes cycle through at least some similar states and undergo similar conformational changes in their head modules, while hydrolyzing ATP and translocating DNA. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13786.map.gz emd_13786.map.gz | 15.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13786-v30.xml emd-13786-v30.xml emd-13786.xml emd-13786.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_13786.png emd_13786.png | 86.7 KB | ||
| その他 |  emd_13786_half_map_1.map.gz emd_13786_half_map_1.map.gz emd_13786_half_map_2.map.gz emd_13786_half_map_2.map.gz | 15.1 MB 15.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13786 http://ftp.pdbj.org/pub/emdb/structures/EMD-13786 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13786 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13786 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13786_validation.pdf.gz emd_13786_validation.pdf.gz | 569.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13786_full_validation.pdf.gz emd_13786_full_validation.pdf.gz | 569.2 KB | 表示 | |
| XML形式データ |  emd_13786_validation.xml.gz emd_13786_validation.xml.gz | 9.5 KB | 表示 | |
| CIF形式データ |  emd_13786_validation.cif.gz emd_13786_validation.cif.gz | 11 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13786 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13786 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13786 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13786 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7q2zMC  7q2xC  7q2yC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13786.map.gz / 形式: CCP4 / 大きさ: 16.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13786.map.gz / 形式: CCP4 / 大きさ: 16.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cropped box=162pix | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: cropped box=162pix
| ファイル | emd_13786_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cropped box=162pix | ||||||||||||
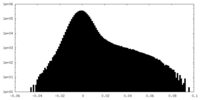
| 投影像・断面図 |
| ||||||||||||
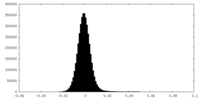
| 密度ヒストグラム |
-ハーフマップ: cropped box=162pix
| ファイル | emd_13786_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cropped box=162pix | ||||||||||||
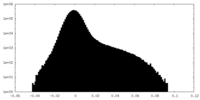
| 投影像・断面図 |
| ||||||||||||
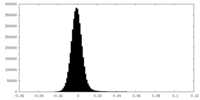
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : complex of condensin ycg1 and DNA
| 全体 | 名称: complex of condensin ycg1 and DNA |
|---|---|
| 要素 |
|
-超分子 #1: complex of condensin ycg1 and DNA
| 超分子 | 名称: complex of condensin ycg1 and DNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: ycg1-DNA structure from the dataset of complex of condensin pentamer (smc2, smc4, brn1, ycs4, ycg1), DNA, ADP-BeF3 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 実験値: 120 kDa/nm |
-分子 #1: Condensin complex subunit 3
| 分子 | 名称: Condensin complex subunit 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 117.981 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MQDPDGIDIN TKIFNSVAEV FQKAQGSYAG HRKHIAVLKK IQSKAVEQGY EDAFNFWFDK LVTKILPLKK NEIIGDRIVK LVAAFIASL ERELILAKKQ NYKLTNDEEG IFSRFVDQFI RHVLRGVESP DKNVRFRVLQ LLAVIMDNIG EIDESLFNLL I LSLNKRIY ...文字列: MQDPDGIDIN TKIFNSVAEV FQKAQGSYAG HRKHIAVLKK IQSKAVEQGY EDAFNFWFDK LVTKILPLKK NEIIGDRIVK LVAAFIASL ERELILAKKQ NYKLTNDEEG IFSRFVDQFI RHVLRGVESP DKNVRFRVLQ LLAVIMDNIG EIDESLFNLL I LSLNKRIY DREPTVRIQA VFCLTKFQDE EQTEHLTELS DNEENFEATR TLVASIQNDP SAEVRRAAML NLINDNNTRP YI LERARDV NIVNRRLVYS RILKSMGRKC FDDIEPHIFD QLIEWGLEDR ELSVRNACKR LIAHDWLNAL DGDLIELLEK LDV SRSSVC VKAIEALFQS RPDILSKIKF PESIWKDFTV EIAFLFRAIY LYCLDNNITE MLEENFPEAS KLSEHLNHYI LLRY HHNDI SNDSQSHFDY NTLEFIIEQL SIAAERYDYS DEVGRRSMLT VVRNMLALTT LSEPLIKIGI RVMKSLSINE KDFVT MAIE IINDIRDDDI EKQEQEEKIK SKKINRRNET SVDEEDENGT HNDEVNEDEE DDNISSFHSA VENLVQGNGN VSESDI INN LPPEKEASSA TIVLCLTRSS YMLELVNTPL TENILIASLM DTLITPAVRN TAPNIRELGV KNLGLCCLLD VKLAIDN MY ILGMCVSKGN ASLKYIALQV IVDIFSVHGN TVVDGEGKVD SISLHKIFYK VLKNNGLPEC QVIAAEGLCK LFLADVFT D DDLFETLVLS YFSPINSSNE ALVQAFAFCI PVYCFSHPAH QQRMSRTAAD ILLRLCVLWD DLQSSVIPEV DREAMLKPN IIFQQLLFWT DPRNLVNQTG STKKDTVQLT FLIDVLKIYA QIEKKEIKKM IITNINAIFL SSEQDYSTLK ELLEYSDDIA ENDNLDNVS KNALDKLRNN LNSLIEEINE RSETQTKDEN NTANDQYSSI LGNSFNKSSN DTIEHAADIT DGNNTELTKT T VNISAVDN TTEQSNSRKR TRSEAEQIDT SKNLENMSIQ DTSTVAKNVS FVLPDEKSDA MSIDEEDKDS ESFSEVC |
-分子 #2: Condensin complex subunit 2
| 分子 | 名称: Condensin complex subunit 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 86.323297 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTTQLRYENN DDDERVEYNL FTNRSTMMAN FEEWIKMATD NKINSRNSWN FALIDYFYDL DVLKDGENNI NFQKASATLD GCIKIYSSR VDSVTTETGK LLSGLAQRKT NGASNGDDSN GGNGEGLGGD SDEANIEIDP LTGMPISNDP DVNNTRRRVY N RVLETTLV ...文字列: MTTQLRYENN DDDERVEYNL FTNRSTMMAN FEEWIKMATD NKINSRNSWN FALIDYFYDL DVLKDGENNI NFQKASATLD GCIKIYSSR VDSVTTETGK LLSGLAQRKT NGASNGDDSN GGNGEGLGGD SDEANIEIDP LTGMPISNDP DVNNTRRRVY N RVLETTLV EFETIKMKEL DQELIIDPLF KKALVDFDEG GAKSLLLNTL NIDNTARVIF DASIKDTQNV GQGKLQRKEE EL IERDSLV DDENEPSQSL ISTRNDSTVN DSVISAPSME DEILSLGMDF IKFDQIAVCE ISGSIEQLRN VVEDINQAKD FIE NVNNRF DNFLTEEELQ AAVPDNAEDD SDGFDMGMQQ ELCYPDENHD NTSHDEQDDD NVNSTTGSIF EKDLMAYFDE NLNR NWRGR EHWKVRNFKK ANLVNKESDL LEETRTTIGD TTDKNTTDDK SMDTKKKHKQ KKVLEIDFFK TDDSFEDKVF ASKGR TKID MPIKNRKNDT HYLLPDDFHF STDRITRLFI KPGQKMSLFS HRKHTRGDVS SGLFEKSTVS ANHSNNDIPT IADEHF WAD NYERKEQEEK EKEQSKEVGD VVGGALDNPF EDDMDGVDFN QAFEGTDDNE EASVKLDLQD DEDHKFPIRE NKVTYSR VS KKVDVRRLKK NVWRSINNLI QEHDSRKNRE QSSNDSETHT EDESTKELKF SDIIQGISKM YSDDTLKDIS TSFCFICL L HLANEHGLQI THTENYNDLI VNYEDLATTQ AAS |
-分子 #3: DNA
| 分子 | 名称: DNA / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 6.951477 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT) |
-分子 #4: DNA
| 分子 | 名称: DNA / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 7.1588 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: CTFFIND (ver. 4) |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 91024 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア: (名称: cryoSPARC, RELION) |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: RELION |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X