+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13642 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Alpha-latrocrustotoxin monomer | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Pore-forming toxin / TOXIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報other organism cell membrane / host cell presynaptic membrane / exocytosis / toxin activity / extracellular region / membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Latrodectus tredecimguttatus (クモ) Latrodectus tredecimguttatus (クモ) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.03 Å | ||||||||||||
 データ登録者 データ登録者 | Chen M / Gatsogiannis C | ||||||||||||
| 資金援助 |  日本, 3件 日本, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Molecular architecture of black widow spider neurotoxins. 著者: Minghao Chen / Daniel Blum / Lena Engelhard / Stefan Raunser / Richard Wagner / Christos Gatsogiannis /  要旨: Latrotoxins (LaTXs) are presynaptic pore-forming neurotoxins found in the venom of Latrodectus spiders. The venom contains a toxic cocktail of seven LaTXs, with one of them targeting vertebrates (α- ...Latrotoxins (LaTXs) are presynaptic pore-forming neurotoxins found in the venom of Latrodectus spiders. The venom contains a toxic cocktail of seven LaTXs, with one of them targeting vertebrates (α-latrotoxin (α-LTX)), five specialized on insects (α, β, γ, δ, ε- latroinsectotoxins (LITs), and one on crustaceans (α-latrocrustatoxin (α-LCT)). LaTXs bind to specific receptors on the surface of neuronal cells, inducing the release of neurotransmitters either by directly stimulating exocytosis or by forming Ca-conductive tetrameric pores in the membrane. Despite extensive studies in the past decades, a high-resolution structure of a LaTX is not yet available and the precise mechanism of LaTX action remains unclear. Here, we report cryoEM structures of the α-LCT monomer and the δ-LIT dimer. The structures reveal that LaTXs are organized in four domains. A C-terminal domain of ankyrin-like repeats shields a central membrane insertion domain of six parallel α-helices. Both domains are flexibly linked via an N-terminal α-helical domain and a small β-sheet domain. A comparison between the structures suggests that oligomerization involves major conformational changes in LaTXs with longer C-terminal domains. Based on our data we propose a cyclic mechanism of oligomerization, taking place prior membrane insertion. Both recombinant α-LCT and δ-LIT form channels in artificial membrane bilayers, that are stabilized by Ca ions and allow calcium flux at negative membrane potentials. Our comparative analysis between α-LCT and δ-LIT provides first crucial insights towards understanding the molecular mechanism of the LaTX family. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13642.map.gz emd_13642.map.gz | 2.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13642-v30.xml emd-13642-v30.xml emd-13642.xml emd-13642.xml | 12.4 KB 12.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_13642.png emd_13642.png | 69.7 KB | ||
| Filedesc metadata |  emd-13642.cif.gz emd-13642.cif.gz | 6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13642 http://ftp.pdbj.org/pub/emdb/structures/EMD-13642 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13642 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13642 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13642_validation.pdf.gz emd_13642_validation.pdf.gz | 326.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13642_full_validation.pdf.gz emd_13642_full_validation.pdf.gz | 325.8 KB | 表示 | |
| XML形式データ |  emd_13642_validation.xml.gz emd_13642_validation.xml.gz | 6.7 KB | 表示 | |
| CIF形式データ |  emd_13642_validation.cif.gz emd_13642_validation.cif.gz | 7.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13642 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13642 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13642 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13642 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ptxMC  7ptyC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10827 (タイトル: CryoEM single particle dataset of alpha-latrocrustotoxin monomer EMPIAR-10827 (タイトル: CryoEM single particle dataset of alpha-latrocrustotoxin monomerData size: 5.1 TB Data #1: Raw images of the alpha-latrocrustatoxin monomer part1 [micrographs - multiframe] Data #2: Raw images of the alpha-latrocrustatoxin monomer part2 [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13642.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13642.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.9 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
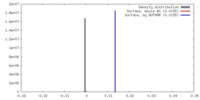
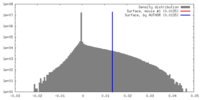
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Alpha-latrocrustotoxin-Lt1a in soluble monomeric state
| 全体 | 名称: Alpha-latrocrustotoxin-Lt1a in soluble monomeric state |
|---|---|
| 要素 |
|
-超分子 #1: Alpha-latrocrustotoxin-Lt1a in soluble monomeric state
| 超分子 | 名称: Alpha-latrocrustotoxin-Lt1a in soluble monomeric state タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Latrodectus tredecimguttatus (クモ) Latrodectus tredecimguttatus (クモ) |
| 分子量 | 理論値: 139.8 KDa |
-分子 #1: Alpha-latrocrustotoxin-Lt1a
| 分子 | 名称: Alpha-latrocrustotoxin-Lt1a / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Latrodectus tredecimguttatus (クモ) Latrodectus tredecimguttatus (クモ) |
| 分子量 | 理論値: 140.010531 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MWSHPQFEKG SAGSAAGSGA GWSHPQFEKG AGLEVLFQGP NSEMKGKRVI SKREMSKADQ CTFLSYQSVA YGTLGDVAGD VSSIEGADL VATPIAAGGH LAKGATDAAM IAMDCSSIPF DEIKQQLNQR FNEVDKKLQK GAEALENVTE LAEKTYSSVE K MRVEMREG ...文字列: MWSHPQFEKG SAGSAAGSGA GWSHPQFEKG AGLEVLFQGP NSEMKGKRVI SKREMSKADQ CTFLSYQSVA YGTLGDVAGD VSSIEGADL VATPIAAGGH LAKGATDAAM IAMDCSSIPF DEIKQQLNQR FNEVDKKLQK GAEALENVTE LAEKTYSSVE K MRVEMREG FNHVIATIEN ANTKQIITGI NQIIQYFNDE RENINNRQKE DYVAKLQEPA SGNFLLYLRK SRTSEDGSLH SL LFKIINQ ELAIPNNAAD NNAIRALFAL FYGTQTFISI MFYLVKQYSY LADYHYQNGN LAEFNSNFDH MKTVFQDFKF TLI GINTSN SKPLVNTVLS IIEDVKNKRF IRNLRSNLYQ KIIKSTKSLL DLREKITKMD LPIIEDTPKS SVLINFREKS SSVP RIETP ILKWTPGTVV KYAIQYEQDG KYSKISKWSN PITVQRLANP YITIDKDRRN RLVFRQFGNE KPELISILDS SQNEF RDIH RDLYNAAQMP YKETALGICR KLIDSGAQVG ASFEMGRKSI HASATAGNDD VARLLLAKNN GLLNVPDKNG YTPLHI ASE RKNNDFVKFL LEKGADVNVR TFANELTPLH LAARQDFTII VKTLMEKRGI DVNAKERAGF TPLHLSITSN SRAARTL IN ETPAGINIKS NSGLTPLHLA VLQNNLSAAK VLVKSNKKVK LNEMDNNGMT PLHYASMLGN LEFVKYFTSE QGIDVNAK T KVKNWTPLHL AILFKKFDVA QSLLQVRNID ISTRADQAIT PLHLAAATGN SQIVKTILNS GAVVDQETAN GFTALHLAI MNPNTETPQF LIAKGANINA KTNDGSTPLH FAAALGKTNI FQLLMDKGAN IKAENLINQM PIHEAVVNGH LAIVKMLIEQ DSSLMNAKN MRDEYPFYLA AEKRYKDVFN YLESKGADVN EKNNDGNTLL HLFSINGEVE VVQFLIQNGA DFRLRNKERK S FFDLAVEF GHAGIVGYAI EENKVDLQEP YRGKTILYHA ICDSVKYDRI EVVRYFVETL NEDQCSPLQE AAAYAHLDLV KY FVQERGI NPTAFNNDNQ VSPLCIAIVG APCGFVKSCD TPERLDVVEY LVDKTPDINK ECDTQQSTPV SSAVYGNKVS ILN YLIRNG ADPNKKVRGD PPLFIAAMIG QYDIVKSLVE QHKIDVNTRN KEQFTPLHAA ASNDHIDVVK YLIQKGADVN AKGD ENLKP IDLAGEKSKA YLRSLGRRFF RNESPSKSFE ISSGLVPRGS HHHHHHHH UniProtKB: Alpha-latrocrustotoxin-Lt1a |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 1 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 69.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7ptx: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)