+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structures of human fucosidase FucA1 reveal insight into substate recognition and catalysis. | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alpha-L-fucosidase / glycolipid catabolic process / glycosaminoglycan catabolic process / Reactions specific to the complex N-glycan synthesis pathway / alpha-L-fucosidase activity / fucose metabolic process / glycoside catabolic process / lysosomal lumen / azurophil granule lumen / lysosome ...alpha-L-fucosidase / glycolipid catabolic process / glycosaminoglycan catabolic process / Reactions specific to the complex N-glycan synthesis pathway / alpha-L-fucosidase activity / fucose metabolic process / glycoside catabolic process / lysosomal lumen / azurophil granule lumen / lysosome / intracellular membrane-bounded organelle / Neutrophil degranulation / extracellular exosome / extracellular region / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.49 Å | |||||||||||||||
 データ登録者 データ登録者 | Armstrong Z / Meek RW / Wu L / Blaza JN / Davies GJ | |||||||||||||||
| 資金援助 |  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2022 ジャーナル: Structure / 年: 2022タイトル: Cryo-EM structures of human fucosidase FucA1 reveal insight into substrate recognition and catalysis. 著者: Zachary Armstrong / Richard W Meek / Liang Wu / James N Blaza / Gideon J Davies /  要旨: Enzymatic hydrolysis of α-L-fucose from fucosylated glycoconjugates is consequential in bacterial infections and the neurodegenerative lysosomal storage disorder fucosidosis. Understanding human α- ...Enzymatic hydrolysis of α-L-fucose from fucosylated glycoconjugates is consequential in bacterial infections and the neurodegenerative lysosomal storage disorder fucosidosis. Understanding human α-L-fucosidase catalysis, in an effort toward drug design, has been hindered by the absence of three-dimensional structural data for any animal fucosidase. Here, we have used cryoelectron microscopy (cryo-EM) to determine the structure of human lysosomal α-L-fucosidase (FucA1) in both an unliganded state and in complex with the inhibitor deoxyfuconojirimycin. These structures, determined at 2.49 Å resolution, reveal the homotetrameric structure of FucA1, the architecture of the catalytic center, and the location of both natural population variations and disease-causing mutations. Furthermore, this work has conclusively identified the hitherto contentious identity of the catalytic acid/base as aspartate-276, representing a shift from both the canonical glutamate acid/base residue and a previously proposed glutamate residue. These findings have furthered our understanding of how FucA1 functions in both health and disease. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13520.map.gz emd_13520.map.gz | 21.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13520-v30.xml emd-13520-v30.xml emd-13520.xml emd-13520.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_13520.png emd_13520.png | 159.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13520 http://ftp.pdbj.org/pub/emdb/structures/EMD-13520 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13520 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13520 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13520_validation.pdf.gz emd_13520_validation.pdf.gz | 325.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13520_full_validation.pdf.gz emd_13520_full_validation.pdf.gz | 325 KB | 表示 | |
| XML形式データ |  emd_13520_validation.xml.gz emd_13520_validation.xml.gz | 7.5 KB | 表示 | |
| CIF形式データ |  emd_13520_validation.cif.gz emd_13520_validation.cif.gz | 8.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13520 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13520 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13520 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13520 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7pm4MC  7plsC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13520.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13520.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
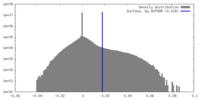
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.656 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : FucA1 homotetramer in complex with deoxyfuconojirimycin
| 全体 | 名称: FucA1 homotetramer in complex with deoxyfuconojirimycin |
|---|---|
| 要素 |
|
-超分子 #1: FucA1 homotetramer in complex with deoxyfuconojirimycin
| 超分子 | 名称: FucA1 homotetramer in complex with deoxyfuconojirimycin タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 分子量 | 実験値: 225 kDa/nm |
-分子 #1: Tissue alpha-L-fucosidase
| 分子 | 名称: Tissue alpha-L-fucosidase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: alpha-L-fucosidase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 50.525625 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GQPPRRYTPD WPSLDSRPLP AWFDEAKFGV FIHWGVFSVP AWGSEWFWWH WQGEGRPQYQ RFMRDNYPPG FSYADFGPQF TARFFHPEE WADLFQAAGA KYVVLTTKHH EGFTNWPSPV SWNWNSKDVG PHRDLVGELG TALRKRNIRY GLYHSLLEWF H PLYLLDKK ...文字列: GQPPRRYTPD WPSLDSRPLP AWFDEAKFGV FIHWGVFSVP AWGSEWFWWH WQGEGRPQYQ RFMRDNYPPG FSYADFGPQF TARFFHPEE WADLFQAAGA KYVVLTTKHH EGFTNWPSPV SWNWNSKDVG PHRDLVGELG TALRKRNIRY GLYHSLLEWF H PLYLLDKK NGFKTQHFVS AKTMPELYDL VNSYKPDLIW SDGEWECPDT YWNSTNFLSW LYNDSPVKDE VVVNDRWGQN CS CHHGGYY NCEDKFKPQS LPDHKWEMCT SIDKFSWGYR RDMALSDVTE ESEIISELVQ TVSLGGNYLL NIGPTKDGLI VPI FQERLL AVGKWLSING EAIYASKPWR VQWEKNTTSV WYTSKGSAVY AIFLHWPENG VLNLESPITT STTKITMLGI QGDL KWSTD PDKGLFISLP QLPPSAVPAE FAWTIKLTGV K |
-分子 #2: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 4 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #3: (2S,3R,4S,5R)-2-METHYLPIPERIDINE-3,4,5-TRIOL
| 分子 | 名称: (2S,3R,4S,5R)-2-METHYLPIPERIDINE-3,4,5-TRIOL / タイプ: ligand / ID: 3 / コピー数: 4 / 式: DFU |
|---|---|
| 分子量 | 理論値: 147.172 Da |
| Chemical component information | 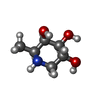 ChemComp-DFU: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 60 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 4.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 2 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | ホルダー冷却材: NITROGEN |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: D2 (2回x2回 2面回転対称) 解像度のタイプ: BY AUTHOR / 解像度: 2.49 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.1.2) / 使用した粒子像数: 98815 |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)