+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a fully assembled T-cell receptor engaging a tumor-associated peptide-MHC I | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of lymphocyte apoptotic process / gamma-delta T cell receptor complex / Fc-gamma receptor III complex / T cell anergy / positive regulation of T cell anergy / positive regulation of cell-cell adhesion mediated by integrin / cis-Golgi network membrane / positive regulation of melanin biosynthetic process / CD4-positive, alpha-beta T cell proliferation / Fc-gamma receptor signaling pathway ...regulation of lymphocyte apoptotic process / gamma-delta T cell receptor complex / Fc-gamma receptor III complex / T cell anergy / positive regulation of T cell anergy / positive regulation of cell-cell adhesion mediated by integrin / cis-Golgi network membrane / positive regulation of melanin biosynthetic process / CD4-positive, alpha-beta T cell proliferation / Fc-gamma receptor signaling pathway / gamma-delta T cell activation / negative thymic T cell selection / melanin biosynthetic process / melanosome membrane / multivesicular body, internal vesicle / melanosome organization / positive regulation of CD4-positive, alpha-beta T cell proliferation / multivesicular body membrane / alpha-beta T cell receptor complex / positive thymic T cell selection / positive regulation of protein localization to cell surface / signal complex assembly / Nef and signal transduction / positive regulation of cell-matrix adhesion / T cell mediated cytotoxicity directed against tumor cell target / T cell receptor complex / positive regulation of memory T cell activation / TAP complex binding / smoothened signaling pathway / establishment or maintenance of cell polarity / positive regulation of CD8-positive, alpha-beta T cell activation / CD8-positive, alpha-beta T cell activation / Golgi medial cisterna / positive regulation of CD8-positive, alpha-beta T cell proliferation / CD8 receptor binding / positive regulation of interleukin-4 production / dendrite development / Translocation of ZAP-70 to Immunological synapse / Phosphorylation of CD3 and TCR zeta chains / antigen processing and presentation of exogenous peptide antigen via MHC class I / endoplasmic reticulum exit site / alpha-beta T cell activation / antigen processing and presentation of endogenous peptide antigen via MHC class I via ER pathway, TAP-dependent / Generation of second messenger molecules / immunological synapse / TAP binding / FCGR activation / protection from natural killer cell mediated cytotoxicity / PD-1 signaling / Role of phospholipids in phagocytosis / beta-2-microglobulin binding / T cell receptor binding / positive regulation of calcium-mediated signaling / negative regulation of smoothened signaling pathway / detection of bacterium / positive regulation of T cell proliferation / cell surface receptor protein tyrosine kinase signaling pathway / T cell costimulation / T cell activation / positive regulation of interleukin-2 production / bioluminescence / cerebellum development / FCGR3A-mediated IL10 synthesis / protein tyrosine kinase binding / positive regulation of ferrous iron binding / positive regulation of transferrin receptor binding / positive regulation of receptor binding / early endosome lumen / Nef mediated downregulation of MHC class I complex cell surface expression / DAP12 interactions / negative regulation of receptor binding / generation of precursor metabolites and energy / antigen processing and presentation of endogenous peptide antigen via MHC class I via ER pathway, TAP-independent / antigen processing and presentation of endogenous peptide antigen via MHC class Ib / calcium-mediated signaling / lumenal side of endoplasmic reticulum membrane / cellular response to iron ion / Endosomal/Vacuolar pathway / apoptotic signaling pathway / Antigen Presentation: Folding, assembly and peptide loading of class I MHC / FCGR3A-mediated phagocytosis / cellular response to iron(III) ion / clathrin-coated endocytic vesicle membrane / antigen processing and presentation of exogenous protein antigen via MHC class Ib, TAP-dependent / negative regulation of forebrain neuron differentiation / regulation of erythrocyte differentiation / ER to Golgi transport vesicle membrane / peptide antigen assembly with MHC class I protein complex / regulation of iron ion transport / response to molecule of bacterial origin / MHC class I peptide loading complex / HFE-transferrin receptor complex / T cell mediated cytotoxicity / antigen processing and presentation of endogenous peptide antigen via MHC class I / positive regulation of T cell cytokine production / Regulation of actin dynamics for phagocytic cup formation / MHC class I protein complex / multicellular organismal-level iron ion homeostasis / negative regulation of neurogenesis / peptide antigen assembly with MHC class II protein complex 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||||||||
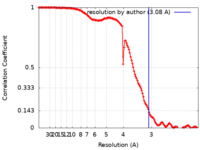
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.08 Å | |||||||||||||||
 データ登録者 データ登録者 | Susac L / Thomas C / Tampe R | |||||||||||||||
| 資金援助 |  ドイツ, 4件 ドイツ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2022 ジャーナル: Cell / 年: 2022タイトル: Structure of a fully assembled tumor-specific T cell receptor ligated by pMHC. 著者: Lukas Sušac / Mai T Vuong / Christoph Thomas / Sören von Bülow / Caitlin O'Brien-Ball / Ana Mafalda Santos / Ricardo A Fernandes / Gerhard Hummer / Robert Tampé / Simon J Davis /   要旨: The T cell receptor (TCR) expressed by T lymphocytes initiates protective immune responses to pathogens and tumors. To explore the structural basis of how TCR signaling is initiated when the ...The T cell receptor (TCR) expressed by T lymphocytes initiates protective immune responses to pathogens and tumors. To explore the structural basis of how TCR signaling is initiated when the receptor binds to peptide-loaded major histocompatibility complex (pMHC) molecules, we used cryogenic electron microscopy to determine the structure of a tumor-reactive TCRαβ/CD3δγεζ complex bound to a melanoma-specific human class I pMHC at 3.08 Å resolution. The antigen-bound complex comprises 11 subunits stabilized by multivalent interactions across three structural layers, with clustered membrane-proximal cystines stabilizing the CD3-εδ and CD3-εγ heterodimers. Extra density sandwiched between transmembrane helices reveals the involvement of sterol lipids in TCR assembly. The geometry of the pMHC/TCR complex suggests that efficient TCR scanning of pMHC requires accurate pre-positioning of T cell and antigen-presenting cell membranes. Comparisons of the ligand-bound and unliganded receptors, along with molecular dynamics simulations, indicate that TCRs can be triggered in the absence of spontaneous structural rearrangements. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13427.map.gz emd_13427.map.gz | 204.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13427-v30.xml emd-13427-v30.xml emd-13427.xml emd-13427.xml | 19 KB 19 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13427_fsc.xml emd_13427_fsc.xml | 13.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13427.png emd_13427.png | 91.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13427 http://ftp.pdbj.org/pub/emdb/structures/EMD-13427 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13427 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13427 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13427_validation.pdf.gz emd_13427_validation.pdf.gz | 450.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13427_full_validation.pdf.gz emd_13427_full_validation.pdf.gz | 450.2 KB | 表示 | |
| XML形式データ |  emd_13427_validation.xml.gz emd_13427_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  emd_13427_validation.cif.gz emd_13427_validation.cif.gz | 18.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13427 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13427 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13427 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13427 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7phrMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13427.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13427.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Fully assembled T-cell receptor engaging a tumor-associated pepti...
+超分子 #1: Fully assembled T-cell receptor engaging a tumor-associated pepti...
+分子 #1: T-cell receptor alpha chain
+分子 #2: T-cell receptor beta chain
+分子 #3: T-cell surface glycoprotein CD3 gamma chain
+分子 #4: T-cell surface glycoprotein CD3 delta chain, green fluorescent protein
+分子 #5: T-cell surface glycoprotein CD3 epsilon chain
+分子 #6: HLA class I histocompatibility antigen, A alpha chain
+分子 #7: Beta-2-microglobulin
+分子 #8: Tumor-associated antigentic peptide gp100
+分子 #9: T-cell surface glycoprotein CD3 zeta chain
+分子 #10: 2-acetamido-2-deoxy-beta-D-glucopyranose
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 61.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー