+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12711 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural basis for VIPP1 oligomerization and maintenance of thylakoid membrane integrity | ||||||||||||||||||||||||
 マップデータ マップデータ | C15 refined map | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
 キーワード キーワード | ESCRT-III / Membrane remodelling / nucleotide hydrolysis / photosynthesis / thylakoid biogenesis / stress response / LIPID BINDING PROTEIN | ||||||||||||||||||||||||
| 機能・相同性 | PspA/IM30 / PspA/IM30 family / plasma membrane / Membrane-associated protein Vipp1 機能・相同性情報 機能・相同性情報 | ||||||||||||||||||||||||
| 生物種 |  | ||||||||||||||||||||||||
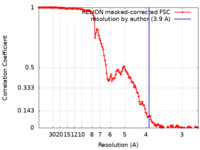
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Gupta TK / Klumpe S | ||||||||||||||||||||||||
| 資金援助 |  ドイツ, ドイツ,  日本, 7件 日本, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2021 ジャーナル: Cell / 年: 2021タイトル: Structural basis for VIPP1 oligomerization and maintenance of thylakoid membrane integrity. 著者: Tilak Kumar Gupta / Sven Klumpe / Karin Gries / Steffen Heinz / Wojciech Wietrzynski / Norikazu Ohnishi / Justus Niemeyer / Benjamin Spaniol / Miroslava Schaffer / Anna Rast / Matthias ...著者: Tilak Kumar Gupta / Sven Klumpe / Karin Gries / Steffen Heinz / Wojciech Wietrzynski / Norikazu Ohnishi / Justus Niemeyer / Benjamin Spaniol / Miroslava Schaffer / Anna Rast / Matthias Ostermeier / Mike Strauss / Jürgen M Plitzko / Wolfgang Baumeister / Till Rudack / Wataru Sakamoto / Jörg Nickelsen / Jan M Schuller / Michael Schroda / Benjamin D Engel /    要旨: Vesicle-inducing protein in plastids 1 (VIPP1) is essential for the biogenesis and maintenance of thylakoid membranes, which transform light into life. However, it is unknown how VIPP1 performs its ...Vesicle-inducing protein in plastids 1 (VIPP1) is essential for the biogenesis and maintenance of thylakoid membranes, which transform light into life. However, it is unknown how VIPP1 performs its vital membrane-remodeling functions. Here, we use cryo-electron microscopy to determine structures of cyanobacterial VIPP1 rings, revealing how VIPP1 monomers flex and interweave to form basket-like assemblies of different symmetries. Three VIPP1 monomers together coordinate a non-canonical nucleotide binding pocket on one end of the ring. Inside the ring's lumen, amphipathic helices from each monomer align to form large hydrophobic columns, enabling VIPP1 to bind and curve membranes. In vivo mutations in these hydrophobic surfaces cause extreme thylakoid swelling under high light, indicating an essential role of VIPP1 lipid binding in resisting stress-induced damage. Using cryo-correlative light and electron microscopy (cryo-CLEM), we observe oligomeric VIPP1 coats encapsulating membrane tubules within the Chlamydomonas chloroplast. Our work provides a structural foundation for understanding how VIPP1 directs thylakoid biogenesis and maintenance. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12711.map.gz emd_12711.map.gz | 140.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12711-v30.xml emd-12711-v30.xml emd-12711.xml emd-12711.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12711_fsc.xml emd_12711_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12711.png emd_12711.png | 50.2 KB | ||
| マスクデータ |  emd_12711_msk_1.map emd_12711_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-12711.cif.gz emd-12711.cif.gz | 6.1 KB | ||
| その他 |  emd_12711_additional_1.map.gz emd_12711_additional_1.map.gz emd_12711_half_map_1.map.gz emd_12711_half_map_1.map.gz emd_12711_half_map_2.map.gz emd_12711_half_map_2.map.gz | 166 MB 140.3 MB 140.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12711 http://ftp.pdbj.org/pub/emdb/structures/EMD-12711 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12711 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12711 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12711_validation.pdf.gz emd_12711_validation.pdf.gz | 1016.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12711_full_validation.pdf.gz emd_12711_full_validation.pdf.gz | 1016.4 KB | 表示 | |
| XML形式データ |  emd_12711_validation.xml.gz emd_12711_validation.xml.gz | 20 KB | 表示 | |
| CIF形式データ |  emd_12711_validation.cif.gz emd_12711_validation.cif.gz | 26.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12711 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12711 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12711 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12711 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12711.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12711.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C15 refined map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12711_msk_1.map emd_12711_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: C15 map before refinement
| ファイル | emd_12711_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C15 map before refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: C15 half map 1
| ファイル | emd_12711_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C15 half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: C15 half map 2
| ファイル | emd_12711_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C15 half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : VIPP1 / IM30 complex
| 全体 | 名称: VIPP1 / IM30 complex |
|---|---|
| 要素 |
|
-超分子 #1: VIPP1 / IM30 complex
| 超分子 | 名称: VIPP1 / IM30 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Protein sll0617
| 分子 | 名称: Protein sll0617 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: PCC 6803 / Kazusa |
| 分子量 | 理論値: 28.822145 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ALFDRLGRVV RANLNDLVSK AEDPEKVLEQ AVIDMQEDLV QLRQAVARTI AEEKRTEQRL NQDTQEAKKW EDRAKLALTN GEENLAREA LARKKSLTDT AAAYQTQLAQ QRTMSENLRR NLAALEAKIS EAKTKKNMLQ ARAKAAKANA ELQQTLGGLG T SSATSAFE ...文字列: ALFDRLGRVV RANLNDLVSK AEDPEKVLEQ AVIDMQEDLV QLRQAVARTI AEEKRTEQRL NQDTQEAKKW EDRAKLALTN GEENLAREA LARKKSLTDT AAAYQTQLAQ QRTMSENLRR NLAALEAKIS EAKTKKNMLQ ARAKAAKANA ELQQTLGGLG T SSATSAFE RMENKVLDME ATSQAAGELA GFGIENQFAQ LEASSGVEDE LAALKASMAG GALPGTSAAT PQLEAAPVDS SV PANNASQ DDAVIDQELD DLRRRLNNL UniProtKB: Membrane-associated protein Vipp1 |
-分子 #2: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Initial model 4whe used for comparative modelling and Rosetta to predict the missing segments |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-7o3x: |
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)