+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10865 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PSII-LHCII C2S2 supercomplex from Pisum sativum grown in high light conditions | |||||||||
 マップデータ マップデータ | PSIILHCII C2S2 supercomplex in high light condition | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cryo-EM / photosystem II / High Light / PHOTOSYNTHESIS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報photosynthesis, light harvesting / photosystem II oxygen evolving complex / photosystem II assembly / oxygen evolving activity / photosystem II stabilization / photosystem II reaction center / photosystem II / oxidoreductase activity, acting on diphenols and related substances as donors, oxygen as acceptor / photosynthetic electron transport chain / photosystem I ...photosynthesis, light harvesting / photosystem II oxygen evolving complex / photosystem II assembly / oxygen evolving activity / photosystem II stabilization / photosystem II reaction center / photosystem II / oxidoreductase activity, acting on diphenols and related substances as donors, oxygen as acceptor / photosynthetic electron transport chain / photosystem I / photosystem II / response to herbicide / chlorophyll binding / photosynthetic electron transport in photosystem II / phosphate ion binding / photosynthesis, light reaction / chloroplast thylakoid membrane / : / photosynthesis / electron transfer activity / protein stabilization / iron ion binding / heme binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Grinzato A / Albanese P | |||||||||
 引用 引用 |  ジャーナル: Int J Mol Sci / 年: 2020 ジャーナル: Int J Mol Sci / 年: 2020タイトル: High-Light versus Low-Light: Effects on Paired Photosystem II Supercomplex Structural Rearrangement in Pea Plants. 著者: Alessandro Grinzato / Pascal Albanese / Roberto Marotta / Paolo Swuec / Guido Saracco / Martino Bolognesi / Giuseppe Zanotti / Cristina Pagliano /  要旨: In plant thylakoid membranes Photosystem II (PSII) associates with a variable number of antenna proteins (LHCII) to form different types of supercomplexes (PSII-LHCII), whose organization is ...In plant thylakoid membranes Photosystem II (PSII) associates with a variable number of antenna proteins (LHCII) to form different types of supercomplexes (PSII-LHCII), whose organization is dynamically adjusted in response to light cues, with the CS more abundant in high-light and the CSM in low-light. Paired PSII-LHCII supercomplexes interacting at their stromal surface from adjacent thylakoid membranes were previously suggested to mediate stacking. Here, we present the cryo-electron microscopy maps of paired CS and CSM supercomplexes isolated from pea plants grown in high-light and low-light, respectively. These maps show a different rotational offset between the two supercomplexes in the pair, responsible for modifying their reciprocal interaction and energetic connectivity. This evidence reveals a different way by which paired PSII-LHCII supercomplexes can mediate stacking at diverse irradiances. Electrostatic stromal interactions between LHCII trimers almost completely overlapping in the paired CS can be the main determinant by which PSII-LHCII supercomplexes mediate stacking in plants grown in high-light, whereas the mutual interaction of stromal N-terminal loops of two facing Lhcb4 subunits in the paired CSM can fulfil this task in plants grown in low-light. The high-light induced accumulation of the Lhcb4.3 protein in PSII-LHCII supercomplexes has been previously reported. Our cryo-electron microscopy map at 3.8 Å resolution of the CS supercomplex isolated from plants grown in high-light suggests the presence of the Lhcb4.3 protein revealing peculiar structural features of this high-light-specific antenna important for photoprotection. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10865.map.gz emd_10865.map.gz | 141.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10865-v30.xml emd-10865-v30.xml emd-10865.xml emd-10865.xml | 43.2 KB 43.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10865.png emd_10865.png | 127.8 KB | ||
| Filedesc metadata |  emd-10865.cif.gz emd-10865.cif.gz | 9.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10865 http://ftp.pdbj.org/pub/emdb/structures/EMD-10865 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10865 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10865 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10865_validation.pdf.gz emd_10865_validation.pdf.gz | 562.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10865_full_validation.pdf.gz emd_10865_full_validation.pdf.gz | 561.9 KB | 表示 | |
| XML形式データ |  emd_10865_validation.xml.gz emd_10865_validation.xml.gz | 7 KB | 表示 | |
| CIF形式データ |  emd_10865_validation.cif.gz emd_10865_validation.cif.gz | 8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10865 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10865 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10865 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10865 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10865.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10865.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PSIILHCII C2S2 supercomplex in high light condition | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
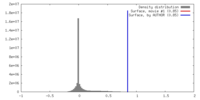
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : PSII-LHCII C2S2 supercomplex
+超分子 #1: PSII-LHCII C2S2 supercomplex
+分子 #1: Chlorophyll a-b binding protein 8, chloroplastic
+分子 #2: Photosystem II protein D1
+分子 #3: Photosystem II CP47 reaction center protein
+分子 #4: Photosystem II CP43 reaction center protein
+分子 #5: Photosystem II D2 protein
+分子 #6: Cytochrome b559 subunit alpha
+分子 #7: Cytochrome b559 subunit beta
+分子 #8: Photosystem II reaction center protein H
+分子 #9: Photosystem II reaction center protein I
+分子 #10: Photosystem II reaction center protein J
+分子 #11: Photosystem II reaction center protein K
+分子 #12: Photosystem II reaction center protein L
+分子 #13: Photosystem II reaction center protein M
+分子 #14: Oxygen-evolving enhancer protein 1, chloroplastic
+分子 #15: Photosystem II reaction center protein T
+分子 #16: Photosystem II reaction center protein W
+分子 #17: Ultraviolet-B-repressible protein
+分子 #18: Photosystem II reaction center protein Z
+分子 #19: Light harvesting chlorophyll a/b-binding protein Lhcb4.3
+分子 #20: Light harvesting chlorophyll a/b-binding protein Lhcb5, CP26
+分子 #21: CHLOROPHYLL B
+分子 #22: CHLOROPHYLL A
+分子 #23: (3R,3'R,6S)-4,5-DIDEHYDRO-5,6-DIHYDRO-BETA,BETA-CAROTENE-3,3'-DIOL
+分子 #24: (3S,5R,6S,3'S,5'R,6'S)-5,6,5',6'-DIEPOXY-5,6,5',6'- TETRAHYDRO-BE...
+分子 #25: (1R,3R)-6-{(3E,5E,7E,9E,11E,13E,15E,17E)-18-[(1S,4R,6R)-4-HYDROXY...
+分子 #26: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE
+分子 #27: CA-MN4-O5 CLUSTER
+分子 #28: FE (II) ION
+分子 #29: CHLORIDE ION
+分子 #30: PHEOPHYTIN A
+分子 #31: BETA-CAROTENE
+分子 #32: 2,3-DIMETHYL-5-(3,7,11,15,19,23,27,31,35-NONAMETHYL-2,6,10,14,18,...
+分子 #33: 1,2-DI-O-ACYL-3-O-[6-DEOXY-6-SULFO-ALPHA-D-GLUCOPYRANOSYL]-SN-GLYCEROL
+分子 #34: BICARBONATE ION
+分子 #35: DIGALACTOSYL DIACYL GLYCEROL (DGDG)
+分子 #36: 1,2-DISTEAROYL-MONOGALACTOSYL-DIGLYCERIDE
+分子 #37: PROTOPORPHYRIN IX CONTAINING FE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K2 BASE (4k x 4k) #0 - 平均電子線量: 40.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K2 BASE (4k x 4k) #1 - 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)