+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10800 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Duck hepatitis B virus capsid | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | duck hepatitis B core protein / extension domain / spike / slowly folding / VIRUS LIKE PARTICLE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報microtubule-dependent intracellular transport of viral material towards nucleus / T=4 icosahedral viral capsid / viral penetration into host nucleus / host cell / host cell cytoplasm / symbiont entry into host cell / structural molecule activity / DNA binding / RNA binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Hepatitis B virus duck/DHBV-16 (ウイルス) Hepatitis B virus duck/DHBV-16 (ウイルス) | ||||||||||||
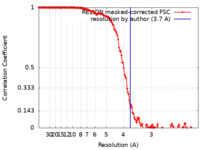
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | ||||||||||||
 データ登録者 データ登録者 | Makbul C / Bottcher B | ||||||||||||
| 資金援助 |  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Slowly folding surface extension in the prototypic avian hepatitis B virus capsid governs stability. 著者: Cihan Makbul / Michael Nassal / Bettina Böttcher /  要旨: Hepatitis B virus (HBV) is an important but difficult to study human pathogen. Most basics of the hepadnaviral life-cycle were unraveled using duck HBV (DHBV) as a model although DHBV has a capsid ...Hepatitis B virus (HBV) is an important but difficult to study human pathogen. Most basics of the hepadnaviral life-cycle were unraveled using duck HBV (DHBV) as a model although DHBV has a capsid protein (CP) comprising ~260 rather than ~180 amino acids. Here we present high-resolution structures of several DHBV capsid-like particles (CLPs) determined by electron cryo-microscopy. As for HBV, DHBV CLPs consist of a dimeric α-helical frame-work with protruding spikes at the dimer interface. A fundamental new feature is a ~ 45 amino acid proline-rich extension in each monomer replacing the tip of the spikes in HBV CP. In vitro, folding of the extension takes months, implying a catalyzed process in vivo. DHBc variants lacking a folding-proficient extension produced regular CLPs in bacteria but failed to form stable nucleocapsids in hepatoma cells. We propose that the extension domain acts as a conformational switch with differential response options during viral infection. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10800.map.gz emd_10800.map.gz | 226.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10800-v30.xml emd-10800-v30.xml emd-10800.xml emd-10800.xml | 19.9 KB 19.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10800_fsc.xml emd_10800_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10800.png emd_10800.png | 364 KB | ||
| マスクデータ |  emd_10800_msk_1.map emd_10800_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-10800.cif.gz emd-10800.cif.gz | 6.4 KB | ||
| その他 |  emd_10800_half_map_1.map.gz emd_10800_half_map_1.map.gz emd_10800_half_map_2.map.gz emd_10800_half_map_2.map.gz | 222.9 MB 222.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10800 http://ftp.pdbj.org/pub/emdb/structures/EMD-10800 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10800 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10800 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10800_validation.pdf.gz emd_10800_validation.pdf.gz | 439 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10800_full_validation.pdf.gz emd_10800_full_validation.pdf.gz | 438.1 KB | 表示 | |
| XML形式データ |  emd_10800_validation.xml.gz emd_10800_validation.xml.gz | 19.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10800 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10800 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10800 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10800 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10800.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10800.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0635 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10800_msk_1.map emd_10800_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10800_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10800_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hepatitis B virus duck/DHBV-16
| 全体 | 名称:  Hepatitis B virus duck/DHBV-16 (ウイルス) Hepatitis B virus duck/DHBV-16 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Hepatitis B virus duck/DHBV-16
| 超分子 | 名称: Hepatitis B virus duck/DHBV-16 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 489543 / 生物種: Hepatitis B virus duck/DHBV-16 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
| 分子量 | 理論値: 7.2 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: duck Hepatitis B virus capsid / 直径: 370.0 Å / T番号(三角分割数): 4 |
-分子 #1: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Hepatitis B virus duck/DHBV-16 (ウイルス) Hepatitis B virus duck/DHBV-16 (ウイルス) |
| 分子量 | 理論値: 30.33851 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDINASRALA NVYDLPDDFF PKIDDLVRDA KDALEPYWKS DSIKKHVLIA THFVDLIEDF WQTTQGMHEI AESLRAVIPP TTTPVPPGY LIQHEEAEEI PLGDLFKHQE ERIVSFQPDY PITARIHAHL KAYAKINEES LDRARRLLWW HYNCLLWGEA Q VTNYISRL ...文字列: MDINASRALA NVYDLPDDFF PKIDDLVRDA KDALEPYWKS DSIKKHVLIA THFVDLIEDF WQTTQGMHEI AESLRAVIPP TTTPVPPGY LIQHEEAEEI PLGDLFKHQE ERIVSFQPDY PITARIHAHL KAYAKINEES LDRARRLLWW HYNCLLWGEA Q VTNYISRL RTWLSTPEKY RGRDAPTIEA ITRPIQVAQG GRKTTTGTRK PRGLEPRRRK VKTTVVYGRR RSKSRERRAP TP QRAGSPL PRSSSSHHRS PSPRK UniProtKB: Capsid protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.2 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.029 kPa | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: For the vitrification, grids (400 mesh copper grids (type R 1.2/1.3. Quantifoil Micro Tools, Jena/Germany) were rendered hydrophilic by glow discharging in air at a pressure of 29 Pa for 2 ...詳細: For the vitrification, grids (400 mesh copper grids (type R 1.2/1.3. Quantifoil Micro Tools, Jena/Germany) were rendered hydrophilic by glow discharging in air at a pressure of 29 Pa for 2 minutes at medium power with a Plasma Cleaner (model PDC-002. Harrick Plasma Ithaca, NY/USA). Then, 3.5 ul of DHBc solution was pipetted onto the grids and they were plunge frozen in liquid ethane with a Vitrobot mark IV (FEI-Thermo Fisher Scientific). The settings for the Vitrobot were 3s blot time, 45 s wait time, blot force 0 at a temperature of 4 C and 100 % humidity. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 2473 / 平均露光時間: 75.0 sec. / 平均電子線量: 77.0 e/Å2 詳細: movie mode, 3 images per hole, 47 fractions per movie |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 2.1 µm / 最小 デフォーカス(補正後): 0.5 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6ygh: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)