+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0990 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Dimethylformamidase, tetramer, Y440A mutant | ||||||||||||
 マップデータ マップデータ | One of the primary map from the refinement in Relion | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | ab polypeptide / mononuclear iron / amidohydrolase / tetramer / HYDROLASE | ||||||||||||
| 機能・相同性 | N,N-dimethylformamidase / N,N-dimethylformamidase activity / N,N-dimethylformamidase beta subunit-like, C-terminal / N,N-dimethylformamidase beta subunit-like, C-terminal / Concanavalin A-like lectin/glucanases superfamily / Concanavalin A-like lectin/glucanase domain superfamily / metal ion binding / N,N-dimethylformamidase large subunit / N,N-dimethylformamidase small subunit 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Paracoccus sp. SSG05 (バクテリア) Paracoccus sp. SSG05 (バクテリア) | ||||||||||||
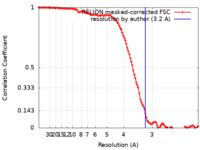
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | ||||||||||||
 データ登録者 データ登録者 | Arya CA / Yadav S | ||||||||||||
| 資金援助 |  インド, 3件 インド, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Angew Chem Int Ed Engl / 年: 2020 ジャーナル: Angew Chem Int Ed Engl / 年: 2020タイトル: A 2-Tyr-1-carboxylate Mononuclear Iron Center Forms the Active Site of a Paracoccus Dimethylformamidase. 著者: Chetan Kumar Arya / Swati Yadav / Jonathan Fine / Ana Casanal / Gaurav Chopra / Gurunath Ramanathan / Kutti R Vinothkumar / Ramaswamy Subramanian /    要旨: N,N-dimethyl formamide (DMF) is an extensively used organic solvent but is also a potent pollutant. Certain bacterial species from genera such as Paracoccus, Pseudomonas, and Alcaligenes have evolved ...N,N-dimethyl formamide (DMF) is an extensively used organic solvent but is also a potent pollutant. Certain bacterial species from genera such as Paracoccus, Pseudomonas, and Alcaligenes have evolved to use DMF as a sole carbon and nitrogen source for growth via degradation by a dimethylformamidase (DMFase). We show that DMFase from Paracoccus sp. strain DMF is a halophilic and thermostable enzyme comprising a multimeric complex of the α β or (α β ) type. One of the three domains of the large subunit and the small subunit are hitherto undescribed protein folds of unknown evolutionary origin. The active site consists of a mononuclear iron coordinated by two Tyr side-chain phenolates and one carboxylate from Glu. The Fe ion in the active site catalyzes the hydrolytic cleavage of the amide bond in DMF. Kinetic characterization reveals that the enzyme shows cooperativity between subunits, and mutagenesis and structural data provide clues to the catalytic mechanism. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0990.map.gz emd_0990.map.gz | 116.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0990-v30.xml emd-0990-v30.xml emd-0990.xml emd-0990.xml | 21.8 KB 21.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0990_fsc.xml emd_0990_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0990.png emd_0990.png | 49.2 KB | ||
| Filedesc metadata |  emd-0990.cif.gz emd-0990.cif.gz | 6.9 KB | ||
| その他 |  emd_0990_half_map_1.map.gz emd_0990_half_map_1.map.gz emd_0990_half_map_2.map.gz emd_0990_half_map_2.map.gz | 97 MB 97 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0990 http://ftp.pdbj.org/pub/emdb/structures/EMD-0990 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0990 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0990 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0990_validation.pdf.gz emd_0990_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0990_full_validation.pdf.gz emd_0990_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_0990_validation.xml.gz emd_0990_validation.xml.gz | 18.7 KB | 表示 | |
| CIF形式データ |  emd_0990_validation.cif.gz emd_0990_validation.cif.gz | 24.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0990 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0990 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0990 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0990 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0990.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0990.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | One of the primary map from the refinement in Relion | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
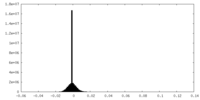
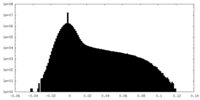
-ハーフマップ: One of the half map from the refinement in Relion
| ファイル | emd_0990_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | One of the half map from the refinement in Relion | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
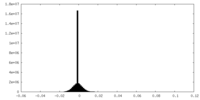
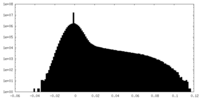
-ハーフマップ: One of the half map from the refinement in Relion
| ファイル | emd_0990_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | One of the half map from the refinement in Relion | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dimethylformamidase
| 全体 | 名称: Dimethylformamidase |
|---|---|
| 要素 |
|
-超分子 #1: Dimethylformamidase
| 超分子 | 名称: Dimethylformamidase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Tetramer, 2x(a2b2) |
|---|---|
| 由来(天然) | 生物種:  Paracoccus sp. SSG05 (バクテリア) Paracoccus sp. SSG05 (バクテリア) |
| 分子量 | 理論値: 400 KDa |
-分子 #1: N,N-dimethylformamidase large subunit
| 分子 | 名称: N,N-dimethylformamidase large subunit / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: N,N-dimethylformamidase |
|---|---|
| 由来(天然) | 生物種:  Paracoccus sp. SSG05 (バクテリア) Paracoccus sp. SSG05 (バクテリア) |
| 分子量 | 理論値: 86.249664 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKDIAIRGYC DRPSVATGET IRFYVSANET RGTFDAELVR LIHGDSNPAG PGYKEEAIKS DLEGQYPARF QRTQFGSYVE VADPDAGLQ PDGAFSVHLF LWSTTPSRGR QGIASRWNDE RQSGWNLAIE DGRVVFTIGD GSGATSSVVS DRPLFQQIWY S ITGVYDPE ...文字列: MKDIAIRGYC DRPSVATGET IRFYVSANET RGTFDAELVR LIHGDSNPAG PGYKEEAIKS DLEGQYPARF QRTQFGSYVE VADPDAGLQ PDGAFSVHLF LWSTTPSRGR QGIASRWNDE RQSGWNLAIE DGRVVFTIGD GSGATSSVVS DRPLFQQIWY S ITGVYDPE KKQLRLYQKS VVNRTNSRFG LVVPLDSDCA VSADATVKAA DSETSLLIAG LGEAAAQDGR TWCIAHYNGK VD APKIYGC ALGQDDAEKL SRGEIVRPIS RLAHWDFSAG IGLNGIPTDH VVDASGYGHH GRCMNQPSRG STGWNWDGHE ENF IHCPEQ YGALWFHEDC LDDCRWEKDF EFTVPEGLKS DFYAVKIRYE DTEDYIPFFV LPPRGTATAP ILVIASTLSY LAYA NEQIM HKADIGQAVA GHTPVLNEND VELHKNLSYY GLSTADGHID GRGVQYTSWR RPIMNLRPKH RQGFGSIWEL PADLH LIDW LNHNGFEYDV ATEHDLNDQG AELLRRYKVV LTGSHPEYQT WANADAWEDY LADGGRGMYL AANGMYWIVE VHPEKP WVM EVRKELGVTA WEAPPGEYHY STNGRRGGRF RGRARATQKI WGTGMSSFGF DHSGYFVQMP DSQDERVAWI MEGIDPE ER IGDGGLVGGG AGGYELDRYD LALGTPPNTL LLASSVEHSV VYTVIPDDKA FPHPGMNGGE HPFVRADITY FSTANGGG M FATSSISWLG SLSWNDYDNN VSKMTKNVLN QFIKDEPAPR VKLAAALEHH HHHH UniProtKB: N,N-dimethylformamidase large subunit |
-分子 #2: N,N-dimethylformamidase small subunit
| 分子 | 名称: N,N-dimethylformamidase small subunit / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO / EC番号: N,N-dimethylformamidase |
|---|---|
| 由来(天然) | 生物種:  Paracoccus sp. SSG05 (バクテリア) Paracoccus sp. SSG05 (バクテリア) |
| 分子量 | 理論値: 16.083823 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTEASESCVR DPSNYRDRSA DWYAFYDERR RKEIIDIIDE HPEIVEEHAA NPFGYRKHPS PYLQRVHNYF RMQPTFGRYY IYSEREWDA YRIATIREFG ELPELGDERF KTEEEAMHAV FLRRIEDVRA ELA UniProtKB: N,N-dimethylformamidase small subunit |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.2 / 構成要素:
| ||||||
| グリッド | モデル: Quantifoil R0.6/1 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. / 前処理 - 雰囲気: AIR 詳細: Glow discharge was performed with Quorum glocube at 25mA, for 90 s. | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291.15 K / 装置: FEI VITROBOT MARK IV 詳細: Blot force was 0 and blotting was done for 3.5 seconds. | ||||||
| 詳細 | Sample at higher salt exists mostly as tetramer |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 77.7 K / 最高: 77.7 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 443 / 平均露光時間: 60.0 sec. / 平均電子線量: 27.5 e/Å2 詳細: A total of 25 frames were saved from the 60 second exposure, resulting in ~1.1 electron/frame |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 4.5314 µm / 最小 デフォーカス(補正後): 1.2165 µm / 倍率(補正後): 130841 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.5314 µm / 最小 デフォーカス(公称値): 1.2165 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: BACKBONE TRACE / 温度因子: 52.5 |
|---|---|
| 得られたモデル |  PDB-6lvd: |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X