+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0126 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the consensus hub of the clathrin coat complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | clathrin / coat protein / endocytosis / trafficking / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報clathrin coat of trans-Golgi network vesicle / clathrin light chain binding / clathrin complex / clathrin coat of coated pit / clathrin coat assembly / clathrin-coated endocytic vesicle / clathrin-coated vesicle / receptor-mediated endocytosis / intracellular protein transport / spindle ...clathrin coat of trans-Golgi network vesicle / clathrin light chain binding / clathrin complex / clathrin coat of coated pit / clathrin coat assembly / clathrin-coated endocytic vesicle / clathrin-coated vesicle / receptor-mediated endocytosis / intracellular protein transport / spindle / disordered domain specific binding / mitotic cell cycle / nucleolus / structural molecule activity / extracellular exosome / nucleoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
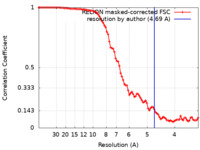
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.69 Å | |||||||||
 データ登録者 データ登録者 | Morris KL / Smith CJ | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2019 ジャーナル: Nat Struct Mol Biol / 年: 2019タイトル: Cryo-EM of multiple cage architectures reveals a universal mode of clathrin self-assembly. 著者: Kyle L Morris / Joseph R Jones / Mary Halebian / Shenping Wu / Michael Baker / Jean-Paul Armache / Amaurys Avila Ibarra / Richard B Sessions / Alexander D Cameron / Yifan Cheng / Corinne J Smith /   要旨: Clathrin forms diverse lattice and cage structures that change size and shape rapidly in response to the needs of eukaryotic cells during clathrin-mediated endocytosis and intracellular trafficking. ...Clathrin forms diverse lattice and cage structures that change size and shape rapidly in response to the needs of eukaryotic cells during clathrin-mediated endocytosis and intracellular trafficking. We present the cryo-EM structure and molecular model of assembled porcine clathrin, providing insights into interactions that stabilize key elements of the clathrin lattice, namely, between adjacent heavy chains, at the light chain-heavy chain interface and within the trimerization domain. Furthermore, we report cryo-EM maps for five different clathrin cage architectures. Fitting structural models to three of these maps shows that their assembly requires only a limited range of triskelion leg conformations, yet inherent flexibility is required to maintain contacts. Analysis of the protein-protein interfaces shows remarkable conservation of contact sites despite architectural variation. These data reveal a universal mode of clathrin assembly that allows variable cage architecture and adaptation of coated vesicle size and shape during clathrin-mediated vesicular trafficking or endocytosis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0126.map.gz emd_0126.map.gz | 11.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0126-v30.xml emd-0126-v30.xml emd-0126.xml emd-0126.xml | 24.8 KB 24.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0126_fsc.xml emd_0126_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0126.png emd_0126.png | 102.8 KB | ||
| マスクデータ |  emd_0126_msk_1.map emd_0126_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-0126.cif.gz emd-0126.cif.gz | 6.7 KB | ||
| その他 |  emd_0126_additional_1.map.gz emd_0126_additional_1.map.gz emd_0126_additional_2.map.gz emd_0126_additional_2.map.gz emd_0126_half_map_1.map.gz emd_0126_half_map_1.map.gz emd_0126_half_map_2.map.gz emd_0126_half_map_2.map.gz | 60 MB 9 MB 14.8 MB 14.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0126 http://ftp.pdbj.org/pub/emdb/structures/EMD-0126 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0126 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0126 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0126_validation.pdf.gz emd_0126_validation.pdf.gz | 604.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0126_full_validation.pdf.gz emd_0126_full_validation.pdf.gz | 604.1 KB | 表示 | |
| XML形式データ |  emd_0126_validation.xml.gz emd_0126_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_0126_validation.cif.gz emd_0126_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0126 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0126 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0126 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0126 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6sctMC  0114C  0115C  0116C  0118C  0120C  0121C  0122C  0123C  0124C  0125C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 電子顕微鏡画像生データ |  EMPIAR-10296 (タイトル: Single particle cryo-EM dataset of the triskelion hub subparticle extraction from clathrin cages EMPIAR-10296 (タイトル: Single particle cryo-EM dataset of the triskelion hub subparticle extraction from clathrin cagesData size: 88.4 Data #1: Hub subparticles of the 28 mini coat [picked particles - multiframe - unprocessed] Data #2: Hub subparticles of the 32 sweet potato [picked particles - multiframe - unprocessed] Data #3: Hub subparticles of the 36 D6 barrel [picked particles - multiframe - unprocessed] Data #4: Hub subparticles of the 36 tennis ball [picked particles - multiframe - unprocessed] Data #5: Hub subparticles of the 37 big apple [picked particles - multiframe - unprocessed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0126.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0126.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.705 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
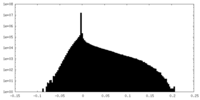
| ファイル |  emd_0126_msk_1.map emd_0126_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
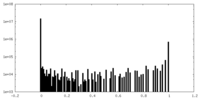
| 投影像・断面図 |
| ||||||||||||
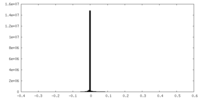
| 密度ヒストグラム |
-追加マップ: Additional map used during model building at ad-hoc...
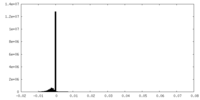
| ファイル | emd_0126_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Additional map used during model building at ad-hoc sharpened b-factor of -250. | ||||||||||||
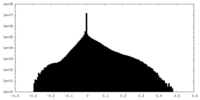
| 投影像・断面図 |
| ||||||||||||
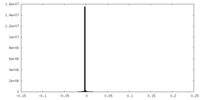
| 密度ヒストグラム |
-追加マップ: Post processed map from resolution measurement.
| ファイル | emd_0126_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post processed map from resolution measurement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Soft spherical masked (diameter 320A) applied to remove...
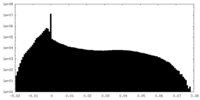
| ファイル | emd_0126_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Soft spherical masked (diameter 320A) applied to remove artifactual density prior to resolution measurement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Soft spherical masked (diameter 320A) applied to remove...
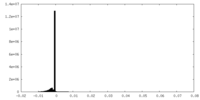
| ファイル | emd_0126_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Soft spherical masked (diameter 320A) applied to remove artifactual density prior to resolution measurement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Assembly of clathrin heavy and light chain into coat complexes
| 全体 | 名称: Assembly of clathrin heavy and light chain into coat complexes |
|---|---|
| 要素 |
|
-超分子 #1: Assembly of clathrin heavy and light chain into coat complexes
| 超分子 | 名称: Assembly of clathrin heavy and light chain into coat complexes タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 540 KDa |
-分子 #1: clathrin heavy chain
| 分子 | 名称: clathrin heavy chain / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: MAQILPIRFQ EHLQLQNLGI NPANIGFSTL TMESDKFICI REKVGEQAQV VIIDMNDPSN PIRRPISAD SAIMNPASKV IALKAGKTLQ IFNIEMKSKM KAHTMTDDVT FWKWISLNTV A LVTDNAVY HWSMEGESQP VKMFDRHSSL AGCQIINYRT DAKQKWLLLT ...文字列: MAQILPIRFQ EHLQLQNLGI NPANIGFSTL TMESDKFICI REKVGEQAQV VIIDMNDPSN PIRRPISAD SAIMNPASKV IALKAGKTLQ IFNIEMKSKM KAHTMTDDVT FWKWISLNTV A LVTDNAVY HWSMEGESQP VKMFDRHSSL AGCQIINYRT DAKQKWLLLT GISAQQNRVV GA MQLYSVD RKVSQPIEGH AASFAQFKME GNAEESTLFC FAVRGQAGGK LHIIEVGTPP TGN QPFPKK AVDVFFPPEA QNDFPVAMQI SEKHDVVFLI TKYGYIHLYD LETGTCIYMN RISG ETIFV TAPHEATAGI IGVNRKGQVL SVCVEEENII PYITNVLQNP DLALRMAVRN NLAGA EELF ARKFNALFAQ GNYSEAAKVA ANAPKGILRT PDTIRRFQSV PAQPGQTSPL LQYFGI LLD QGQLNKYESL ELCRPVLQQG RKQLLEKWLK EDKLECSEEL GDLVKSVDPT LALSVYL RA NVPNKVIQCF AETGQVQKIV LYAKKVGYTP DWIFLLRNVM RISPDQGQQF AQMLVQDE E PLADITQIVD VFMEYNLIQQ CTAFLLDALK NNRPSEGPLQ TRLLEMNLMH APQVADAIL GNQMFTHYDR AHIAQLCEKA GLLQRALEHF TDLYDIKRAV VHTHLLNPEW LVNYFGSLSV EDSLECLRA MLSANIRQNL QICVQVASKY HEQLSTQSLI ELFESFKSFE GLFYFLGSIV N FSQDPDVH FKYIQAACKT GQIKEVERIC RESNCYDPER VKNFLKEAKL TDQLPLIIVC DR FDFVHDL VLYLYRNNLQ KYIEIYVQKV NPSRLPVVIG GLLDVDCSED VIKNLILVVR GQF STDELV AEVEKRNRLK LLLPWLEARI HEGCEEPATH NALAKIYIDS NNNPERFLRE NPYY DSRVV GKYCEKRDPH LACVAYERGQ CDLELINVCN ENSLFKSLSR YLVRRKDPEL WGSVL LESN PYRRPLIDQV VQTALSETQD PEEVSVTVKA FMTADLPNEL IELLEKIVLD NSVFSE HRN LQNLLILTAI KADRTRVMEY INRLDNYDAP DIANIAISNE LFEEAFAIFR KFDVNTS AV QVLIEHIGNL DRAYEFAERC NEPAVWSQLA KAQLQKGMVK EAIDSYIKAD DPSSYMEV V QAANTSGNWE ELVKYLQMAR KKARESYVET ELIFALAKTN RLAELEEFIN GPNNAHIQQ VGDRCYDEKM YDAAKLLYNN VSNFGRLAST LVHLGEYQAA VDGARKANST RTWKEVCFAC VDGKEFRLA QMCGLHIVVH ADELEELINY YQDRGYFEEL ITMLEAALGL ERAHMGMFTE L AILYSKFK PQKMREHLEL FWSRVNIPKV LRAAEQAHLW AELVFLYDKY EEYDNAIITM MN HPTDAWK EGQFKDIITK VANVELYYRA IQFYLEFKPL LLNDLLMVLS PRLDHTRAVN YFS KVKQLP LVKPYLRSVQ NHNNKSVNES LNNLFITEED YQALRTSIDA YDNFDNISLA QRLE KHELI EFRRIAAYLF KGNNRWKQSV ELCKKDSLYK DAMQYASESK DTELAEELLQ WFLQE EKRE CFGACLFTCY DLLRPDVVLE TAWRHNIMDF AMPYFIQVMK EYLTKVDKLD ASESLR KEE EQATETQPIV YGQPQLMLTA GPSVAVPPQA PFGYGYTAPP YGQPQPGFGY SM UniProtKB: Clathrin heavy chain |
-分子 #2: clathrin light chain b
| 分子 | 名称: clathrin light chain b / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: MADDFGFFSS SESGAPEVAE EDPAAAFLAQ QESEIAGIEN DEGFGAPAGS QAALAQPGPA SGAGPEDMG TTVNGDVFQD ANGPADGYAA IAQADRLTQE PESIRKWREE QRKRLQELDA A SKVTEQEW REKAKKDLEE WNQRQSEQVE KNKINNRIAD KAFYQQPDAD ...文字列: MADDFGFFSS SESGAPEVAE EDPAAAFLAQ QESEIAGIEN DEGFGAPAGS QAALAQPGPA SGAGPEDMG TTVNGDVFQD ANGPADGYAA IAQADRLTQE PESIRKWREE QRKRLQELDA A SKVTEQEW REKAKKDLEE WNQRQSEQVE KNKINNRIAD KAFYQQPDAD IIGYVASEEA FV KESKEET PGTEWEKVAQ LCDFNPKSSK QCKDVSRLRS VLMSLKQTPL SR UniProtKB: Nucleolar protein 16 |
-分子 #3: clathrin light chain a
| 分子 | 名称: clathrin light chain a / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: MADLDPFGAP AGPSLGNGVA GEEDPAAAFL AQQESEIAGI ENDEAFAILD GGAPGPQPHG EPPGGPDAV DGVMNGEYYQ ESNGPTDSYA AISQVDRLQS EPESIRKWRE EQTERLEALD A NSRKQEAE WKEKAIKELE EWYARQDEQL QKTKANNRVA DEAFYKQPFA ...文字列: MADLDPFGAP AGPSLGNGVA GEEDPAAAFL AQQESEIAGI ENDEAFAILD GGAPGPQPHG EPPGGPDAV DGVMNGEYYQ ESNGPTDSYA AISQVDRLQS EPESIRKWRE EQTERLEALD A NSRKQEAE WKEKAIKELE EWYARQDEQL QKTKANNRVA DEAFYKQPFA DVIGYVAAEE AF VNDIEES SPGTEWERVA RLCDFNPKSS KQAKDVSRMR SVLISLKQAP LVH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6.32 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.4 構成要素:
| |||||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / 装置: HOMEMADE PLUNGER 詳細: 3 uL applied to a grid at room temperature and humidity. 3 second hand blot and plunge.. | |||||||||||||||
| 詳細 | Clathrin coat complexes, end point assembly exhibiting architectural heterogeneity |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 平均露光時間: 3.0 sec. / 平均電子線量: 53.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 82111 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.2 µm 最小 デフォーカス(公称値): 1.4000000000000001 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT 当てはまり具合の基準: Cross-correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6sct: |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X