+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7000 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram average of microtubule-bound dynein-dynactin-BICD2N complex | |||||||||
 マップデータ マップデータ | Microtubule-bound dynein-dynactin-BICD2 complex | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 38.0 Å | |||||||||
 データ登録者 データ登録者 | Grotjahn DA / Chowdhury S / Xu Y / McKenney RJ / Schroer TA / Lander GC | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2018 ジャーナル: Nat Struct Mol Biol / 年: 2018タイトル: Cryo-electron tomography reveals that dynactin recruits a team of dyneins for processive motility. 著者: Danielle A Grotjahn / Saikat Chowdhury / Yiru Xu / Richard J McKenney / Trina A Schroer / Gabriel C Lander /  要旨: Cytoplasmic dynein is a protein complex that transports molecular cargo along microtubules (MTs), playing a key role in the intracellular trafficking network. Vertebrate dynein's movement becomes ...Cytoplasmic dynein is a protein complex that transports molecular cargo along microtubules (MTs), playing a key role in the intracellular trafficking network. Vertebrate dynein's movement becomes strikingly enhanced upon interacting with dynactin and a cargo adaptor such as BicaudalD2. However, the mechanisms responsible for increased transport activity are not well understood, largely owing to limited structural information. We used cryo-electron tomography (cryo-ET) to visualize the 3D structure of the MT-bound dynein-dynactin complex from Mus musculus and show that the dynactin-cargo adaptor complex binds two dimeric dyneins. This configuration imposes spatial and conformational constraints on both dynein dimers, positioning the four motor domains in proximity to one another and oriented toward the MT minus end. We propose that grouping multiple dyneins onto a single dynactin scaffold promotes collective force production, increased processivity, and unidirectional movement, suggesting mechanistic parallels to axonemal dynein. These findings provide structural insights into a previously unknown mechanism for dynein regulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7000.map.gz emd_7000.map.gz | 2.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7000-v30.xml emd-7000-v30.xml emd-7000.xml emd-7000.xml | 19.3 KB 19.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7000.png emd_7000.png | 94.7 KB | ||
| その他 |  emd_7000_additional_1.map.gz emd_7000_additional_1.map.gz emd_7000_additional_2.map.gz emd_7000_additional_2.map.gz emd_7000_additional_3.map.gz emd_7000_additional_3.map.gz | 2.4 MB 2.3 MB 2.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7000 http://ftp.pdbj.org/pub/emdb/structures/EMD-7000 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7000 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7000 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7000_validation.pdf.gz emd_7000_validation.pdf.gz | 79.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7000_full_validation.pdf.gz emd_7000_full_validation.pdf.gz | 78.3 KB | 表示 | |
| XML形式データ |  emd_7000_validation.xml.gz emd_7000_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7000 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7000 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7000 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7000 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7001C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10520 (タイトル: Cryo-electron tomography reveals that dynactin recruits a team of dyneins for processive motility. EMPIAR-10520 (タイトル: Cryo-electron tomography reveals that dynactin recruits a team of dyneins for processive motility.Data size: 200.1 / Data #1: tilt series in mrc format [tilt series]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7000.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7000.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Microtubule-bound dynein-dynactin-BICD2 complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 8.52 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
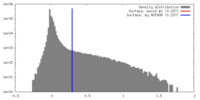
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: focused reconstruction of dynein dimer-2 motor domains
| ファイル | emd_7000_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | focused reconstruction of dynein dimer-2 motor domains | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
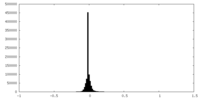
| 密度ヒストグラム |
-追加マップ: focused reconstruction of dynein tails associated with dynactin-BICD2N...
| ファイル | emd_7000_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | focused reconstruction of dynein tails associated with dynactin-BICD2N | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
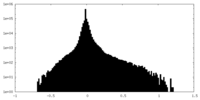
| 密度ヒストグラム |
-追加マップ: focused reconstruction of dynein dimer-1 motor domains
| ファイル | emd_7000_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | focused reconstruction of dynein dimer-1 motor domains | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
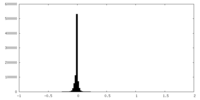
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : microtubule-bound dynein-dynactin-BICD2N complex
| 全体 | 名称: microtubule-bound dynein-dynactin-BICD2N complex |
|---|---|
| 要素 |
|
-超分子 #1: microtubule-bound dynein-dynactin-BICD2N complex
| 超分子 | 名称: microtubule-bound dynein-dynactin-BICD2N complex / タイプ: complex / ID: 1 / 親要素: 0 詳細: GFP-BICD2N fragment recombinantly expressed and purified. Microtubule-bound dynein-dynactin complexes isolated from mouse brain tissue. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 4.2 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 詳細: PMEE buffer (35 mM PIPES, 5 mM MgSO4, 1 mM EGTA, 0.5 mM EDTA, 1 mM GTP, 4 mM AMPPNP, 20 uM Taxol) |
|---|---|
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / チャンバー内温度: 277.15 K / 装置: HOMEMADE PLUNGER 詳細: 4 uL of sample was applied to one side of a plasma-cleaned grid, and the grid was manually blotted on the opposite side for 5-7 seconds immediately before vitrification.. |
| 詳細 | Microtubule-bound dynein-dynactin-BICD2N complexes isolated from mouse brain tissue in the presence of non-hydrolyzable ATP analog AMPPNP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 98.15 K / 最高: 98.15 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-15 / 撮影したグリッド数: 1 / 平均露光時間: 1.16 sec. / 平均電子線量: 0.95 e/Å2 詳細: Images were collected in movie mode with a per-frame exposure time of 80 ms. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 14000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 8.0 µm / 最小 デフォーカス(公称値): 6.0 µm / 倍率(公称値): 14000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Movie frames per tilt were aligned using MotionCorr. |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 38.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: RELION (ver. 1.4) 詳細: Initial angular assignments were obtained by manually docking available structures/reconstructions into extracted subvolumes using UCSF Chimera. These were further refined using the RELION 1. ...詳細: Initial angular assignments were obtained by manually docking available structures/reconstructions into extracted subvolumes using UCSF Chimera. These were further refined using the RELION 1.4 subtomogram averaging auto-refinement program, followed by focused refinement of individual subregions of the complex by application of binary masks and continuation of refinement. The resulting focused maps were stitched together using the "vop maximum" function in UCSF Chimera. 使用したサブトモグラム数: 502 |
| 抽出 | トモグラム数: 127 / 使用した粒子像数: 502 / 手法: manual / ソフトウェア - 名称: EMAN2 (ver. 2.11) / ソフトウェア - 詳細: e2spt_boxer 詳細: Subvolumes were picked using the EMAN2 manual picker for subvolumes. |
| 最終 角度割当 | タイプ: NOT APPLICABLE / ソフトウェア - 名称: RELION (ver. 1.4) / ソフトウェア - 詳細: Subtomogram Averaging |
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)