+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle reconstruction of the human LINE-1 ORF2p without substrate (apo) | |||||||||
 マップデータ マップデータ | Main map of apo ORF2p core | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | reverse transcriptase / LINE-1 / RNA BINDING PROTEIN | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
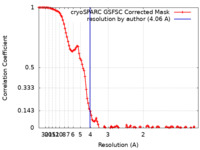
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.06 Å | |||||||||
 データ登録者 データ登録者 | van Eeuwen T / Taylor MS / Rout MP | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Structures, functions and adaptations of the human LINE-1 ORF2 protein. 著者: Eric T Baldwin / Trevor van Eeuwen / David Hoyos / Arthur Zalevsky / Egor P Tchesnokov / Roberto Sánchez / Bryant D Miller / Luciano H Di Stefano / Francesc Xavier Ruiz / Matthew Hancock / ...著者: Eric T Baldwin / Trevor van Eeuwen / David Hoyos / Arthur Zalevsky / Egor P Tchesnokov / Roberto Sánchez / Bryant D Miller / Luciano H Di Stefano / Francesc Xavier Ruiz / Matthew Hancock / Esin Işik / Carlos Mendez-Dorantes / Thomas Walpole / Charles Nichols / Paul Wan / Kirsi Riento / Rowan Halls-Kass / Martin Augustin / Alfred Lammens / Anja Jestel / Paula Upla / Kera Xibinaku / Samantha Congreve / Maximiliaan Hennink / Kacper B Rogala / Anna M Schneider / Jennifer E Fairman / Shawn M Christensen / Brian Desrosiers / Gregory S Bisacchi / Oliver L Saunders / Nafeeza Hafeez / Wenyan Miao / Rosana Kapeller / Dennis M Zaller / Andrej Sali / Oliver Weichenrieder / Kathleen H Burns / Matthias Götte / Michael P Rout / Eddy Arnold / Benjamin D Greenbaum / Donna L Romero / John LaCava / Martin S Taylor /      要旨: The LINE-1 (L1) retrotransposon is an ancient genetic parasite that has written around one-third of the human genome through a 'copy and paste' mechanism catalysed by its multifunctional enzyme, open ...The LINE-1 (L1) retrotransposon is an ancient genetic parasite that has written around one-third of the human genome through a 'copy and paste' mechanism catalysed by its multifunctional enzyme, open reading frame 2 protein (ORF2p). ORF2p reverse transcriptase (RT) and endonuclease activities have been implicated in the pathophysiology of cancer, autoimmunity and ageing, making ORF2p a potential therapeutic target. However, a lack of structural and mechanistic knowledge has hampered efforts to rationally exploit it. We report structures of the human ORF2p 'core' (residues 238-1061, including the RT domain) by X-ray crystallography and cryo-electron microscopy in several conformational states. Our analyses identified two previously undescribed folded domains, extensive contacts to RNA templates and associated adaptations that contribute to unique aspects of the L1 replication cycle. Computed integrative structural models of full-length ORF2p show a dynamic closed-ring conformation that appears to open during retrotransposition. We characterize ORF2p RT inhibition and reveal its underlying structural basis. Imaging and biochemistry show that non-canonical cytosolic ORF2p RT activity can produce RNA:DNA hybrids, activating innate immune signalling through cGAS/STING and resulting in interferon production. In contrast to retroviral RTs, L1 RT is efficiently primed by short RNAs and hairpins, which probably explains cytosolic priming. Other biochemical activities including processivity, DNA-directed polymerization, non-templated base addition and template switching together allow us to propose a revised L1 insertion model. Finally, our evolutionary analysis demonstrates structural conservation between ORF2p and other RNA- and DNA-dependent polymerases. We therefore provide key mechanistic insights into L1 polymerization and insertion, shed light on the evolutionary history of L1 and enable rational drug development targeting L1. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40856.map.gz emd_40856.map.gz | 61.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40856-v30.xml emd-40856-v30.xml emd-40856.xml emd-40856.xml | 22.9 KB 22.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40856_fsc.xml emd_40856_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40856.png emd_40856.png | 36 KB | ||
| マスクデータ |  emd_40856_msk_1.map emd_40856_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-40856.cif.gz emd-40856.cif.gz | 6.8 KB | ||
| その他 |  emd_40856_additional_1.map.gz emd_40856_additional_1.map.gz emd_40856_half_map_1.map.gz emd_40856_half_map_1.map.gz emd_40856_half_map_2.map.gz emd_40856_half_map_2.map.gz | 117 MB 116.2 MB 116.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40856 http://ftp.pdbj.org/pub/emdb/structures/EMD-40856 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40856 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40856 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40856_validation.pdf.gz emd_40856_validation.pdf.gz | 856.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40856_full_validation.pdf.gz emd_40856_full_validation.pdf.gz | 856.1 KB | 表示 | |
| XML形式データ |  emd_40856_validation.xml.gz emd_40856_validation.xml.gz | 19 KB | 表示 | |
| CIF形式データ |  emd_40856_validation.cif.gz emd_40856_validation.cif.gz | 24.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40856 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40856 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40856 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40856 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40856.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40856.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map of apo ORF2p core | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.65 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40856_msk_1.map emd_40856_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
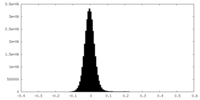
| 密度ヒストグラム |
-追加マップ: 4.5 angstrom lowpass filtered map
| ファイル | emd_40856_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 4.5 angstrom lowpass filtered map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
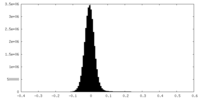
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_40856_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
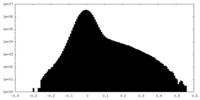
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_40856_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Long Interspersed Nuclear Element (LINE)-1 ORF2 protein
| 全体 | 名称: Long Interspersed Nuclear Element (LINE)-1 ORF2 protein |
|---|---|
| 要素 |
|
-超分子 #1: Long Interspersed Nuclear Element (LINE)-1 ORF2 protein
| 超分子 | 名称: Long Interspersed Nuclear Element (LINE)-1 ORF2 protein タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: LINE-1 retrotransposable element ORF2 protein
| 分子 | 名称: LINE-1 retrotransposable element ORF2 protein / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTGSNSHITI LTLNVNGLNS PIKRHRLASW IKSQDPSVCC IQETHLTCRD THRLKIKGWR KIYQANGKQK KAGVAILVSD KTDFKPTKIK RDKEGHYIMV KGSIQQEELT ILNIYAPNTG APRFIKQVLS DLQRDLDSHT LIMGDFNTPL SILDRSTRQK VNKDTQELNS ...文字列: MTGSNSHITI LTLNVNGLNS PIKRHRLASW IKSQDPSVCC IQETHLTCRD THRLKIKGWR KIYQANGKQK KAGVAILVSD KTDFKPTKIK RDKEGHYIMV KGSIQQEELT ILNIYAPNTG APRFIKQVLS DLQRDLDSHT LIMGDFNTPL SILDRSTRQK VNKDTQELNS ALHQTDLIDI YRTLHPKSTE YTFFSAPHHT YSKIDHIVGS KALLSKCKRT EIITNYLSDH SAIKLELRIK NLTQSRSTTW KLNNLLLNDY WVHNEMKAEI KMFFETNENK DTTYQNLWDA FKAVCRGKFI ALNAYKRKQE RSKIDTLTSQ LKELEKQEQT HSKASRRQEI TKIRAELKEI ETQKTLQKIN ESRSWFFERI NKIDRPLARL IKKKREKNQI DTIKNDKGDI TTDPTEIQTT IREYYKHLYA NKLENLEEMD TFLDTYTLPR LNQEEVESLN RPITGSEIVA IINSLPTKKS PGPDGFTAEF YQRYKEELVP FLLKLFQSIE KEGILPNSFY EASIILIPKP GRDTTKKENF RPISLMNIDA KILNKILANR IQQHIKKLIH HDQVGFIPGM QGWFNIRKSI NVIQHINRAK DKNHVIISID AEKAFDKIQQ PFMLKTLNKL GIDGMYLKII RAIYDKPTAN IILNGQKLEA FPLKTGTRQG CPLSPLLFNI VLEVLARAIR QEKEIKGIQL GKEEVKLSLF ADDMIVYLEN PIVSAQNLLK LISNFSKVSG YKINVQKSQA FLYNNNRQTE SQIMGELPFT IASKRIKYLG IQLTRDVKDL FKENYKPLLK EIKEDTNKWK NIPCSWVGRI NIVKMAILPK VIYRFNAIPI KLPMTFFTEL EKTTLKFIWN QKRARIAKSI LSQKNKAGGI TLPDFKLYYK ATVTKTAWYW YQNRDIDQWN RTEPSEIMPH IYNYLIFDKP EKNKQWGKDS LLNKWCWENW LAICRKLKLD PFLTPYTKIN SRWIKDLNVK PKTIKTLEEN LGITIQDIGV GKDFMSKTPK AMATKDKIDK WDLIKLKSFC TAKETTIRVN RQPTTWEKIF ATYSSDKGLI SRIYNELKQI YKKKTNNPIK KWAKDMNRHF SKEDIYAAKK HMKKCSSSLA IREMQIKTTM RYHLTPVRMA IIKKSGNNRC WRGCGEIGTL VHCWWDCKLV QPLWKSVWRF LRDLELEIPF DPAIPLLGIY PKDYKSCCYK DTCTRMFIAA LFTIAKTWNQ PNCPTMIDWI KKMWHIYTME YYAAIKNDEF ISFVGTWMKL ETIILSKLSQ EQKTKHRIFS LIGGN |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.15 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
詳細: 20mM HEPES pH 7.6, 150mM NaCl, 2mM MgOAc, 2mM DTT, 2mM dTTP | |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: LEICA EM CPC / 詳細: Manually blotted from the back. | |||||||||||||||
| 詳細 | Sample was monodisperse though unstable |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 6411 / 平均電子線量: 51.0 e/Å2 詳細: Super-resolution images collected in dose-fractionation mode over 38 frames wiht a dose per frame of 1.32 e/A2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)