+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3400 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
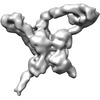
| タイトル | Lambda excision HJ intermediate | |||||||||
 マップデータ マップデータ | Map calculated from 10956 particles and fully CTF corrected | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | bacteriophage lambda / excision / site-specfific recombination / Holliday junction | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報IHF-DNA complex / provirus excision / integrase activity / DNA-binding transcription activator activity / protein-DNA complex / DNA integration / viral genome integration into host DNA / establishment of integrated proviral latency / structural constituent of chromatin / 転移酵素; リンを含む基を移すもの; 核酸を移すもの ...IHF-DNA complex / provirus excision / integrase activity / DNA-binding transcription activator activity / protein-DNA complex / DNA integration / viral genome integration into host DNA / establishment of integrated proviral latency / structural constituent of chromatin / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / regulation of translation / chromosome / transferase activity / DNA recombination / 加水分解酵素; エステル加水分解酵素 / hydrolase activity / transcription cis-regulatory region binding / symbiont entry into host cell / DNA-templated transcription / regulation of DNA-templated transcription / DNA binding / identical protein binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Enterobacteria phage lambda (λファージ) / Enterobacteria phage lambda (λファージ) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 11.0 Å | |||||||||
 データ登録者 データ登録者 | Laxmikanthan G / Xu C / Brilot AF / Warren D / Steele L / Seah N / Tong W / Grigorieff N / Landy A / Van Duyne G | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2016 ジャーナル: Elife / 年: 2016タイトル: Structure of a Holliday junction complex reveals mechanisms governing a highly regulated DNA transaction. 著者: Gurunathan Laxmikanthan / Chen Xu / Axel F Brilot / David Warren / Lindsay Steele / Nicole Seah / Wenjun Tong / Nikolaus Grigorieff / Arthur Landy / Gregory D Van Duyne /  要旨: The molecular machinery responsible for DNA expression, recombination, and compaction has been difficult to visualize as functionally complete entities due to their combinatorial and structural ...The molecular machinery responsible for DNA expression, recombination, and compaction has been difficult to visualize as functionally complete entities due to their combinatorial and structural complexity. We report here the structure of the intact functional assembly responsible for regulating and executing a site-specific DNA recombination reaction. The assembly is a 240-bp Holliday junction (HJ) bound specifically by 11 protein subunits. This higher-order complex is a key intermediate in the tightly regulated pathway for the excision of bacteriophage λ viral DNA out of the E. coli host chromosome, an extensively studied paradigmatic model system for the regulated rearrangement of DNA. Our results provide a structural basis for pre-existing data describing the excisive and integrative recombination pathways, and they help explain their regulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3400.map.gz emd_3400.map.gz | 7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3400-v30.xml emd-3400-v30.xml emd-3400.xml emd-3400.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-3400.jpg EMD-3400.jpg | 47 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3400 http://ftp.pdbj.org/pub/emdb/structures/EMD-3400 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3400 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3400 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3400_validation.pdf.gz emd_3400_validation.pdf.gz | 308.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3400_full_validation.pdf.gz emd_3400_full_validation.pdf.gz | 308.1 KB | 表示 | |
| XML形式データ |  emd_3400_validation.xml.gz emd_3400_validation.xml.gz | 5.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3400 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3400 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3400 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3400 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5j0nMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10065 (タイトル: Lambda excision HJ intermediate / Data size: 4.0 EMPIAR-10065 (タイトル: Lambda excision HJ intermediate / Data size: 4.0 Data #1: Particle stack and Frealign parameter file [picked particles - multiframe - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3400.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3400.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map calculated from 10956 particles and fully CTF corrected | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.8 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Lambda excision HJ intermediate
+超分子 #1000: Lambda excision HJ intermediate
+分子 #1: attB(-21) to attP(+117)
+分子 #2: attB(-19 to +21)
+分子 #3: attP(-79) to attB(+19)
+分子 #4: attP (-117 to +79)
+分子 #5: Int
+分子 #6: Int
+分子 #7: IHFa
+分子 #8: IHFb
+分子 #9: Xis
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 10mM Tris, 50mM NaCl |
| グリッド | 詳細: 1.2/1.3 400-mesh copper C-flat |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / 装置: FEI VITROBOT MARK II / 手法: 10 seconds blotting (two-sided) |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 温度 | 最低: 90 K / 最高: 90 K |
| 詳細 | Images were collected as 25-frame movies with a 2-second exposure |
| 日付 | 2014年6月11日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) デジタル化 - サンプリング間隔: 14 µm / 実像数: 1359 / 平均電子線量: 35.5 e/Å2 詳細: Every image is the average of all 25 aligned movie frames |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 100000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 4.793 µm / 最小 デフォーカス(公称値): 1.627 µm / 倍率(公称値): 78000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Manual particle selection with e2boxer (EMAN2), 2D classification with ISAC, initial map generation with e2initialmodel (EMAN2), 3D refinement and classification with Frealign v9.11 |
|---|---|
| CTF補正 | 詳細: Each particle |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 11.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Frealign, v9 詳細: Final map was the best class, out of size 3D classes 使用した粒子像数: 10956 |
| 最終 2次元分類 | クラス数: 6 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D / Chain - #4 - Chain ID: I / Chain - #5 - Chain ID: J / Chain - #6 - Chain ID: K / Chain - #7 - Chain ID: L |
|---|---|
| ソフトウェア | 名称: CNS 1.3 |
| 詳細 | Manual docking followed by highly restrained DEN-refinement in CNS |
| 精密化 | 空間: RECIPROCAL / プロトコル: FLEXIBLE FIT / 温度因子: 500 / 当てはまり具合の基準: HLML |
| 得られたモデル |  PDB-5j0n: |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - #0 - Chain ID: 1 / Chain - #1 - Chain ID: 2 / Chain - #2 - Chain ID: 3 |
|---|---|
| ソフトウェア | 名称: CNS 1.3 |
| 詳細 | Manual docking followed by highly restrained DEN-refinement in CNS |
| 精密化 | 空間: RECIPROCAL / プロトコル: FLEXIBLE FIT / 温度因子: 500 / 当てはまり具合の基準: HLML |
| 得られたモデル |  PDB-5j0n: |
-原子モデル構築 3
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D / Chain - #4 - Chain ID: E / Chain - #5 - Chain ID: F |
|---|---|
| ソフトウェア | 名称: CNS 1.3 |
| 詳細 | Manual docking followed by highly restrained DEN-refinement in CNS |
| 精密化 | 空間: RECIPROCAL / プロトコル: FLEXIBLE FIT / 温度因子: 500 / 当てはまり具合の基準: HLML |
| 得られたモデル |  PDB-5j0n: |
 ムービー
ムービー コントローラー
コントローラー