+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of Formate Dehydrogenase 1 from Methylorubrum extorquens AM1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex / OXIDOREDUCTASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報formate dehydrogenase / formate metabolic process / formate dehydrogenase / oxidoreductase complex / formate dehydrogenase (NAD+) activity / molybdopterin cofactor binding / cellular respiration / NADH dehydrogenase (ubiquinone) activity / 2 iron, 2 sulfur cluster binding / FMN binding ...formate dehydrogenase / formate metabolic process / formate dehydrogenase / oxidoreductase complex / formate dehydrogenase (NAD+) activity / molybdopterin cofactor binding / cellular respiration / NADH dehydrogenase (ubiquinone) activity / 2 iron, 2 sulfur cluster binding / FMN binding / 4 iron, 4 sulfur cluster binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Methylorubrum extorquens AM1 (バクテリア) Methylorubrum extorquens AM1 (バクテリア) | |||||||||
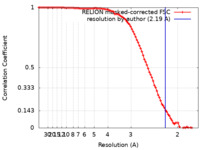
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.19 Å | |||||||||
 データ登録者 データ登録者 | Yoshikawa T / Makino F / Miyata T / Suzuki Y / Tanaka H / Namba K / Sowa K / Kitazumi Y / Shirai O | |||||||||
| 資金援助 |  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: Chem Commun (Camb) / 年: 2022 ジャーナル: Chem Commun (Camb) / 年: 2022タイトル: Multiple electron transfer pathways of tungsten-containing formate dehydrogenase in direct electron transfer-type bioelectrocatalysis. 著者: Tatsushi Yoshikawa / Fumiaki Makino / Tomoko Miyata / Yohei Suzuki / Hideaki Tanaka / Keiichi Namba / Kenji Kano / Keisei Sowa / Yuki Kitazumi / Osamu Shirai /  要旨: Tungsten-containing formate dehydrogenase from AM1 (FoDH1)-a promising biocatalyst for the interconversion of carbon dioxide/formate and nicotine adenine dinucleotide (NAD)/NADH redox couples-was ...Tungsten-containing formate dehydrogenase from AM1 (FoDH1)-a promising biocatalyst for the interconversion of carbon dioxide/formate and nicotine adenine dinucleotide (NAD)/NADH redox couples-was investigated using structural biology and bioelectrochemistry. FoDH1 is reported to be an enzyme that can realize "direct electron transfer (DET)-type bioelectrocatalysis." However, its 3-D structure, electrode-active sites, and electron transfer (ET) pathways remain unclear. The ET pathways were investigated using structural information, electrostatic interactions between the electrode and the enzyme, and the differences in the substrates. Two electrode-active sites and multiple ET pathways in FoDH1 were discovered. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32151.map.gz emd_32151.map.gz | 60 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32151-v30.xml emd-32151-v30.xml emd-32151.xml emd-32151.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32151_fsc.xml emd_32151_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32151.png emd_32151.png | 71 KB | ||
| Filedesc metadata |  emd-32151.cif.gz emd-32151.cif.gz | 6.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32151 http://ftp.pdbj.org/pub/emdb/structures/EMD-32151 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32151 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32151 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32151_validation.pdf.gz emd_32151_validation.pdf.gz | 631.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32151_full_validation.pdf.gz emd_32151_full_validation.pdf.gz | 630.7 KB | 表示 | |
| XML形式データ |  emd_32151_validation.xml.gz emd_32151_validation.xml.gz | 10.3 KB | 表示 | |
| CIF形式データ |  emd_32151_validation.cif.gz emd_32151_validation.cif.gz | 13.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32151 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32151 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32151 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32151 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7vw6MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32151.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32151.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.87 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Formate Dehydrogenase 1 from Methylorubrum extorquens AM1
| 全体 | 名称: Formate Dehydrogenase 1 from Methylorubrum extorquens AM1 |
|---|---|
| 要素 |
|
-超分子 #1: Formate Dehydrogenase 1 from Methylorubrum extorquens AM1
| 超分子 | 名称: Formate Dehydrogenase 1 from Methylorubrum extorquens AM1 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 / 詳細: Soluble Heterodimer |
|---|---|
| 由来(天然) | 生物種:  Methylorubrum extorquens AM1 (バクテリア) Methylorubrum extorquens AM1 (バクテリア) |
| 分子量 | 理論値: 170 KDa |
-分子 #1: Formate dehydrogenase
| 分子 | 名称: Formate dehydrogenase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: formate dehydrogenase |
|---|---|
| 由来(天然) | 生物種:  Methylorubrum extorquens AM1 (バクテリア) / 株: ATCC 14718 / DSM 1338 / JCM 2805 / NCIMB 9133 / AM1 Methylorubrum extorquens AM1 (バクテリア) / 株: ATCC 14718 / DSM 1338 / JCM 2805 / NCIMB 9133 / AM1 |
| 分子量 | 理論値: 107.464 KDa |
| 配列 | 文字列: MSNGPEPHGN KIEQPEIRAD ERQDAGGPAN GAPSTSGGAY SQGAKSGGQA APDPSGSYGI KDAPVAPATI AFEFDGQQVE AQPGETIWA VAKRLGTHIP HLCHKPDPGY RPDGNCRACM VEIEGERVLA ASCKRTPAIG MKVKSATERA TKARAMVLEL L VADQPERA ...文字列: MSNGPEPHGN KIEQPEIRAD ERQDAGGPAN GAPSTSGGAY SQGAKSGGQA APDPSGSYGI KDAPVAPATI AFEFDGQQVE AQPGETIWA VAKRLGTHIP HLCHKPDPGY RPDGNCRACM VEIEGERVLA ASCKRTPAIG MKVKSATERA TKARAMVLEL L VADQPERA TSHDPSSHFW VQADVLDVTE SRFPAAERWT SDVSHPAMSV NLDACIQCNL CVRACREVQV NDVIGMAYRA AG SKVVFDF DDPMGGSTCV ACGECVQACP TGALMPAAYL DANQTRTVYP DREVKSLCPY CGVGCQVSYK VKDERIVYAE GVN GPANQN RLCVKGRFGF DYVHHPHRLT VPLIRLENVP KDANDQVDPA NPWTHFREAT WEEALDRAAG GLKAIRDTNG RKAL AGFGS AKGSNEEAYL FQKLVRLGFG TNNVDHCTRL CHASSVAALM EGLNSGAVTA PFSAALDAEV IVVIGANPTV NHPVA ATFL KNAVKQRGAK LIIMDPRRQT LSRHAYRHLA FRPGSDVAML NAMLNVIVTE GLYDEQYIAG YTENFEALRE KIVDFT PEK MASVCGIDAE TLREVARLYA RAKSSLIFWG MGVSQHVHGT DNSRCLIALA LITGQIGRPG TGLHPLRGQN NVQGASD AG LIPMVYPDYQ SVEKDAVREL FEEFWGQSLD PQKGLTVVEI MRAIHAGEIR GMFVEGENPA MSDPDLNHAR HALAMLDH L VVQDLFLTET AFHADVVLPA SAFAEKAGTF TNTDRRVQIA QPVVAPPGDA RQDWWIIQEL ARRLDLDWNY GGPADIFAE MAQVMPSLNN ITWERLEREG AVTYPVDAPD QPGNEIIFYA GFPTESGRAK IVPAAIVPPD EVPDDEFPMV LSTGRVLEHW HTGSMTRRA GVLDALEPEA VAFMAPKELY RLGLRPGGSM RLETRRGAVV LKVRSDRDVP IGMIFMPFCY AEAAANLLTN P ALDPLGKI PEFKFCAARV VPAEAAPMAA E UniProtKB: formate dehydrogenase |
-分子 #2: Tungsten-containing formate dehydrogenase beta subunit
| 分子 | 名称: Tungsten-containing formate dehydrogenase beta subunit タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: formate dehydrogenase |
|---|---|
| 由来(天然) | 生物種:  Methylorubrum extorquens AM1 (バクテリア) / 株: ATCC 14718 / DSM 1338 / JCM 2805 / NCIMB 9133 / AM1 Methylorubrum extorquens AM1 (バクテリア) / 株: ATCC 14718 / DSM 1338 / JCM 2805 / NCIMB 9133 / AM1 |
| 分子量 | 理論値: 62.397508 KDa |
| 配列 | 文字列: MSEASGTVRS FAHPGRGRNV ARAVPKGRQV DPHAKVEIEE LLGTRPRQRD LLIEHLHLIQ DTYGQISADH LAALADEMSL AFAEVFETA TFYAHFDVVK EGEADIPRLT IRVCDSITCA MFGADELLET LQRELASDAV RVVRAPCVGL CDHAPAVEVG H NFLHRADL ...文字列: MSEASGTVRS FAHPGRGRNV ARAVPKGRQV DPHAKVEIEE LLGTRPRQRD LLIEHLHLIQ DTYGQISADH LAALADEMSL AFAEVFETA TFYAHFDVVK EGEADIPRLT IRVCDSITCA MFGADELLET LQRELASDAV RVVRAPCVGL CDHAPAVEVG H NFLHRADL ASVRAAVEAE DTHAHIPTYV DYDAYRAGGG YATLERLRSG ELPVDDVLKV LDDGGLRGLG GAGFPTGRKW RS VRGEPGP RLMAVNGDEG EPGTFKDQLY LNTDPHRFLE GMLIGAHVVE AADVYIYLRD EYPISREILA REIAKLPEGG TRI HLRRGA GAYICGEESS LIESLEGKRG LPRHKPPFPF QVGLFNRPTL INNIETLFWV RDLIERGAEW WKSHGRNGRV GLRS YSVSG RVKEPGVKLA PAGLTIQELI DEYCGGISDG HSFAAYLPGG ASGGILPASM NDIPLDFGTL EKYGCFIGSA AVVIL SDQD DVRGAALNLM KFFEDESCGQ CTPCRSGTQK ARMLMENGVW DTDLLGELAQ CMRDASICGL GQAASNPVST VIKYFP DLF PEPRAVAAE UniProtKB: Tungsten-containing formate dehydrogenase beta subunit |
-分子 #3: IRON/SULFUR CLUSTER
| 分子 | 名称: IRON/SULFUR CLUSTER / タイプ: ligand / ID: 3 / コピー数: 4 / 式: SF4 |
|---|---|
| 分子量 | 理論値: 351.64 Da |
| Chemical component information |  ChemComp-FS1: |
-分子 #4: FE2/S2 (INORGANIC) CLUSTER
| 分子 | 名称: FE2/S2 (INORGANIC) CLUSTER / タイプ: ligand / ID: 4 / コピー数: 2 / 式: FES |
|---|---|
| 分子量 | 理論値: 175.82 Da |
| Chemical component information |  ChemComp-FES: |
-分子 #5: 2-AMINO-5,6-DIMERCAPTO-7-METHYL-3,7,8A,9-TETRAHYDRO-8-OXA-1,3,9,1...
| 分子 | 名称: 2-AMINO-5,6-DIMERCAPTO-7-METHYL-3,7,8A,9-TETRAHYDRO-8-OXA-1,3,9,10-TETRAAZA-ANTHRACEN-4-ONE GUANOSINE DINUCLEOTIDE タイプ: ligand / ID: 5 / コピー数: 2 / 式: MGD |
|---|---|
| 分子量 | 理論値: 740.557 Da |
| Chemical component information |  ChemComp-MGD: |
-分子 #6: TUNGSTEN ION
| 分子 | 名称: TUNGSTEN ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: W |
|---|---|
| 分子量 | 理論値: 183.84 Da |
-分子 #7: UNKNOWN ATOM OR ION
| 分子 | 名称: UNKNOWN ATOM OR ION / タイプ: ligand / ID: 7 / コピー数: 1 / 式: UNX |
|---|
-分子 #8: FLAVIN MONONUCLEOTIDE
| 分子 | 名称: FLAVIN MONONUCLEOTIDE / タイプ: ligand / ID: 8 / コピー数: 1 / 式: FMN |
|---|---|
| 分子量 | 理論値: 456.344 Da |
| Chemical component information |  ChemComp-FMN: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 80.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 7650 / 平均露光時間: 3.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源: OTHER |
| 電子光学系 | C2レンズ絞り径: 40.0 µm / 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 0.5 µm / 倍率(補正後): 57471 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-7vw6: |
 ムービー
ムービー コントローラー
コントローラー