+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EsN-dhsU36mm1 local map (map 2) | |||||||||
 マップデータ マップデータ | Raw map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | promoter-bound / initiation / TRANSCRIPTION | |||||||||
| 生物種 |    Aquifex aeolicus (バクテリア) Aquifex aeolicus (バクテリア) | |||||||||
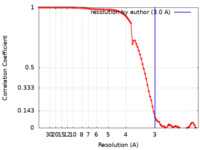
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Mueller AU / Chen J / Darst SA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: A general mechanism for transcription bubble nucleation in bacteria. 著者: Andreas U Mueller / James Chen / Mengyu Wu / Courtney Chiu / B Tracy Nixon / Elizabeth A Campbell / Seth A Darst /  要旨: Bacterial transcription initiation requires σ factors for nucleation of the transcription bubble. The canonical housekeeping σ factor, σ, nucleates DNA melting via recognition of conserved bases ...Bacterial transcription initiation requires σ factors for nucleation of the transcription bubble. The canonical housekeeping σ factor, σ, nucleates DNA melting via recognition of conserved bases of the promoter -10 motif, which are unstacked and captured in pockets of σ. By contrast, the mechanism of transcription bubble nucleation and formation during the unrelated σ-mediated transcription initiation is poorly understood. Herein, we combine structural and biochemical approaches to establish that σ, like σ, captures a flipped, unstacked base in a pocket formed between its N-terminal region I (RI) and extra-long helix features. Strikingly, RI inserts into the nascent bubble to stabilize the nucleated bubble prior to engagement of the obligate ATPase activator. Our data suggest a general paradigm of transcription initiation that requires σ factors to nucleate an early melted intermediate prior to productive RNA synthesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28794.map.gz emd_28794.map.gz | 32.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28794-v30.xml emd-28794-v30.xml emd-28794.xml emd-28794.xml | 22.1 KB 22.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28794_fsc.xml emd_28794_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28794.png emd_28794.png | 78.1 KB | ||
| マスクデータ |  emd_28794_msk_1.map emd_28794_msk_1.map emd_28794_msk_2.map emd_28794_msk_2.map | 64 MB 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-28794.cif.gz emd-28794.cif.gz | 4.9 KB | ||
| その他 |  emd_28794_additional_1.map.gz emd_28794_additional_1.map.gz emd_28794_additional_2.map.gz emd_28794_additional_2.map.gz emd_28794_additional_3.map.gz emd_28794_additional_3.map.gz emd_28794_half_map_1.map.gz emd_28794_half_map_1.map.gz emd_28794_half_map_2.map.gz emd_28794_half_map_2.map.gz | 59.8 MB 2.4 MB 3 MB 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28794 http://ftp.pdbj.org/pub/emdb/structures/EMD-28794 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28794 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28794 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28794_validation.pdf.gz emd_28794_validation.pdf.gz | 878.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28794_full_validation.pdf.gz emd_28794_full_validation.pdf.gz | 878.5 KB | 表示 | |
| XML形式データ |  emd_28794_validation.xml.gz emd_28794_validation.xml.gz | 16.5 KB | 表示 | |
| CIF形式データ |  emd_28794_validation.cif.gz emd_28794_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28794 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28794 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28794 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28794 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28794.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28794.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.083 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
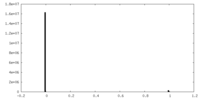
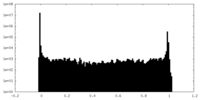
| ファイル |  emd_28794_msk_1.map emd_28794_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
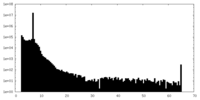
| 密度ヒストグラム |
-マスク #2
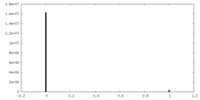
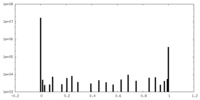
| ファイル |  emd_28794_msk_2.map emd_28794_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
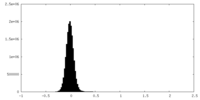
| 密度ヒストグラム |
-追加マップ: B-factor sharpened map (-110.1)
| ファイル | emd_28794_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | B-factor sharpened map (-110.1) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
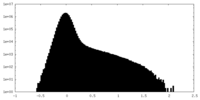
| 密度ヒストグラム |
-追加マップ: Locally filtered map (blocfilt)
| ファイル | emd_28794_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Locally filtered map (blocfilt) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
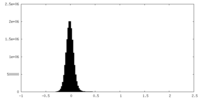
| 密度ヒストグラム |
-追加マップ: Local resolution map (blocres)
| ファイル | emd_28794_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution map (blocres) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_28794_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_28794_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : EsN-dhsU36mm1
| 全体 | 名称: EsN-dhsU36mm1 |
|---|---|
| 要素 |
|
-超分子 #1: EsN-dhsU36mm1
| 超分子 | 名称: EsN-dhsU36mm1 / タイプ: complex / ID: 1 / 親要素: 0 |
|---|
-超分子 #2: RNA polymerase complex
| 超分子 | 名称: RNA polymerase complex / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Aquifex aeolicus (バクテリア) / Synthetically produced: Yes Aquifex aeolicus (バクテリア) / Synthetically produced: Yes |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV 詳細: Octyl beta-D-glucopyranoside (beta-OG) was added to the sample to 0.1%w/v final concentration (from 10x stock) just prior to plunge vitrification. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 12294 / 平均露光時間: 2.0 sec. / 平均電子線量: 51.0 e/Å2 詳細: dose-fractionation with 0.05 seconds per frame (=40 frames) |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X