+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryogenic electron microscopy 3D map of human full-length monomeric alpha-catenin | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | F-actin / cell-cell junction / CELL ADHESION | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
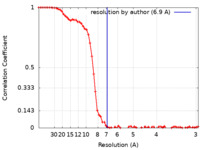
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.9 Å | |||||||||
 データ登録者 データ登録者 | Rangarajan ES / Izard T | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2023 ジャーナル: Commun Biol / 年: 2023タイトル: Distinct inter-domain interactions of dimeric versus monomeric α-catenin link cell junctions to filaments. 著者: Erumbi S Rangarajan / Emmanuel W Smith / Tina Izard /  要旨: Attachment between cells is crucial for almost all aspects of the life of cells. These inter-cell adhesions are mediated by the binding of transmembrane cadherin receptors of one cell to cadherins of ...Attachment between cells is crucial for almost all aspects of the life of cells. These inter-cell adhesions are mediated by the binding of transmembrane cadherin receptors of one cell to cadherins of a neighboring cell. Inside the cell, cadherin binds β-catenin, which interacts with α-catenin. The transitioning of cells between migration and adhesion is modulated by α-catenin, which links cell junctions and the plasma membrane to the actin cytoskeleton. At cell junctions, a single β-catenin/α-catenin heterodimer slips along filamentous actin in the direction of cytoskeletal tension which unfolds clustered heterodimers to form catch bonds with F-actin. Outside cell junctions, α-catenin dimerizes and links the plasma membrane to F-actin. Under cytoskeletal tension, α-catenin unfolds and forms an asymmetric catch bond with F-actin. To understand the mechanism of this important α-catenin function, we determined the 2.7 Å cryogenic electron microscopy (cryoEM) structures of filamentous actin alone and bound to human dimeric α-catenin. Our structures provide mechanistic insights into the role of the α-catenin interdomain interactions in directing α-catenin function and suggest a bivalent mechanism. Further, our cryoEM structure of human monomeric α-catenin provides mechanistic insights into α-catenin autoinhibition. Collectively, our structures capture the initial α-catenin interaction with F-actin before the sensing of force, which is a crucial event in cell adhesion and human disease. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27717.map.gz emd_27717.map.gz | 20.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27717-v30.xml emd-27717-v30.xml emd-27717.xml emd-27717.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27717_fsc.xml emd_27717_fsc.xml | 8.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27717.png emd_27717.png | 85.5 KB | ||
| その他 |  emd_27717_half_map_1.map.gz emd_27717_half_map_1.map.gz emd_27717_half_map_2.map.gz emd_27717_half_map_2.map.gz | 20.7 MB 20.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27717 http://ftp.pdbj.org/pub/emdb/structures/EMD-27717 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27717 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27717 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27717_validation.pdf.gz emd_27717_validation.pdf.gz | 681 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27717_full_validation.pdf.gz emd_27717_full_validation.pdf.gz | 680.6 KB | 表示 | |
| XML形式データ |  emd_27717_validation.xml.gz emd_27717_validation.xml.gz | 11.7 KB | 表示 | |
| CIF形式データ |  emd_27717_validation.cif.gz emd_27717_validation.cif.gz | 16 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27717 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27717 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27717 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27717 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27717.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27717.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.48 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_27717_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
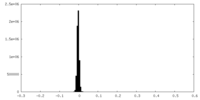
| 投影像・断面図 |
| ||||||||||||
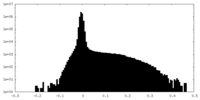
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27717_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
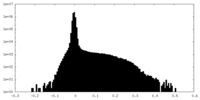
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Monomeric alpha-catenin (residues 1-906)
| 全体 | 名称: Monomeric alpha-catenin (residues 1-906) |
|---|---|
| 要素 |
|
-超分子 #1: Monomeric alpha-catenin (residues 1-906)
| 超分子 | 名称: Monomeric alpha-catenin (residues 1-906) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 100.3 KDa |
-分子 #1: alpha-catenin
| 分子 | 名称: alpha-catenin / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPLGSMTAVH AGNINFKWDP KSLEIRTLAV ERLLEPLVTQ VTTLVNTNSK GPSNKKRGRS KKAHVLAASV EQATENFLEK GDKIAKESQF LKEELVAAVE DVRKQGDLMK AAAGEFADDP CSSVKRGNMV RAARALLSAV TRLLILADMA DVYKLLVQLK VVEDGILKLR ...文字列: GPLGSMTAVH AGNINFKWDP KSLEIRTLAV ERLLEPLVTQ VTTLVNTNSK GPSNKKRGRS KKAHVLAASV EQATENFLEK GDKIAKESQF LKEELVAAVE DVRKQGDLMK AAAGEFADDP CSSVKRGNMV RAARALLSAV TRLLILADMA DVYKLLVQLK VVEDGILKLR NAGNEQDLGI QYKALKPEVD KLNIMAAKRQ QELKDVGHRD QMAAARGILQ KNVPILYTAS QACLQHPDVA AYKANRDLIY KQLQQAVTGI SNAAQATASD DASQHQGGGG GELAYALNNF DKQIIVDPLS FSEERFRPSL EERLESIISG AALMADSSCT RDDRRERIVA ECNAVRQALQ DLLSEYMGNA GRKERSDALN SAIDKMTKKT RDLRRQLRKA VMDHVSDSFL ETNVPLLVLI EAAKNGNEKE VKEYAQVFRE HANKLIEVAN LACSISNNEE GVKLVRMSAS QLEALCPQVI NAALALAAKP QSKLAQENMD LFKEQWEKQV RVLTDAVDDI TSIDDFLAVS ENHILEDVNK CVIALQEKDV DGLDRTAGAI RGRAARVIHV VTSEMDNYEP GVYTEKVLEA TKLLSNTVMP RFTEQVEAAV EALSSDPAQP MDENEFIDAS RLVYDGIRDI RKAVLMIRTP EELDDSDFET EDFDVRSRTS VQTEDDQLIA GQSARAIMAQ LPQEQKAKIA EQVASFQEEK SKLDAEVSKW DDSGNDIIVL AKQMCMIMME MTDFTRGKGP LKNTSDVISA AKKIAEAGSR MDKLGRTIAD HCPDSACKQD LLAYLQRIAL YCHQLNICSK VKAEVQNLGG ELVVSGVDSA MSLIQAAKNL MNAVVQTVKA SYVASTKYQK SQGMASLNLP AVSWKMKAPE KKPLVKREKQ DETQTKIKRA SQKKHVNPVQ ALSEFKAMDS I |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.25 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 294.15 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 特殊光学系 | 位相板: OTHER エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均露光時間: 0.1 sec. / 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X