+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of VcINDY-apo | ||||||||||||||||||
 マップデータ マップデータ | Phenix Sharpened map | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | TRANSPORTER / MEMBRANE PROTEIN | ||||||||||||||||||
| 機能・相同性 | Citrate transporter-like domain / Citrate transporter / Sodium/sulphate symporter, conserved site / Sodium:sulfate symporter family signature. / Solute carrier family 13 / transmembrane transporter activity / membrane / Transporter, NadC family 機能・相同性情報 機能・相同性情報 | ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
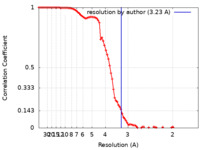
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.23 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Sauer DB / Marden JJ | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural basis of ion - substrate coupling in the Na-dependent dicarboxylate transporter VcINDY. 著者: David B Sauer / Jennifer J Marden / Joseph C Sudar / Jinmei Song / Christopher Mulligan / Da-Neng Wang /   要旨: The Na-dependent dicarboxylate transporter from Vibrio cholerae (VcINDY) is a prototype for the divalent anion sodium symporter (DASS) family. While the utilization of an electrochemical Na gradient ...The Na-dependent dicarboxylate transporter from Vibrio cholerae (VcINDY) is a prototype for the divalent anion sodium symporter (DASS) family. While the utilization of an electrochemical Na gradient to power substrate transport is well established for VcINDY, the structural basis of this coupling between sodium and substrate binding is not currently understood. Here, using a combination of cryo-EM structure determination, succinate binding and site-directed cysteine alkylation assays, we demonstrate that the VcINDY protein couples sodium- and substrate-binding via a previously unseen cooperative mechanism by conformational selection. In the absence of sodium, substrate binding is abolished, with the succinate binding regions exhibiting increased flexibility, including HPb, TM10b and the substrate clamshell motifs. Upon sodium binding, these regions become structurally ordered and create a proper binding site for the substrate. Taken together, these results provide strong evidence that VcINDY's conformational selection mechanism is a result of the sodium-dependent formation of the substrate binding site. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25756.map.gz emd_25756.map.gz | 58.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25756-v30.xml emd-25756-v30.xml emd-25756.xml emd-25756.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25756_fsc.xml emd_25756_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25756.png emd_25756.png | 80.7 KB | ||
| Filedesc metadata |  emd-25756.cif.gz emd-25756.cif.gz | 5.8 KB | ||
| その他 |  emd_25756_additional_1.map.gz emd_25756_additional_1.map.gz emd_25756_half_map_1.map.gz emd_25756_half_map_1.map.gz emd_25756_half_map_2.map.gz emd_25756_half_map_2.map.gz | 31.9 MB 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25756 http://ftp.pdbj.org/pub/emdb/structures/EMD-25756 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25756 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25756 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25756_validation.pdf.gz emd_25756_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25756_full_validation.pdf.gz emd_25756_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_25756_validation.xml.gz emd_25756_validation.xml.gz | 16.6 KB | 表示 | |
| CIF形式データ |  emd_25756_validation.cif.gz emd_25756_validation.cif.gz | 21 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25756 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25756 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25756 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25756 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7t9fMC  7t9gC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 電子顕微鏡画像生データ |  EMPIAR-10970 (タイトル: Particle stacks for VcINDY in Choline Chloride EMPIAR-10970 (タイトル: Particle stacks for VcINDY in Choline ChlorideData size: 383.6 Data #1: Particle stack of VcINDY in Choline Chloride Class 1 [picked particles - multiframe - processed] Data #2: Particle stack of VcINDY in Choline Chloride Class 1 [picked particles - multiframe - processed] Data #3: Particle stack of VcINDY in Choline Chloride Class 3 [picked particles - multiframe - processed] Data #4: Particle stack of VcINDY in Choline Chloride Class 4 [picked particles - multiframe - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25756.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25756.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Phenix Sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
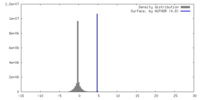
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_25756_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
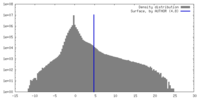
| 密度ヒストグラム |
-ハーフマップ: Halfmap A
| ファイル | emd_25756_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
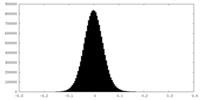
| 密度ヒストグラム |
-ハーフマップ: Halfmap B
| ファイル | emd_25756_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
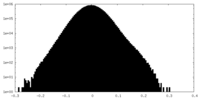
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : VcINDY in the Ci-apo state
| 全体 | 名称: VcINDY in the Ci-apo state |
|---|---|
| 要素 |
|
-超分子 #1: VcINDY in the Ci-apo state
| 超分子 | 名称: VcINDY in the Ci-apo state / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: DASS family sodium-coupled anion symporter
| 分子 | 名称: DASS family sodium-coupled anion symporter / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 48.157359 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: REWFLHRNSL IVLADVALFL ALYHFLPFEH NVVLGISMLA FIAVLWLTEA LHVTVTAILV PVMAVFFGIF ETQAALNNFA NSIIFLFLG GFALAAAMHH QGLDKVIADK VLAMAQGKMS VAVFMLFGVT ALLSMWISNT ATAAMMLPLV LGVLSKVDAD K QRSTYVFV ...文字列: REWFLHRNSL IVLADVALFL ALYHFLPFEH NVVLGISMLA FIAVLWLTEA LHVTVTAILV PVMAVFFGIF ETQAALNNFA NSIIFLFLG GFALAAAMHH QGLDKVIADK VLAMAQGKMS VAVFMLFGVT ALLSMWISNT ATAAMMLPLV LGVLSKVDAD K QRSTYVFV LLGVAYSASI GGIATLVGSP PNAIAAAEVG LSFTDWMKFG LPTAMMMLPM AIAILYFLLK PTLNGMFELD RA PVNWDKG KVVTLGIFGL TVFLWIFSSP INAALGGFKS FDTLVALGAI LMLSFARVVH WKEIQKTADW GVLLLFGGGL CLS NVLKQT GTSVFLANAL SDMVSHMGIF VVILVVATFV VFLTEFASNT ASAALLIPVF ATVAEAFGMS PVLLSVLIAV AASC AFMLP VATPPNAIVF ASGHIKQSEM MRVGLYLNIA CIGLLTAIAM LFWQ UniProtKB: Transporter, NadC family |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 4514 / 平均露光時間: 2.34 sec. / 平均電子線量: 65.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)