+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | T-cylinder of Synechocystis PCC 6803 Phycobilisome, complex with OCP - local refinement | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex / light harvesting / pigment / PHOTOSYNTHESIS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報light absorption / 付加脱離酵素(リアーゼ) / phycobilisome / chloride ion binding / plasma membrane-derived thylakoid membrane / photoreceptor activity / photosynthesis / lyase activity 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Sauer PV / Sutter M | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: Structures of a phycobilisome in light-harvesting and photoprotected states. 著者: María Agustina Domínguez-Martín / Paul V Sauer / Henning Kirst / Markus Sutter / David Bína / Basil J Greber / Eva Nogales / Tomáš Polívka / Cheryl A Kerfeld /    要旨: Phycobilisome (PBS) structures are elaborate antennae in cyanobacteria and red algae. These large protein complexes capture incident sunlight and transfer the energy through a network of embedded ...Phycobilisome (PBS) structures are elaborate antennae in cyanobacteria and red algae. These large protein complexes capture incident sunlight and transfer the energy through a network of embedded pigment molecules called bilins to the photosynthetic reaction centres. However, light harvesting must also be balanced against the risks of photodamage. A known mode of photoprotection is mediated by orange carotenoid protein (OCP), which binds to PBS when light intensities are high to mediate photoprotective, non-photochemical quenching. Here we use cryogenic electron microscopy to solve four structures of the 6.2 MDa PBS, with and without OCP bound, from the model cyanobacterium Synechocystis sp. PCC 6803. The structures contain a previously undescribed linker protein that binds to the membrane-facing side of PBS. For the unquenched PBS, the structures also reveal three different conformational states of the antenna, two previously unknown. The conformational states result from positional switching of two of the rods and may constitute a new mode of regulation of light harvesting. Only one of the three PBS conformations can bind to OCP, which suggests that not every PBS is equally susceptible to non-photochemical quenching. In the OCP-PBS complex, quenching is achieved through the binding of four 34 kDa OCPs organized as two dimers. The complex reveals the structure of the active form of OCP, in which an approximately 60 Å displacement of its regulatory carboxy terminal domain occurs. Finally, by combining our structure with spectroscopic properties, we elucidate energy transfer pathways within PBS in both the quenched and light-harvesting states. Collectively, our results provide detailed insights into the biophysical underpinnings of the control of cyanobacterial light harvesting. The data also have implications for bioengineering PBS regulation in natural and artificial light-harvesting systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25033.map.gz emd_25033.map.gz | 88.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25033-v30.xml emd-25033-v30.xml emd-25033.xml emd-25033.xml | 23.8 KB 23.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_25033.png emd_25033.png | 185.1 KB | ||
| マスクデータ |  emd_25033_msk_1.map emd_25033_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-25033.cif.gz emd-25033.cif.gz | 7.1 KB | ||
| その他 |  emd_25033_half_map_1.map.gz emd_25033_half_map_1.map.gz emd_25033_half_map_2.map.gz emd_25033_half_map_2.map.gz | 165 MB 165 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25033 http://ftp.pdbj.org/pub/emdb/structures/EMD-25033 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25033 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25033 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25033_validation.pdf.gz emd_25033_validation.pdf.gz | 975.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25033_full_validation.pdf.gz emd_25033_full_validation.pdf.gz | 975.5 KB | 表示 | |
| XML形式データ |  emd_25033_validation.xml.gz emd_25033_validation.xml.gz | 15.1 KB | 表示 | |
| CIF形式データ |  emd_25033_validation.cif.gz emd_25033_validation.cif.gz | 18 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25033 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25033 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25033 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25033 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25033.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25033.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_25033_msk_1.map emd_25033_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
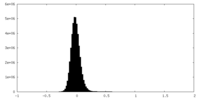
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_25033_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_25033_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
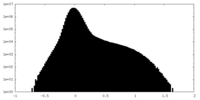
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of phycobilisome from Synechocystis PCC 6803 with OCP
| 全体 | 名称: Complex of phycobilisome from Synechocystis PCC 6803 with OCP |
|---|---|
| 要素 |
|
-超分子 #1: Complex of phycobilisome from Synechocystis PCC 6803 with OCP
| 超分子 | 名称: Complex of phycobilisome from Synechocystis PCC 6803 with OCP タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Allophycocyanin alpha chain
| 分子 | 名称: Allophycocyanin alpha chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: PCC 6803 / Kazusa |
| 分子量 | 理論値: 17.429807 KDa |
| 配列 | 文字列: MSIVTKSIVN ADAEARYLSP GELDRIKAFV TGGAARLRIA ETLTGSRETI VKQAGDRLFQ KRPDIVSPGG NAYGEEMTAT CLRDMDYYL RLVTYGVVSG DVTPIEEIGL VGVREMYRSL GTPIEAVAQS VREMKEVASG LMSSDDAAEA SAYFDFVIGK M S UniProtKB: Allophycocyanin alpha chain |
-分子 #2: Allophycocyanin beta chain
| 分子 | 名称: Allophycocyanin beta chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: PCC 6803 / Kazusa |
| 分子量 | 理論値: 17.231604 KDa |
| 配列 | 文字列: MQDAITAVIN SADVQGKYLD GAAMDKLKSY FASGELRVRA ASVISANAAT IVKEAVAKSL LYSDVTRPGG NMYTTRRYAA CIRDLDYYL RYATYAMLAG DASILDERVL NGLKETYNSL GVPISSTVQA IQAIKEVTAS LVGADAGKEM GVYLDYICSG L S UniProtKB: Allophycocyanin beta chain |
-分子 #3: Phycobilisome 7.8 kDa linker polypeptide, allophycocyanin-associa...
| 分子 | 名称: Phycobilisome 7.8 kDa linker polypeptide, allophycocyanin-associated, core タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: PCC 6803 / Kazusa |
| 分子量 | 理論値: 7.817174 KDa |
| 配列 | 文字列: MRMFRITACV PSQTRIRTQR ELQNTYFTKL VPYDNWFREQ QRIMKMGGKI VKVELATGRP GTNAGLA UniProtKB: Phycobilisome 7.8 kDa linker polypeptide, allophycocyanin-associated, core |
-分子 #4: Phycobiliprotein ApcE
| 分子 | 名称: Phycobiliprotein ApcE / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO / EC番号: 付加脱離酵素(リアーゼ) |
|---|---|
| 由来(天然) | 生物種:  株: PCC 6803 / Kazusa |
| 分子量 | 理論値: 100.412703 KDa |
| 配列 | 文字列: MSVKASGGSS LARPQLYQTV PVSAISQAEQ QDRFLEGSEL NELTAYFQSG ALRLEIAETL TQNADLIVSR AANRIFTGGS PLSYLEKPV ERQPALVGAS SDSRNGSVTY AESNGSGGLF GGLRSVFSST GPIPPGFRPI NIARYGPSNM QKSLRDMSWF L RYTTYAIV ...文字列: MSVKASGGSS LARPQLYQTV PVSAISQAEQ QDRFLEGSEL NELTAYFQSG ALRLEIAETL TQNADLIVSR AANRIFTGGS PLSYLEKPV ERQPALVGAS SDSRNGSVTY AESNGSGGLF GGLRSVFSST GPIPPGFRPI NIARYGPSNM QKSLRDMSWF L RYTTYAIV AGDPNIIVVN TRGLKEVIEN ACSIDATIVA IQEMRAASAD YFRNNAQAKE IVLQYFDILL SEFKAPTPAN KV RQGPSND IQGLELPQSY FNAAAKRQKY AMKPGLSALE KNAVIKAAYR QIFERDITKA YSQSISYLES QVRNGDISMK EFV RRLAKS PLYRKQFFEP FINSRALELA FRHILGRGPS SREEVQKYFS IVSSGGLPAL VDALVDSQEY ADYFGEETVP YLRG LGVEA QECRNWGMQQ DLFSYSAPFR KVPQFITTFA QYDRPLPDQH VYGSGNDPLE IQFGAIFPKE TRNPSKRPAP FNKDT KRIL IHRGPAVNNQ VGNPSAVGEF PGSLGAKVFR LNGGLPGAKV GKNTGTSVKF GESSTQALIR AAYRQVFGRD LYEGQR LSV AEIQLENGDI SVREFIKRLA KSELFLKLYW APHYVCKAIE YMHRRLLGRP TYGRQEMNQY FDIASKQGFY AVVEAMI DS KEYSDAFGED TVPYERYLTP GGLQMRSARV GSLREDIGQR VDKEVTPRFV ELGQVSAIRT EPEIAYRSNQ GVTRQRQQ T KVFKLVSTYD KVAVKNAIRA AYRQVFERDL EPYIINSEFT ALESKLSNNE INVKEFIEGL GTSELYMKEF YAPYPNTKV IEMGTKHFLG RAPLNQKEIQ QYNQILASQG LKAFIGAMVN GMEYLQTFGE DTVPYRRFPT LPAANFPNTE RLYNKLTKQD KELVVPSFT PVVKVGG UniProtKB: Phycobiliprotein ApcE |
-分子 #5: Orange carotenoid-binding protein
| 分子 | 名称: Orange carotenoid-binding protein / タイプ: protein_or_peptide / ID: 5 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: PCC 6803 / Kazusa |
| 分子量 | 理論値: 34.684605 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPFTIDSARG IFPNTLAADV VPATIARFSQ LNAEDQLALI WFAYLEMGKT LTIAAPGAAS MQLAENALKE IQAMGPLQQT QAMCDLANR ADTPLCRTYA SWSPNIKLGF WYRLGELMEQ GFVAPIPAGY QLSANANAVL ATIQGLESGQ QITVLRNAVV D MGFTAGKD ...文字列: MPFTIDSARG IFPNTLAADV VPATIARFSQ LNAEDQLALI WFAYLEMGKT LTIAAPGAAS MQLAENALKE IQAMGPLQQT QAMCDLANR ADTPLCRTYA SWSPNIKLGF WYRLGELMEQ GFVAPIPAGY QLSANANAVL ATIQGLESGQ QITVLRNAVV D MGFTAGKD GKRIAEPVVP PQDTASRTKV SIEGVTNATV LNYMDNLNAN DFDTLIELFT SDGALQPPFQ RPIVGKENVL RF FREECQN LKLIPERGVT EPAEDGFTQI KVTGKVQTPW FGGNVGMNIA WRFLLNPEGK IFFVAIDLLA SPKELLNFAR UniProtKB: Orange carotenoid-binding protein |
-分子 #6: Phycobilisome rod-core linker polypeptide CpcG
| 分子 | 名称: Phycobilisome rod-core linker polypeptide CpcG / タイプ: protein_or_peptide / ID: 6 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: PCC 6803 / Kazusa |
| 分子量 | 理論値: 28.938758 KDa |
| 配列 | 文字列: MALPLLNYAP KSQNVRVEGY EIGSEEKPVV FTTENILSSS DMDNLIEAAY RQIFFHAFKW DREKVLESQL RNGQITVRDF VRGLLLSNT FRNSFYEKNS NYRFVEHCVQ KILGRDVYSE REKIAWSIVV ATKGYQGLID DLLNSDEYLN NFGYDTVPYQ R RRNLPGRE ...文字列: MALPLLNYAP KSQNVRVEGY EIGSEEKPVV FTTENILSSS DMDNLIEAAY RQIFFHAFKW DREKVLESQL RNGQITVRDF VRGLLLSNT FRNSFYEKNS NYRFVEHCVQ KILGRDVYSE REKIAWSIVV ATKGYQGLID DLLNSDEYLN NFGYDTVPYQ R RRNLPGRE AGELPFNIKS PRYDAYHRRQ LGFPQIVWQN EVRRFIPQEK KLTAGNPMNF LGMARSINPA ANTIPKVSAQ NI NIEASVP RR UniProtKB: Phycobilisome rod-core linker polypeptide CpcG |
-分子 #7: PHYCOCYANOBILIN
| 分子 | 名称: PHYCOCYANOBILIN / タイプ: ligand / ID: 7 / コピー数: 24 / 式: CYC |
|---|---|
| 分子量 | 理論値: 588.694 Da |
| Chemical component information |  ChemComp-CYC: |
-分子 #8: beta,beta-carotene-4,4'-dione
| 分子 | 名称: beta,beta-carotene-4,4'-dione / タイプ: ligand / ID: 8 / コピー数: 2 / 式: 45D |
|---|---|
| 分子量 | 理論値: 564.84 Da |
| Chemical component information |  ChemComp-45D: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE-PROPANE / 装置: FEI VITROBOT MARK IV / 詳細: Manual blotting. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-7scc: |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)