+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Sub-tomogram average of the C. elegans ATP synthase dimer | |||||||||
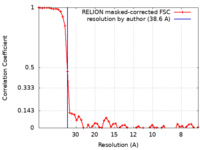
 マップデータ マップデータ | Post-processed sub-tomogram averaging map of the C. elegans ATP synthase dimer at a nominal resolution of 38.6 Angstrom. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Supercomplex / dimer / enzyme / respiration. / MEMBRANE PROTEIN | |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 38.6 Å | |||||||||
 データ登録者 データ登録者 | Buzzard EJ / McLaren M / Gold VAM | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Biochem J / 年: 2024 ジャーナル: Biochem J / 年: 2024タイトル: The consequence of ATP synthase dimer angle on mitochondrial morphology studied by cryo-electron tomography. 著者: Emma Buzzard / Mathew McLaren / Piotr Bragoszewski / Andrea Brancaccio / Holly Ford / Bertram Daum / Patricia Kuwabara / Ian Collinson / Vicki Gold /    要旨: Mitochondrial ATP synthases form rows of dimers, which induce membrane curvature to give cristae their characteristic lamellar or tubular morphology. The angle formed between the central stalks of ...Mitochondrial ATP synthases form rows of dimers, which induce membrane curvature to give cristae their characteristic lamellar or tubular morphology. The angle formed between the central stalks of ATP synthase dimers varies between species. Using cryo-electron tomography and sub-tomogram averaging, we determined the structure of the ATP synthase dimer from the nematode worm C. elegans and show that the dimer angle differs from previously determined structures. The consequences of this species-specific difference at the dimer interface were investigated by comparing C. elegans and S. cerevisiae mitochondrial morphology. We reveal that C. elegans has a larger ATP synthase dimer angle with more lamellar (flatter) cristae when compared to yeast. The underlying cause of this difference was investigated by generating an atomic model of the C. elegans ATP synthase dimer by homology modelling. A comparison of our C. elegans model to an existing S. cerevisiae structure reveals the presence of extensions and rearrangements in C. elegans subunits associated with maintaining the dimer interface. We speculate that increasing dimer angles could provide an advantage for species that inhabit variable-oxygen environments by forming flatter more energetically efficient cristae. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18991.map.gz emd_18991.map.gz | 2.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18991-v30.xml emd-18991-v30.xml emd-18991.xml emd-18991.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18991_fsc.xml emd_18991_fsc.xml | 5.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18991.png emd_18991.png | 53.9 KB | ||
| マスクデータ |  emd_18991_msk_1.map emd_18991_msk_1.map | 10.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18991.cif.gz emd-18991.cif.gz | 4.7 KB | ||
| その他 |  emd_18991_half_map_1.map.gz emd_18991_half_map_1.map.gz emd_18991_half_map_2.map.gz emd_18991_half_map_2.map.gz | 7.3 MB 7.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18991 http://ftp.pdbj.org/pub/emdb/structures/EMD-18991 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18991 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18991 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18991_validation.pdf.gz emd_18991_validation.pdf.gz | 758.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18991_full_validation.pdf.gz emd_18991_full_validation.pdf.gz | 758.2 KB | 表示 | |
| XML形式データ |  emd_18991_validation.xml.gz emd_18991_validation.xml.gz | 10.7 KB | 表示 | |
| CIF形式データ |  emd_18991_validation.cif.gz emd_18991_validation.cif.gz | 14 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18991 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18991 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18991 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18991 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18991.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18991.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed sub-tomogram averaging map of the C. elegans ATP synthase dimer at a nominal resolution of 38.6 Angstrom. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.58 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18991_msk_1.map emd_18991_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1 from relion refinement job used...
| ファイル | emd_18991_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 from relion refinement job used to generate final post-processed map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2 from relion refinement job used...
| ファイル | emd_18991_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 from relion refinement job used to generate final post-processed map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : C. elegans mitochondrial ATP synthase dimer
| 全体 | 名称: C. elegans mitochondrial ATP synthase dimer |
|---|---|
| 要素 |
|
-超分子 #1: C. elegans mitochondrial ATP synthase dimer
| 超分子 | 名称: C. elegans mitochondrial ATP synthase dimer / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | Mitochondria isolated from C. elegans underwent 2-3 freeze-thaw cycles, to break open the mitochondria and release the cristae. Tomography was completed using these crista membranes. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 3 / 平均露光時間: 2.8 sec. / 平均電子線量: 1.36 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 7.0 µm / 最小 デフォーカス(公称値): 5.0 µm / 倍率(公称値): 39000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)