+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AApoAII amyloid fibril Morphology II (ex vivo) | |||||||||
 マップデータ マップデータ | AApoAII fibril density map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | amyloid / protein fibril / systemic amyloidosis / misfolding disease / helical | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報triglyceride-rich lipoprotein particle remodeling / lipase inhibitor activity / high-density lipoprotein particle receptor binding / spherical high-density lipoprotein particle / high-density lipoprotein particle binding / apolipoprotein receptor binding / lipoprotein metabolic process / chylomicron / phosphatidylcholine binding / high-density lipoprotein particle remodeling ...triglyceride-rich lipoprotein particle remodeling / lipase inhibitor activity / high-density lipoprotein particle receptor binding / spherical high-density lipoprotein particle / high-density lipoprotein particle binding / apolipoprotein receptor binding / lipoprotein metabolic process / chylomicron / phosphatidylcholine binding / high-density lipoprotein particle remodeling / very-low-density lipoprotein particle / high-density lipoprotein particle assembly / cholesterol transport / low-density lipoprotein particle remodeling / cholesterol binding / cholesterol metabolic process / cholesterol homeostasis / blood microparticle 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
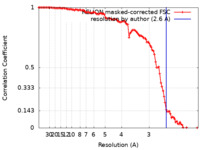
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Andreotti G / Schmidt M / Faendrich M | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2024 ジャーナル: J Mol Biol / 年: 2024タイトル: Insights into the Structural Basis of Amyloid Resistance Provided by Cryo-EM Structures of AApoAII Amyloid Fibrils. 著者: Giada Andreotti / Julian Baur / Marijana Ugrina / Peter Benedikt Pfeiffer / Max Hartmann / Sebastian Wiese / Hiroki Miyahara / Keiichi Higuchi / Nadine Schwierz / Matthias Schmidt / Marcus Fändrich /   要旨: Amyloid resistance is the inability or the reduced susceptibility of an organism to develop amyloidosis. In this study we have analysed the molecular basis of the resistance to systemic AApoAII ...Amyloid resistance is the inability or the reduced susceptibility of an organism to develop amyloidosis. In this study we have analysed the molecular basis of the resistance to systemic AApoAII amyloidosis, which arises from the formation of amyloid fibrils from apolipoprotein A-II (ApoA-II). The disease affects humans and animals, including SAMR1C mice that express the C allele of ApoA-II protein, whereas other mouse strains are resistant to development of amyloidosis due to the expression of other ApoA-II alleles, such as ApoA-IIF. Using cryo-electron microscopy, molecular dynamics simulations and other methods, we have determined the structures of pathogenic AApoAII amyloid fibrils from SAMR1C mice and analysed the structural effects of ApoA-IIF-specific mutational changes. Our data show that these changes render ApoA-IIF incompatible with the specific fibril morphologies, with which ApoA-II protein can become pathogenic in vivo. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17104.map.gz emd_17104.map.gz | 834 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17104-v30.xml emd-17104-v30.xml emd-17104.xml emd-17104.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17104_fsc.xml emd_17104_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17104.png emd_17104.png | 60.5 KB | ||
| マスクデータ |  emd_17104_msk_1.map emd_17104_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17104.cif.gz emd-17104.cif.gz | 5.6 KB | ||
| その他 |  emd_17104_additional_1.map.gz emd_17104_additional_1.map.gz emd_17104_half_map_1.map.gz emd_17104_half_map_1.map.gz emd_17104_half_map_2.map.gz emd_17104_half_map_2.map.gz | 5.9 MB 80.3 MB 80.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17104 http://ftp.pdbj.org/pub/emdb/structures/EMD-17104 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17104 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17104 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17104_validation.pdf.gz emd_17104_validation.pdf.gz | 783.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17104_full_validation.pdf.gz emd_17104_full_validation.pdf.gz | 782.9 KB | 表示 | |
| XML形式データ |  emd_17104_validation.xml.gz emd_17104_validation.xml.gz | 17.8 KB | 表示 | |
| CIF形式データ |  emd_17104_validation.cif.gz emd_17104_validation.cif.gz | 23.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17104 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17104 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17104 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17104 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8oq4MC  8oq5C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17104.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17104.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | AApoAII fibril density map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17104_msk_1.map emd_17104_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
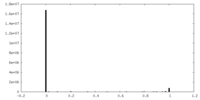
| 密度ヒストグラム |
-追加マップ: Extended AApoAII fibril density map
| ファイル | emd_17104_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Extended AApoAII fibril density map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
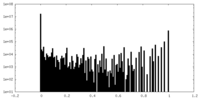
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_17104_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
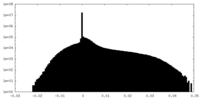
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_17104_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
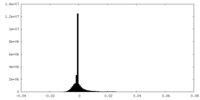
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : AApoAII amyloid fibril
| 全体 | 名称: AApoAII amyloid fibril |
|---|---|
| 要素 |
|
-超分子 #1: AApoAII amyloid fibril
| 超分子 | 名称: AApoAII amyloid fibril / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: AApoAII amyloid fibrils extracted from SAMR1C mice. |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Apolipoprotein A-II
| 分子 | 名称: Apolipoprotein A-II / タイプ: protein_or_peptide / ID: 1 / コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 8.746728 KDa |
| 配列 | 文字列: QADGQDMQSL FTQYFQSMTE YGKDLVEKAK TSEIQSQAKA YFEKTHEQLT PLVRSAGTSL VNFFSSLMNL EEKPAPAA UniProtKB: Apolipoprotein A-II |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 詳細: Water |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 294.15 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 平均露光時間: 12.0 sec. / 平均電子線量: 42.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: SPOT SCAN / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 130000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8oq4: |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X