+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The lipid linked oligosaccharide polymerase Wzy and its regulating co-polymerase Wzz form a complex in vivo and in vitro | |||||||||||||||
 マップデータ マップデータ | WzzE:WzyE complex of Pectobacterium atrosepticum. Sharpened map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | enterobacterial common antigen / lipopolysaccharide / Wzx/Wzy pathway / polysaccharide polymerase / polysaccharide co-polymerase / MEMBRANE PROTEIN | |||||||||||||||
| 機能・相同性 | ECA polysaccharide chain length modulation protein WzzE / enterobacterial common antigen biosynthetic process / Polysaccharide chain length determinant N-terminal domain / Chain length determinant protein / lipopolysaccharide biosynthetic process / plasma membrane / ECA polysaccharide chain length modulation protein 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Pectobacterium atrosepticum (バクテリア) Pectobacterium atrosepticum (バクテリア) | |||||||||||||||
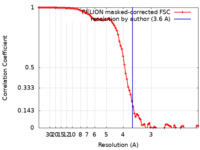
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||||||||
 データ登録者 データ登録者 | Weckener M / Woodward LS / Clarke BR / Liu H / Ward PN / Le Bas A / Bhella D / Whitfield C / Naismith JH | |||||||||||||||
| 資金援助 |  英国, 英国,  カナダ, 4件 カナダ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Open Biol / 年: 2023 ジャーナル: Open Biol / 年: 2023タイトル: The lipid linked oligosaccharide polymerase Wzy and its regulating co-polymerase, Wzz, from enterobacterial common antigen biosynthesis form a complex. 著者: Miriam Weckener / Laura S Woodward / Bradley R Clarke / Huanting Liu / Philip N Ward / Audrey Le Bas / David Bhella / Chris Whitfield / James H Naismith /   要旨: The enterobacterial common antigen (ECA) is a carbohydrate polymer that is associated with the cell envelope in the . ECA contains a repeating trisaccharide which is polymerized by WzyE, a member of ...The enterobacterial common antigen (ECA) is a carbohydrate polymer that is associated with the cell envelope in the . ECA contains a repeating trisaccharide which is polymerized by WzyE, a member of the Wzy membrane protein polymerase superfamily. WzyE activity is regulated by a membrane protein polysaccharide co-polymerase, WzzE. Förster resonance energy transfer experiments demonstrate that WzyE and WzzE from form a complex , and immunoblotting and cryo-electron microscopy (cryo-EM) analysis confirm a defined stoichiometry of approximately eight WzzE to one WzyE. Low-resolution cryo-EM reconstructions of the complex, aided by an antibody recognizing the C-terminus of WzyE, reveals WzyE sits in the central membrane lumen formed by the octameric arrangement of the transmembrane helices of WzzE. The pairing of Wzy and Wzz is found in polymerization systems for other bacterial polymers, including lipopolysaccharide O-antigens and capsular polysaccharides. The data provide new structural insight into a conserved mechanism for regulating polysaccharide chain length in bacteria. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16364.map.gz emd_16364.map.gz | 59.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16364-v30.xml emd-16364-v30.xml emd-16364.xml emd-16364.xml | 20.3 KB 20.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16364_fsc.xml emd_16364_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16364.png emd_16364.png | 131.6 KB | ||
| Filedesc metadata |  emd-16364.cif.gz emd-16364.cif.gz | 6.3 KB | ||
| その他 |  emd_16364_additional_1.map.gz emd_16364_additional_1.map.gz emd_16364_half_map_1.map.gz emd_16364_half_map_1.map.gz emd_16364_half_map_2.map.gz emd_16364_half_map_2.map.gz | 56.2 MB 45.7 MB 45.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16364 http://ftp.pdbj.org/pub/emdb/structures/EMD-16364 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16364 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16364 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16364_validation.pdf.gz emd_16364_validation.pdf.gz | 922.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16364_full_validation.pdf.gz emd_16364_full_validation.pdf.gz | 922.5 KB | 表示 | |
| XML形式データ |  emd_16364_validation.xml.gz emd_16364_validation.xml.gz | 16.1 KB | 表示 | |
| CIF形式データ |  emd_16364_validation.cif.gz emd_16364_validation.cif.gz | 20.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16364 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16364 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16364 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16364 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8c0eMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16364.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16364.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | WzzE:WzyE complex of Pectobacterium atrosepticum. Sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
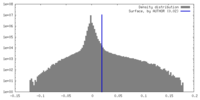
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: WzzE:WzyE complex of Pectobacterium atrosepticum. DeepEMhancer processed map...
| ファイル | emd_16364_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | WzzE:WzyE complex of Pectobacterium atrosepticum. DeepEMhancer processed map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
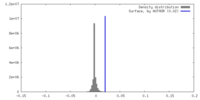
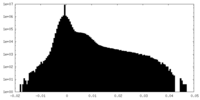
| 密度ヒストグラム |
-ハーフマップ: WzzE:WzyE complex of Pectobacterium atrosepticum. Half map 1
| ファイル | emd_16364_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | WzzE:WzyE complex of Pectobacterium atrosepticum. Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
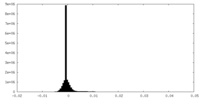
| 密度ヒストグラム |
-ハーフマップ: WzzE:WzyE complex of Pectobacterium atrosepticum. Half map 2
| ファイル | emd_16364_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | WzzE:WzyE complex of Pectobacterium atrosepticum. Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
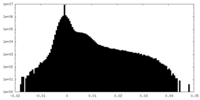
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : WzzE from Pectobacterium atrosepticum
| 全体 | 名称: WzzE from Pectobacterium atrosepticum |
|---|---|
| 要素 |
|
-超分子 #1: WzzE from Pectobacterium atrosepticum
| 超分子 | 名称: WzzE from Pectobacterium atrosepticum / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Polysaccharide polymerase co-factor |
|---|---|
| 由来(天然) | 生物種:  Pectobacterium atrosepticum (バクテリア) Pectobacterium atrosepticum (バクテリア) |
-分子 #1: ECA polysaccharide chain length modulation protein
| 分子 | 名称: ECA polysaccharide chain length modulation protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pectobacterium atrosepticum (バクテリア) Pectobacterium atrosepticum (バクテリア) |
| 分子量 | 理論値: 39.546781 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GAMVKSENLS TGNALIDNEL DIRGLFRLLW RGKVWPIAIG LLFAVVALGY SYLVKQEWGA TSITDKPTVN MLGGYYSQQQ FLRNLDARS FSSPQPEQPS ISAGAYDEFI MQLAAYDTRR DFWLQTDYYK QRLEGDEKAD AALLDELVNN IQFTARDDGK K TNDSVKLV ...文字列: GAMVKSENLS TGNALIDNEL DIRGLFRLLW RGKVWPIAIG LLFAVVALGY SYLVKQEWGA TSITDKPTVN MLGGYYSQQQ FLRNLDARS FSSPQPEQPS ISAGAYDEFI MQLAAYDTRR DFWLQTDYYK QRLEGDEKAD AALLDELVNN IQFTARDDGK K TNDSVKLV AETSSDSNTL LRQYVVFASQ RAANHLNEEI KGAWAARTIF MKSQIKRQEA VAKAIYDREV RSVELALKIA QQ QGISRSQ TDTPADEIPA SEMFLLGRPM LQARLETLQT SGPHYELDYD QNRAMLATLN VGPTLEASFQ TYRYLRTPEE PVK RDSPRR AFLMVMWGAI GVLVGAGVAL ARRLR UniProtKB: ECA polysaccharide chain length modulation protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 詳細: 50 mM phosphate buffer, pH 8, 200 mM NaCl, 0.026% (w/v) DDM |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Quantifoil R2/2 400 mesh grids were glow-discharged in the presence of pentyl-amine to drive protein into the ice.. |
| 詳細 | This membrane protein was purified in the presence of DDM |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 2347 / 平均露光時間: 8.5 sec. / 平均電子線量: 85.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | The model was built into a density modified map - processed using DeepEMhancer |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-8c0e: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)