+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-14022 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Volta phase plate cryo-ET of Magnetospirillum gryphiswaldense overproducing PopZ-Mgr | |||||||||
 マップデータ マップデータ | Volta phase plate cryo-electron tomography of Magnetospirillum gryphiswaldense (wild-type) overproducing PopZ-Mgr | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | PopZ / Alphaproteobacteria / cytoskeleton / polarity / CELL CYCLE | |||||||||
| 生物種 |  Magnetospirillum gryphiswaldense MSR-1 (走磁性) Magnetospirillum gryphiswaldense MSR-1 (走磁性) | |||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 | |||||||||
 データ登録者 データ登録者 | Toro-Nahuelpan M / Plitzko JM / Schueler D / Pfeiffer D | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2022 ジャーナル: J Mol Biol / 年: 2022タイトル: In vivo Architecture of the Polar Organizing Protein Z (PopZ) Meshwork in the Alphaproteobacteria Magnetospirillum gryphiswaldense and Caulobacter crescentus. 著者: Mauricio Toro-Nahuelpan / Jürgen M Plitzko / Dirk Schüler / Daniel Pfeiffer /  要旨: The polar organizing protein Z (PopZ) forms a polar microdomain that is inaccessible to larger macromolecules such as ribosomes, and selectively sequesters proteins crucial for cell cycle control and ...The polar organizing protein Z (PopZ) forms a polar microdomain that is inaccessible to larger macromolecules such as ribosomes, and selectively sequesters proteins crucial for cell cycle control and polar morphogenesis in various Alphaproteobacteria. However, the in vivo architecture of this microdomain has remained elusive. Here, we analyzed the three-dimensional ultrastructural organization of the PopZ network in Magnetospirillum gryphiswaldense and Caulobacter crescentus by Volta phase plate cryo-electron tomography, which provides high spatial resolution and improved image contrast. Our results suggest that PopZ forms a porous network of disordered short, flexible, and branching filaments. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14022.map.gz emd_14022.map.gz | 196.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14022-v30.xml emd-14022-v30.xml emd-14022.xml emd-14022.xml | 13.9 KB 13.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_14022.png emd_14022.png | 52 KB | ||
| マスクデータ |  emd_14022_msk_1.map emd_14022_msk_1.map | 1 GB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14022.cif.gz emd-14022.cif.gz | 5.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14022 http://ftp.pdbj.org/pub/emdb/structures/EMD-14022 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14022 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14022 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14022_validation.pdf.gz emd_14022_validation.pdf.gz | 672.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14022_full_validation.pdf.gz emd_14022_full_validation.pdf.gz | 672 KB | 表示 | |
| XML形式データ |  emd_14022_validation.xml.gz emd_14022_validation.xml.gz | 5 KB | 表示 | |
| CIF形式データ |  emd_14022_validation.cif.gz emd_14022_validation.cif.gz | 6.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14022 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14022 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14022 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14022 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 電子顕微鏡画像生データ |  EMPIAR-10885 (タイトル: In vivo architecture of the polar organizing protein Z (PopZ) meshwork in the Alphaproteobacteria Magnetospirillum gryphiswaldense and Caulobacter crescentus EMPIAR-10885 (タイトル: In vivo architecture of the polar organizing protein Z (PopZ) meshwork in the Alphaproteobacteria Magnetospirillum gryphiswaldense and Caulobacter crescentusData size: 6.3 Data #1: Volta phase plate cryo-electron tomography of Magnetospirillum gryphiswaldense (wild-type) overproducing PopZ-Mgr [reconstructed volumes] Data #2: Volta phase plate cryo-electron tomography of Magnetospirillum gryphiswaldense delta-mamK overproducing PopZ-Mgr [reconstructed volumes] Data #3: Volta phase plate cryo-electron tomography of purified PopZ-Mgr [reconstructed volumes] Data #4: Cryo-electron tomography of Magnetospirillum gryphiswaldense delta-popZ overproducing PopZ-Mgr [reconstructed volumes] Data #5: Volta phase plate cryo-electron tomography of Caulobacter crescentus NA1000 [reconstructed volumes] Data #6: Volta phase plate cryo-electron tomography of Caulobacter crescentus NA1000 overproducing PopZ-Cc [reconstructed volumes] Data #7: Cryo-electron tomography of Magnetospirillum gryphiswaldense delta-popZ [reconstructed volumes] Data #8: Volta phase plate cryo-electron tomography of Magnetospirillum gryphiswaldense delta-mamK-mamY [reconstructed volumes]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14022.map.gz / 形式: CCP4 / 大きさ: 263.6 MB / タイプ: IMAGE STORED AS SIGNED BYTE ダウンロード / ファイル: emd_14022.map.gz / 形式: CCP4 / 大きさ: 263.6 MB / タイプ: IMAGE STORED AS SIGNED BYTE | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Volta phase plate cryo-electron tomography of Magnetospirillum gryphiswaldense (wild-type) overproducing PopZ-Mgr | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 13.68 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
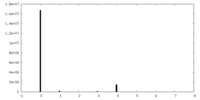
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_14022_msk_1.map emd_14022_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
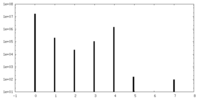
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Magnetospirillum gryphiswaldense (wild-type) overproducing PopZ-Mgr
| 全体 | 名称: Magnetospirillum gryphiswaldense (wild-type) overproducing PopZ-Mgr |
|---|---|
| 要素 |
|
-超分子 #1: Magnetospirillum gryphiswaldense (wild-type) overproducing PopZ-Mgr
| 超分子 | 名称: Magnetospirillum gryphiswaldense (wild-type) overproducing PopZ-Mgr タイプ: cell / ID: 1 / 親要素: 0 詳細: Individual segmented structures are depicted using different colors in the EMDB entry page image and in the corresponding publication: Putative PopZ filaments are depicted in white. MamK ...詳細: Individual segmented structures are depicted using different colors in the EMDB entry page image and in the corresponding publication: Putative PopZ filaments are depicted in white. MamK filaments are green. The flagellum is colored in gold. The cellular envelope inner and outer membranes are depicted in blue. |
|---|---|
| 由来(天然) | 生物種:  Magnetospirillum gryphiswaldense MSR-1 (走磁性) Magnetospirillum gryphiswaldense MSR-1 (走磁性) |
-超分子 #2: PopZ
| 超分子 | 名称: PopZ / タイプ: organelle_or_cellular_component / ID: 2 / 親要素: 1 詳細: The polar organizing protein Z (PopZ) forms a polar microdomain that is inaccessible to larger macromolecules such as ribosomes, and selectively sequesters proteins crucial for cell cycle ...詳細: The polar organizing protein Z (PopZ) forms a polar microdomain that is inaccessible to larger macromolecules such as ribosomes, and selectively sequesters proteins crucial for cell cycle control and polar morphogenesis in various Alphaproteobacteria. In the present strain PopZ overproduction was achieved via insertion of a Tn5-Ptet-based popZ overexpression cassette into the genome of the Magnetospirillum gryphiswaldense wild-type strain. |
|---|---|
| 由来(天然) | 生物種:  Magnetospirillum gryphiswaldense MSR-1 (走磁性) Magnetospirillum gryphiswaldense MSR-1 (走磁性) |
-超分子 #3: MamK
| 超分子 | 名称: MamK / タイプ: organelle_or_cellular_component / ID: 3 / 親要素: 1 詳細: The filament-forming bacterial actin MamK is important for organizing magnetosome organelles into chains that are used for navigation along geomagnetic field lines. Magnetosomes are ...詳細: The filament-forming bacterial actin MamK is important for organizing magnetosome organelles into chains that are used for navigation along geomagnetic field lines. Magnetosomes are membranous organelles containing nanometer-sized crystals of magnetite present in magnetotactic bacteria. |
|---|---|
| 由来(天然) | 生物種:  Magnetospirillum gryphiswaldense MSR-1 (走磁性) Magnetospirillum gryphiswaldense MSR-1 (走磁性) |
-超分子 #4: Cellular envelope
| 超分子 | 名称: Cellular envelope / タイプ: organelle_or_cellular_component / ID: 4 / 親要素: 1 / 詳細: inner and outer membranes |
|---|---|
| 由来(天然) | 生物種:  Magnetospirillum gryphiswaldense MSR-1 (走磁性) Magnetospirillum gryphiswaldense MSR-1 (走磁性) |
-超分子 #5: Flagellum
| 超分子 | 名称: Flagellum / タイプ: organelle_or_cellular_component / ID: 5 / 親要素: 1 詳細: Flagella are helical protein filaments powered by a rotary motor to mediate motility of bacteria. |
|---|---|
| 由来(天然) | 生物種:  Magnetospirillum gryphiswaldense MSR-1 (走磁性) Magnetospirillum gryphiswaldense MSR-1 (走磁性) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 詳細: modified flask standard medium (FSM) |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: MOLYBDENUM / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE 詳細: The mixture was blotted and embedded in vitreous ice by plunge freezing into liquid ethane (< - 170 C). The grids were stored in sealed boxes in liquid nitrogen until used.. |
| 切片作成 | その他: NO SECTIONING |
| 位置合わせマーカー | Manufacturer: Sigma-Aldrich / 直径: 15 nm |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 詳細 | Tomography was performed under low-dose conditions using a Titan Krios transmission electron microscope (FEI) equipped with a 300 kV field emission gun, and a Gatan Quantum post-column energy filter. Tilt series were acquired using Serial EM software. The specimen was tilted about one axis with 1.5 degree increments over a typical total angular range of -+ 60 degree. To account for the increased specimen thickness at high tilt angles, the exposure time was multiplied by a factor of 1/cos alpha. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 1.5 e/Å2 詳細: Data collection was performed at 300 kV, with the energy filter operated in the zero-loss mode (slit width of 20 eV). The cumulative electron dose during the tilt series was kept below 150 e- A-2. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 0.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Tomograms were reconstructed with (15 nm gold fiducials) the IMOD software package (https://bio3d.colorado.edu/imod/) and treated with an anisotropic non-linear diffusion denoising algorithm to improve signal-to-noise ratio (K: 1, Iterations: 10). Segmentation was performed using Amira software on binned volumes with a voxel size of 10.48 A and 13.68 A. Filaments were traced in Amira using an automated segmentation algorithm based on a generic cylinder as a template implemented in the X-Tracing extension. Prior to filament tracing, binned volumes were subjected to nonlocal-means filtering using Amira software (Thermo Fisher Scientific). The cylindrical templates were generated with a diameter and length of 6 and 15 nm, respectively. To reduce background noise, short filamentous structures with lengths below 30 nm were filtered out. Membrane segmentation was done using the software TomoSegMemTV and a complementary package, SynapSegTools, both for Matlab, and refined manually in Amira (Thermo Fisher Scientific). |
|---|---|
| 最終 再構成 | 使用した粒子像数: 321 |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X