+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13185 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Helical structure of the toxin MakA from Vibrio cholera | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Pore-forming toxin / Vibrio cholerae / TOXIN | |||||||||||||||
| 機能・相同性 | : / Hemolysin BL-binding component / Bacillus haemolytic enterotoxin (HBL) / membrane / Non-hemolytic enterotoxin lytic component L1 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Vibrio cholerae O1 biovar El Tor str. N16961 (コレラ菌) / Vibrio cholerae O1 biovar El Tor str. N16961 Vibrio cholerae O1 biovar El Tor str. N16961 (コレラ菌) / Vibrio cholerae O1 biovar El Tor str. N16961 | |||||||||||||||
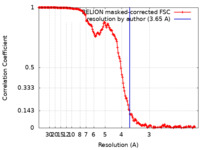
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.65 Å | |||||||||||||||
 データ登録者 データ登録者 | Berg A / Nadeem A | |||||||||||||||
| 資金援助 |  スウェーデン, European Union, 4件 スウェーデン, European Union, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: Protein-lipid interaction at low pH induces oligomerization of the MakA cytotoxin from . 著者: Aftab Nadeem / Alexandra Berg / Hudson Pace / Athar Alam / Eric Toh / Jörgen Ådén / Nikola Zlatkov / Si Lhyam Myint / Karina Persson / Gerhard Gröbner / Anders Sjöstedt / Marta Bally / ...著者: Aftab Nadeem / Alexandra Berg / Hudson Pace / Athar Alam / Eric Toh / Jörgen Ådén / Nikola Zlatkov / Si Lhyam Myint / Karina Persson / Gerhard Gröbner / Anders Sjöstedt / Marta Bally / Jonas Barandun / Bernt Eric Uhlin / Sun Nyunt Wai /  要旨: The α-pore-forming toxins (α-PFTs) from pathogenic bacteria damage host cell membranes by pore formation. We demonstrate a remarkable, hitherto unknown mechanism by an α-PFT protein from . As part ...The α-pore-forming toxins (α-PFTs) from pathogenic bacteria damage host cell membranes by pore formation. We demonstrate a remarkable, hitherto unknown mechanism by an α-PFT protein from . As part of the MakA/B/E tripartite toxin, MakA is involved in membrane pore formation similar to other α-PFTs. In contrast, MakA in isolation induces tube-like structures in acidic endosomal compartments of epithelial cells in vitro. The present study unravels the dynamics of tubular growth, which occurs in a pH-, lipid-, and concentration-dependent manner. Within acidified organelle lumens or when incubated with cells in acidic media, MakA forms oligomers and remodels membranes into high-curvature tubes leading to loss of membrane integrity. A 3.7 Å cryo-electron microscopy structure of MakA filaments reveals a unique protein-lipid superstructure. MakA forms a pinecone-like spiral with a central cavity and a thin annular lipid bilayer embedded between the MakA transmembrane helices in its active α-PFT conformation. Our study provides insights into a novel tubulation mechanism of an α-PFT protein and a new mode of action by a secreted bacterial toxin. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13185.map.gz emd_13185.map.gz | 342.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13185-v30.xml emd-13185-v30.xml emd-13185.xml emd-13185.xml | 19.4 KB 19.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13185_fsc.xml emd_13185_fsc.xml | 16.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13185.png emd_13185.png | 213 KB | ||
| Filedesc metadata |  emd-13185.cif.gz emd-13185.cif.gz | 6 KB | ||
| その他 |  emd_13185_additional_1.map.gz emd_13185_additional_1.map.gz emd_13185_half_map_1.map.gz emd_13185_half_map_1.map.gz emd_13185_half_map_2.map.gz emd_13185_half_map_2.map.gz | 5.9 MB 295.8 MB 295.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13185 http://ftp.pdbj.org/pub/emdb/structures/EMD-13185 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13185 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13185 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13185_validation.pdf.gz emd_13185_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13185_full_validation.pdf.gz emd_13185_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_13185_validation.xml.gz emd_13185_validation.xml.gz | 23.8 KB | 表示 | |
| CIF形式データ |  emd_13185_validation.cif.gz emd_13185_validation.cif.gz | 31.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13185 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13185 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13185 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13185 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7p3rMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10869 (タイトル: Helical structure of the toxin MakA from Vibrio cholera EMPIAR-10869 (タイトル: Helical structure of the toxin MakA from Vibrio choleraData size: 195.8 Data #1: Unaligned multi-frame micrographs of the oligomerized Vibrio cholerae toxin MakA [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13185.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13185.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.042 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: additional map 1
| ファイル | emd_13185_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | additional map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: hafl map 2
| ファイル | emd_13185_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | hafl map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: hafl map 1
| ファイル | emd_13185_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | hafl map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Vibrio cholera MakA filament
| 全体 | 名称: Vibrio cholera MakA filament |
|---|---|
| 要素 |
|
-超分子 #1: Vibrio cholera MakA filament
| 超分子 | 名称: Vibrio cholera MakA filament / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Vibrio cholerae O1 biovar El Tor str. N16961 (コレラ菌) Vibrio cholerae O1 biovar El Tor str. N16961 (コレラ菌) |
-分子 #1: MakA tetramer
| 分子 | 名称: MakA tetramer / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: Vibrio cholerae O1 biovar El Tor str. N16961 |
| 分子量 | 理論値: 39.028582 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSQQVTQLNP TQQTTQSAFL ATTVITAQCH AILNTQFTPP TVKPDWFDDL SKKLDSAKLV AKQWIDDLGP QVSASIPSSV INFDATFQA SIDAIHELYK ADPTASGKDN TTVQQASQIM TALSSQVSGI EATVKGMNKE LSDWGVKMQA AHDDLVNGAT N IQKTIIDL ...文字列: MSQQVTQLNP TQQTTQSAFL ATTVITAQCH AILNTQFTPP TVKPDWFDDL SKKLDSAKLV AKQWIDDLGP QVSASIPSSV INFDATFQA SIDAIHELYK ADPTASGKDN TTVQQASQIM TALSSQVSGI EATVKGMNKE LSDWGVKMQA AHDDLVNGAT N IQKTIIDL QTDIESMNNA IDNNRAAIEK LNKDLVYAQV AVGVGIFMLV AGVALTVATA GTAAAVSGGI AAVGAASIIA GG VTWGVLQ NQIDDDYDSI AQEQKQKAED QQQIIALQGL SNASSAVVSA IETSTSVLSD FETTWTVFGN ELDDVVTKLN NGA SMQSII MEKVMSDAAK NEWDDAVELA KQLASAKIAI ETKELAPAVK QAA UniProtKB: Non-hemolytic enterotoxin lytic component L1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 6.5 / 構成要素 - 濃度: 120.0 mM / 構成要素 - 名称: Sodium Citrate |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK I |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 2476 / 平均電子線量: 43.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.75 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X