+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10736 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Lumazine Synthase | |||||||||
 マップデータ マップデータ | Reconstruction of Lumazine Synthase, sharpened and filtered at 2 angstroms | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Aquifex aeolicus (バクテリア) Aquifex aeolicus (バクテリア) | |||||||||
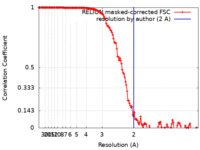
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.0 Å | |||||||||
 データ登録者 データ登録者 | Bhella D / Streetley J / Clarke M / Cowton V / Patel A / Bammes B / Hosogi N | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Biophys Rev / 年: 2019 ジャーナル: Biophys Rev / 年: 2019タイトル: Cryo-electron microscopy: an introduction to the technique, and considerations when working to establish a national facility. 著者: David Bhella /  | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10736.map.gz emd_10736.map.gz | 227.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10736-v30.xml emd-10736-v30.xml emd-10736.xml emd-10736.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10736_fsc.xml emd_10736_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10736.png emd_10736.png | 251.7 KB | ||
| マスクデータ |  emd_10736_msk_1.map emd_10736_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10736_half_map_1.map.gz emd_10736_half_map_1.map.gz emd_10736_half_map_2.map.gz emd_10736_half_map_2.map.gz | 193.5 MB 193.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10736 http://ftp.pdbj.org/pub/emdb/structures/EMD-10736 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10736 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10736 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10736_validation.pdf.gz emd_10736_validation.pdf.gz | 541.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10736_full_validation.pdf.gz emd_10736_full_validation.pdf.gz | 540.3 KB | 表示 | |
| XML形式データ |  emd_10736_validation.xml.gz emd_10736_validation.xml.gz | 20.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10736 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10736 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10736 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10736 | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| 電子顕微鏡画像生データ |  EMPIAR-10384 (タイトル: Cryo-EM structure of Lumazine Synthase / Data size: 3.0 TB EMPIAR-10384 (タイトル: Cryo-EM structure of Lumazine Synthase / Data size: 3.0 TBData #1: Frame aligned multi-frame movies of Lumazine Synthase imaged on a CryoARM300 and DE64 [micrographs - multiframe])  EMPIAR-10385 (タイトル: Cryo-EM structure of Lumazine Synthase / Data size: 6.8 TB EMPIAR-10385 (タイトル: Cryo-EM structure of Lumazine Synthase / Data size: 6.8 TBData #1: Frame aligned multi-frame movies of Lumazine Synthase imaged on a CryoARM300 and DE64 [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10736.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10736.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of Lumazine Synthase, sharpened and filtered at 2 angstroms | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.597 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10736_msk_1.map emd_10736_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_10736_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_10736_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Icosahedral complex of Lumazine Synthase
| 全体 | 名称: Icosahedral complex of Lumazine Synthase |
|---|---|
| 要素 |
|
-超分子 #1: Icosahedral complex of Lumazine Synthase
| 超分子 | 名称: Icosahedral complex of Lumazine Synthase / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Aquifex aeolicus (バクテリア) Aquifex aeolicus (バクテリア) |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 |
| グリッド | モデル: C-flat-1.2/1.3 4C / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
| 詳細 | Purified by immobilised metal affinity chromatography. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-64 (8k x 8k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 13.0 µm / デジタル化 - 画像ごとのフレーム数: 1-41 / 撮影したグリッド数: 1 / 実像数: 1199 / 平均露光時間: 10.0 sec. / 平均電子線量: 1.66 e/Å2 詳細: Images were acquired at 141 FPS with 2 x binning and electron counting. A total of 41 summed frames were written out. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 200000 |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X