+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10615 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | FAK structure from single particle analysis of 2D crystals | |||||||||
 マップデータ マップデータ | Map obtained from single particle analysis of FAK 2D crystals. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Kinase / Focal Adhesion / Membrane / CELL ADHESION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Apoptotic cleavage of cellular proteins / NCAM signaling for neurite out-growth / RAF/MAP kinase cascade / RHO GTPases Activate WASPs and WAVEs / Regulation of actin dynamics for phagocytic cup formation / radial glia-guided pyramidal neuron migration / negative regulation of protein autophosphorylation / calcium-dependent cysteine-type endopeptidase activity / positive regulation of substrate-dependent cell migration, cell attachment to substrate / Integrin signaling ...Apoptotic cleavage of cellular proteins / NCAM signaling for neurite out-growth / RAF/MAP kinase cascade / RHO GTPases Activate WASPs and WAVEs / Regulation of actin dynamics for phagocytic cup formation / radial glia-guided pyramidal neuron migration / negative regulation of protein autophosphorylation / calcium-dependent cysteine-type endopeptidase activity / positive regulation of substrate-dependent cell migration, cell attachment to substrate / Integrin signaling / GRB2:SOS provides linkage to MAPK signaling for Integrins / DCC mediated attractive signaling / MET activates PTK2 signaling / Extra-nuclear estrogen signaling / EPHB-mediated forward signaling / p130Cas linkage to MAPK signaling for integrins / VEGFA-VEGFR2 Pathway / angiogenesis involved in wound healing / signal complex assembly / response to pH / negative regulation of cell-substrate adhesion / wound healing, spreading of cells / positive regulation of focal adhesion assembly / positive regulation of protein tyrosine kinase activity / negative regulation of anoikis / regulation of cell adhesion / response to muscle stretch / ciliary basal body / molecular function activator activity / actin filament organization / non-specific protein-tyrosine kinase / non-membrane spanning protein tyrosine kinase activity / epidermal growth factor receptor signaling pathway / sarcolemma / integrin binding / positive regulation of protein binding / cell cortex / protein tyrosine kinase activity / protease binding / protein autophosphorylation / dendritic spine / positive regulation of cell migration / focal adhesion / centrosome / positive regulation of cell population proliferation / perinuclear region of cytoplasm / ATP binding / identical protein binding / nucleus / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
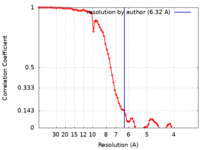
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.32 Å | |||||||||
 データ登録者 データ登録者 | Acebron I / Righetto R | |||||||||
| 資金援助 |  スペイン, スペイン,  スイス, 2件 スイス, 2件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2020 ジャーナル: EMBO J / 年: 2020タイトル: Structural basis of Focal Adhesion Kinase activation on lipid membranes. 著者: Iván Acebrón / Ricardo D Righetto / Christina Schoenherr / Svenja de Buhr / Pilar Redondo / Jayne Culley / Carlos F Rodríguez / Csaba Daday / Nikhil Biyani / Oscar Llorca / Adam Byron / ...著者: Iván Acebrón / Ricardo D Righetto / Christina Schoenherr / Svenja de Buhr / Pilar Redondo / Jayne Culley / Carlos F Rodríguez / Csaba Daday / Nikhil Biyani / Oscar Llorca / Adam Byron / Mohamed Chami / Frauke Gräter / Jasminka Boskovic / Margaret C Frame / Henning Stahlberg / Daniel Lietha /     要旨: Focal adhesion kinase (FAK) is a key component of the membrane proximal signaling layer in focal adhesion complexes, regulating important cellular processes, including cell migration, proliferation, ...Focal adhesion kinase (FAK) is a key component of the membrane proximal signaling layer in focal adhesion complexes, regulating important cellular processes, including cell migration, proliferation, and survival. In the cytosol, FAK adopts an autoinhibited state but is activated upon recruitment into focal adhesions, yet how this occurs or what induces structural changes is unknown. Here, we employ cryo-electron microscopy to reveal how FAK associates with lipid membranes and how membrane interactions unlock FAK autoinhibition to promote activation. Intriguingly, initial binding of FAK to the membrane causes steric clashes that release the kinase domain from autoinhibition, allowing it to undergo a large conformational change and interact itself with the membrane in an orientation that places the active site toward the membrane. In this conformation, the autophosphorylation site is exposed and multiple interfaces align to promote FAK oligomerization on the membrane. We show that interfaces responsible for initial dimerization and membrane attachment are essential for FAK autophosphorylation and resulting cellular activity including cancer cell invasion, while stable FAK oligomerization appears to be needed for optimal cancer cell proliferation in an anchorage-independent manner. Together, our data provide structural details of a key membrane bound state of FAK that is primed for efficient autophosphorylation and activation, hence revealing the critical event in integrin mediated FAK activation and signaling at focal adhesions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10615.map.gz emd_10615.map.gz | 48 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10615-v30.xml emd-10615-v30.xml emd-10615.xml emd-10615.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10615_fsc.xml emd_10615_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10615.png emd_10615.png | 225.7 KB | ||
| マスクデータ |  emd_10615_msk_1.map emd_10615_msk_1.map | 51.4 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-10615.cif.gz emd-10615.cif.gz | 6 KB | ||
| その他 |  emd_10615_half_map_1.map.gz emd_10615_half_map_1.map.gz emd_10615_half_map_2.map.gz emd_10615_half_map_2.map.gz | 19.7 MB 19.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10615 http://ftp.pdbj.org/pub/emdb/structures/EMD-10615 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10615 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10615 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10615_validation.pdf.gz emd_10615_validation.pdf.gz | 492.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10615_full_validation.pdf.gz emd_10615_full_validation.pdf.gz | 491.9 KB | 表示 | |
| XML形式データ |  emd_10615_validation.xml.gz emd_10615_validation.xml.gz | 14 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10615 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10615 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10615 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10615 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6ty3MC  6ty4C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10347 (タイトル: FAK structure from single particle analysis of 2D crystals EMPIAR-10347 (タイトル: FAK structure from single particle analysis of 2D crystalsData size: 1.8 TB Data #1: Dose-weighted aligned movie averages of the FAK AMP-PNP dataset [micrographs - single frame] Data #2: Unaligned movies of the FAK AMP-PNP dataset [micrographs - multiframe] Data #3: Stack of particles extracted from the FAK AMP-PNP dataset [picked particles - single frame - unprocessed] Data #4: Dose-weighted aligned movie averages of the FAK APO Polara dataset [micrographs - single frame] Data #5: Dose-weighted aligned movie averages of the FAK APO Titan dataset [micrographs - single frame] Data #6: Unaligned movies of the FAK APO Titan dataset [micrographs - multiframe] Data #7: Stack of particles extracted from the FAK APO Titan and Polara datasets [picked particles - single frame - unprocessed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10615.map.gz / 形式: CCP4 / 大きさ: 51.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10615.map.gz / 形式: CCP4 / 大きさ: 51.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map obtained from single particle analysis of FAK 2D crystals. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
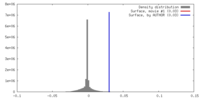
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10615_msk_1.map emd_10615_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
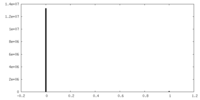
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10615_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
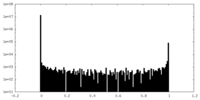
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10615_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
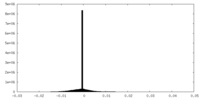
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Focal adhesion kinase
| 全体 | 名称: Focal adhesion kinase |
|---|---|
| 要素 |
|
-超分子 #1: Focal adhesion kinase
| 超分子 | 名称: Focal adhesion kinase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 150 KDa |
-分子 #1: Focal adhesion kinase 1
| 分子 | 名称: Focal adhesion kinase 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: non-specific protein-tyrosine kinase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 75.476445 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GPGAMERVLK VFHYFENSSE PTTWASIIRH GDATDVRGII QKIVDCHKVK NVACYGLRLS HLQSEEVHWL HLDMGVSNVR EKFELAHPP EEWKYELRIR YLPKGFLNQF TEDKPTLNFF YQQVKNDYML EIADQVDQEI ALKLGCLEIR RSYGEMRGNA L EKKSNYEV ...文字列: GPGAMERVLK VFHYFENSSE PTTWASIIRH GDATDVRGII QKIVDCHKVK NVACYGLRLS HLQSEEVHWL HLDMGVSNVR EKFELAHPP EEWKYELRIR YLPKGFLNQF TEDKPTLNFF YQQVKNDYML EIADQVDQEI ALKLGCLEIR RSYGEMRGNA L EKKSNYEV LEKDVGLRRF FPKSLLDSVK AKTLRKLIQQ TFRQFANLNR EESILKFFEI LSPVYRFDKE CFKCALGSSW II SVELAIG PEEGISYLTD KGANPTHLAD FNQVQTIQYS NSEDKDRKGM LQLKIAGAPE PLTVTAPSLT IAENMADLID GYC RLVNGA TQSFIIRPQK EGERALPSIP KLANNEKQGV RSHTVSVSET DDYAEIIDEE DTYTMPSTRD YEIQRERIEL GRCI GEGQF GDVHQGIYMS PENPAMAVAI KTCKNCTSDS VREKFLQEAL TMRQFDHPHI VKLIGVITEN PVWIIMELCT LGELR SFLQ VRKFSLDLAS LILYAYQLST ALAYLESKRF VHRDIAARNV LVSATDCVKL GDFGLSRYME DSTYYKASKG KLPIKW MAP ESINFRRFTS ASDVWMFGVC MWEILMHGVK PFQGVKNNDV IGRIENGERL PMPPNCPPTL YSLMTKCWAY DPSRRPR FT ELKAQLSTIL EEEKLQ UniProtKB: Focal adhesion kinase 1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 緩衝液 | pH: 5.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 撮影 | Image recording ID: 1 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #1~
電子顕微鏡法 #1~
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI POLARA 300 |
| 撮影 | Image recording ID: 2 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.26 mm |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT | ||||||
| 得られたモデル |  PDB-6ty3: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)