+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9392 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bacteriophage L virion | |||||||||
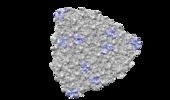
 マップデータ マップデータ | 1/8th of the virion for phage L-segmentation shows capsid protein only | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Dec protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Enterobacteria phage L (ファージ) Enterobacteria phage L (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Parent KN / Schrad JR | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019タイトル: The phage L capsid decoration protein has a novel OB-fold and an unusual capsid binding strategy. 著者: Rebecca L Newcomer / Jason R Schrad / Eddie B Gilcrease / Sherwood R Casjens / Michael Feig / Carolyn M Teschke / Andrei T Alexandrescu / Kristin N Parent /  要旨: The major coat proteins of dsDNA tailed phages (order ) and herpesviruses form capsids by a mechanism that includes active packaging of the dsDNA genome into a precursor procapsid, followed by ...The major coat proteins of dsDNA tailed phages (order ) and herpesviruses form capsids by a mechanism that includes active packaging of the dsDNA genome into a precursor procapsid, followed by expansion and stabilization of the capsid. These viruses have evolved diverse strategies to fortify their capsids, such as non-covalent binding of auxiliary 'decoration' (Dec) proteins. The Dec protein from the P22-like phage L has a highly unusual binding strategy that distinguishes between nearly identical three-fold and quasi-three-fold sites of the icosahedral capsid. Cryo-electron microscopy and three-dimensional image reconstruction were employed to determine the structure of native phage L particles. NMR was used to determine the structure/dynamics of Dec in solution. The NMR structure and the cryo-EM density envelope were combined to build a model of the capsid-bound Dec trimer. Key regions that modulate the binding interface were verified by site-directed mutagenesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9392.map.gz emd_9392.map.gz | 64.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9392-v30.xml emd-9392-v30.xml emd-9392.xml emd-9392.xml | 13.1 KB 13.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9392.png emd_9392.png | 272.3 KB | ||
| その他 |  emd_9392_additional.map.gz emd_9392_additional.map.gz | 688.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9392 http://ftp.pdbj.org/pub/emdb/structures/EMD-9392 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9392 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9392 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9392_validation.pdf.gz emd_9392_validation.pdf.gz | 77.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9392_full_validation.pdf.gz emd_9392_full_validation.pdf.gz | 76.9 KB | 表示 | |
| XML形式データ |  emd_9392_validation.xml.gz emd_9392_validation.xml.gz | 492 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9392 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9392 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9392 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9392 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9392.map.gz / 形式: CCP4 / 大きさ: 76.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9392.map.gz / 形式: CCP4 / 大きさ: 76.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 1/8th of the virion for phage L-segmentation shows capsid protein only | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.26 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
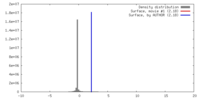
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: single Dec trimer segmented from phage L virion map
| ファイル | emd_9392_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | single Dec trimer segmented from phage L virion map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
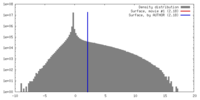
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Enterobacteria phage L
| 全体 | 名称:  Enterobacteria phage L (ファージ) Enterobacteria phage L (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Enterobacteria phage L
| 超分子 | 名称: Enterobacteria phage L / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1 / NCBI-ID: 45441 / 生物種: Enterobacteria phage L / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) |
| 分子量 | 理論値: 60 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: coat protein / T番号(三角分割数): 7 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
| |||||||||
| グリッド | 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 100.0 nm / 詳細: unspecified | |||||||||
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER | |||||||||
| 詳細 | concentration is 1x10^12 phage/mL |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-20 (5k x 3k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 5000 pixel / デジタル化 - サイズ - 縦: 4000 pixel / デジタル化 - サンプリング間隔: 6.4 µm / デジタル化 - 画像ごとのフレーム数: 0-53 / 撮影したグリッド数: 1 / 実像数: 494 / 平均露光時間: 2.0 sec. / 平均電子線量: 27.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.49 µm / 最小 デフォーカス(公称値): 0.35 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)