+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9023 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ structure of 96-nm repeat unit of Tetrahymena ciliary axoneme by TYGRESS method | |||||||||
 マップデータ マップデータ | In situ structure of 96 nm repeat unit from intact Tetrahymena ciliary axoneme resolved by a newly developed TYGRESS method | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Tetrahymena thermophila CU428 (真核生物) Tetrahymena thermophila CU428 (真核生物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 12.0 Å | |||||||||
 データ登録者 データ登録者 | Song K / Shang Z / Fu X / Lou X / Grigorieff N / Nicastro D | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2020 ジャーナル: Nat Methods / 年: 2020タイトル: In situ structure determination at nanometer resolution using TYGRESS. 著者: Kangkang Song / Zhiguo Shang / Xiaofeng Fu / Xiaochu Lou / Nikolaus Grigorieff / Daniela Nicastro /  要旨: The resolution of subtomogram averages calculated from cryo-electron tomograms (cryo-ET) of crowded cellular environments is often limited owing to signal loss in, and misalignment of, the ...The resolution of subtomogram averages calculated from cryo-electron tomograms (cryo-ET) of crowded cellular environments is often limited owing to signal loss in, and misalignment of, the subtomograms. By contrast, single-particle cryo-electron microscopy (SP-cryo-EM) routinely reaches near-atomic resolution of isolated complexes. We report a method called 'tomography-guided 3D reconstruction of subcellular structures' (TYGRESS) that is a hybrid of cryo-ET and SP-cryo-EM, and is able to achieve close-to-nanometer resolution of complexes inside crowded cellular environments. TYGRESS combines the advantages of SP-cryo-EM (images with good signal-to-noise ratio and contrast, as well as minimal radiation damage) and subtomogram averaging (three-dimensional alignment of macromolecules in a complex sample). Using TYGRESS, we determined the structure of the intact ciliary axoneme with up to resolution of 12 Å. These results reveal many structural details that were not visible by cryo-ET alone. TYGRESS is generally applicable to cellular complexes that are amenable to subtomogram averaging. #1:  ジャーナル: biorXiv ジャーナル: biorXivタイトル: Structure of the ciliary axoneme at nanometer resolution reconstructed by TYGRESS 著者: Song K / Shang Z / Fu X / Lou X / Grigorieff N / Nicastro D | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9023.map.gz emd_9023.map.gz | 747.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9023-v30.xml emd-9023-v30.xml emd-9023.xml emd-9023.xml | 9.7 KB 9.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9023.png emd_9023.png | 112.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9023 http://ftp.pdbj.org/pub/emdb/structures/EMD-9023 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9023 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9023 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9023_validation.pdf.gz emd_9023_validation.pdf.gz | 78.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9023_full_validation.pdf.gz emd_9023_full_validation.pdf.gz | 78.1 KB | 表示 | |
| XML形式データ |  emd_9023_validation.xml.gz emd_9023_validation.xml.gz | 492 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9023 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9023 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9023 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9023 | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9023.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9023.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | In situ structure of 96 nm repeat unit from intact Tetrahymena ciliary axoneme resolved by a newly developed TYGRESS method | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.112 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
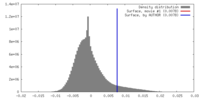
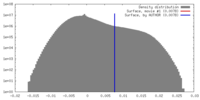
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : 96 nm repeat unit from Tetrahymena ciliary axoneme
| 全体 | 名称: 96 nm repeat unit from Tetrahymena ciliary axoneme |
|---|---|
| 要素 |
|
-超分子 #1: 96 nm repeat unit from Tetrahymena ciliary axoneme
| 超分子 | 名称: 96 nm repeat unit from Tetrahymena ciliary axoneme / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Tetrahymena thermophila CU428 (真核生物) / Organelle: Cilia Tetrahymena thermophila CU428 (真核生物) / Organelle: Cilia |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 1.5 µm / 倍率(補正後): 9400 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 12.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 詳細: TYGRESS was used for the reconstruction method / 使用した粒子像数: 18857 |
|---|---|
| 初期 角度割当 | タイプ: OTHER / 詳細: From subtomogram alignment |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)